Suomalaisen hirven (Alces alces) geneettinen monimuotoisuus ja populaatiorakenne. Veli-Matti Kangas
|
|
|
- Timo Halttunen
- 6 vuotta sitten
- Katselukertoja:
Transkriptio
1 Suomalaisen hirven (Alces alces) geneettinen monimuotoisuus ja populaatiorakenne Veli-Matti Kangas Pro gradu-tutkielma Oulun yliopisto Biologian laitos Toukokuu 2010
2 Sisällysluettelo 1. JOHDANTO HIRVI Hirven levinneisyys Lisääntyminen Dispersaali, elinpiiri ja vaellus Hirvikannan historia ja nykytila Suomessa Hirvikannan säätely ja valikoiva verotus POPULAATIOGENETIIKKA Mikrosatelliitit geenimerkkeinä Populaatioiden geneettinen muuntelu Efektiivinen populaatiokoko Populaation pullonkaula Sukusiitos pienessä populaatiossa Populaatiorakenne ja geenivirta Geneettinen muuntelu hirvellä TÄMÄN TUTKIMUKSEN TARKOITUS AINEISTO JA MENETELMÄT NÄYTTEET DNA:N ERISTYS MIKROSATELLIITIT PCR AGAROOSIELEKTROFOREESI KAPILLAARIELEKTROFOREESI JA NÄYTTEIDEN GENOTYYPPIEN MÄÄRITYS AINEISTON ANALYSOINTI Hardy-Weinberg-tasapaino ja kytkentäepätasapaino Populaatioiden geneettinen muuntelu ja sukusiitos Populaatiorakenne Geenivirta Efektiivinen populaatiokoko Pullonkaula TULOKSET HARDY-WEINBERG-TASAPAINO JA KYTKENTÄTASAPAINO LOKUSTEN JA POPULAATIOIDEN GENEETTINEN MUUNTELU POPULAATIORAKENNE GEENIVIRTA EFEKTIIVINEN POPULAATIOKOKO PULLONKAULA POHDINTA AINEISTON LUOTETTAVUUS SUOMALAISEN HIRVEN GENEETTINEN MUUNTELU HIRVEN GENEETTINEN POPULAATIORAKENNE JA GEENIVIRTA SUOMESSA PULLONKAULA METSÄSTYKSEN GENEETTISET VAIKUTUKSET JATKOTUTKIMUKSET YHTEENVETO KIITOKSET LÄHTEET LIITTEET... 66
3 1. JOHDANTO 1.1 Hirvi Hirvi (Alces alces) on sorkkaeläinten lahkoon (Artiodactyla) kuuluvan hirvieläinten heimon (Cervidae) kookkain laji sekä sukunsa ainoa edustaja. Se on levittäytynyt koko pohjoisen pallonpuoliskon havumetsävyöhykkeelle, ja on yksi tämän alueen suurimmista maanisäkkäistä (Telfer 1984). Fossiiliaineiston perusteella varhaisimmat todisteet modernin hirven olemassaolosta ajoittuvat noin vuoden päähän (Telfer 1984). Hirven oletetaan kehittyneen Aasiassa, josta se pleistoseenikauden aikana siirtyi sekä Eurooppaan että Beringin maasillan kautta Pohjois-Amerikkaan. Jäätiköiden ollessa laajimmillaan hirven nykyiset elinalueet olivat joko jään, tundran tai kylmän aron valtaamia. Merenpinnan muutokset ja kylmän ilmaston leviäminen kohti päiväntasaajaa loivat hirvelle sopivat ilmasto- ja kasvillisuusolot alueille, joista laji nykyään puuttuu. Pleistoseenikaudella boreaalisen kasvillisuusvyöhykkeen alaraja oli mantereesta riippuen 30-40º pohjoista leveyttä (Telfer 1984). Mannerjäätiköiden sulaessa hirvet levisivät uusille, aiemmin jään peitossa olleille alueille. Hirven nykyinen levinneisyysalue sijoittuu pääasiassa välille 50-70º pohjoista leveyttä (alimmillaan se on lähellä 40º) Hirven levinneisyys Hirvi on levinneisyydeltään sirkumpolaarinen laji (Kuva 1), joka jaetaan nykyisin luokittelusta riippuen 7-8 alalajiin (Telfer 1984, Hundertmark ym. 2002a), joista sekä Euraasiassa ja Pohjois-Amerikassa esiintyy neljää. Euroopanhirven (A. a. alces) levinneisyysaluetta on Eurooppa ja läntinen Aasia, siperianhirven (A. a. pfizenmayeri) Keski-Aasia, mantšurianhirven (A. a. cameloides) Mongolia, Luoteis-Kiina ja Kaakkois-Venäjä ja kamšatkanhirven (A. a. burtulini) taas Venäjän Kaukoitä. Pohjoisamerikkalaisista alalajeista alaskanhirveä (A. a. gigas) tavataan Alaskassa sekä Yukonin alueella Kanadassa, länsikanadanhirveä (A. a. andersoni) Länsi- ja Keski-Kanadassa sekä USA:n pohjois-keskiosassa, kalliovuortenhirveä (A. a. shirasi) Kalliovuorten alueella ja itäkanadanhirveä (A. a. americanus) Pohjois-Amerikan itäosissa (Hundertmark ym. 2002a) Mustanmeren ja Kaspianmeren välimaastoa asuttanut kaukasianhirvi (A. a. caucasicus) kuoli sukupuuttoon 1800-luvulla (Ruusila & Malinen 2006). 1
4 Kuva 1. Hirven maailmanlaajuinen levinneisyys alalajeittain Hundertmarkin (2002b) mukaan. Hirven elintavat vaihtelevat laajan levinneisyysalueen eri osissa. Hirvi on sopeutunut pohjoisen havumetsävyöhykkeen elinympäristöihin, mutta voi hyödyntää myös kylmempien (tundra) ja lauhkeampien (lehtimetsät, arojen jokivarsien metsät) biomien elinympäristöjä (Nygren & Wallen 2001). Hirviä esiintyy havumetsävyöhykkeen eteläpuolisella havu- ja lehtimetsien välisellä vaihtumisvyöhykkeellä aina Mantšuriasta Puolaan ja Ruotsiin saakka. Pohjoisessa, metsän ja tundran välivyöhykkeellä, sekä boreaalisen vyöhykkeen korkeimmilla seuduilla hirvet elävät tyypillisesti laaksoissa. Norjassa, Newfoundlandissa ja Alaskassa hirvet asuttavat puurajan läheisiä pensasalueita (Telfer 1984). Hirven elinalue keskittynyt sekä Euraasiassa että Pohjois-Amerikassa laajoille mantereisille tasangoille, mutta sitä tavataan myös vuoristoisilla seuduilla (Telfer 1984). Alalajeista erityisesti mantšurianhirvi ja kalliovuortenhirvi ovat sopeutuneet elämään vuoristoalueilla (Geist 1987, Jia 1996, Hundertmark & Bowyer 2004). 2
5 Hirveä tavattiin ennen suuressa osassa läntistä Eurooppaa, josta se kuitenkin hävisi holoseenikaudelta keskiajalle ulottuvalla aikajaksolla (Schmölcke & Zachos 2005). Pohjois-Amerikassa hirvi on pääasiassa säilyttänyt historiallisen levinneisyysalueensa muutamia poikkeuksia lukuun ottamatta. Lisäksi ihminen on laajentanut hirven elinaluetta Amerikassa istuttamalla lajia uusille alueille (Telfer 1984). Telfer (1984) pitää käyttäytymisen joustavuutta yhtenä syynä sille, että hirvi on säilyttänyt niin suuren osan historiallisesta elinalueestaan aina luvulle asti huolimatta sekä luonnollisista että maa- ja metsätalouden aiheuttamista muutoksista hirven habitaateissa. Hirvi on hämärä- ja päiväaktiivinen laji. Ravintonsa se saa suurimmaksi osaksi puista, pensaista ja ruohokasveista. Laji suosii nuoria lehtipuuvaltaisia metsiä (Siivonen & Sulkava 1994), ja erityisesti Euraasiassa tärkeä osa sen elinympäristöä ovat suot, joiden kasvillisuutta se käyttää hyödykseen metsäkasvien kasvukauden loputtua. Kesällä suot antavat suojaa helteiltä ja hyönteisiltä, minkä lisäksi myös talvehtimisalueet sijaitsevat usein soiden reunaalueilla, joista löytyy syötäväksi havupuiden versoja sekä lehtipuiden (kuten pajujen) silmuja, oksien kärkiä ja kuorta (Nygren & Wallen 2001). Vaikka vasallista naarasta lukuun ottamatta hirvet ovat tavallisesti yksineläjiä, voivat ne talvisin kerääntyä jopa satapäisiksi laumoiksi (Nygren & Wallen 2001) Lisääntyminen Hirvien kiima-aika ajoittuu lajin koko levinneisyysalueella syksyyn, mutta lisääntymisstrategiat poikkeavat toisistaan eri biomien välillä. Tundralla pariutuminen tapahtuu suurissa paikallaan pysyttelevissä laumoissa, mutta vastaavasti havumetsävyöhykkeellä hirvet ovat peräkkäisyksiavioisia (serial monogamy) (Bubenik 1987). Tässä Suomessakin vallalla olevassa pariutumisjärjestelmässä kiimaan tulossa oleva hirvinaaras alkaa kierrellä elinpiiriään hakeutuen urosten puolustamille ns. kiimakentille. Niiltä ne voivat saada seuraansa useita hirvisonneja, jotka taistelevat keskenään oikeudesta paritella. Tapahtumaketju on täysin naaraan säätelemä (matriarkaatti), sillä urosten keskinäisistä voimainkoitoksista huolimatta se valitsee mieleisensä pariutumiskumppanin, sekä säätelee lisäksi uroksen kiimakäyttäytymistä, astumisen ajoittumista ja frekvenssiä (Bubenik 1987, Nygren & Wallen 2001). Kun naaraan kiiman huippu on ohitettu ja sen käytös muuttuu torjuvaksi, voi uros lähteä etsimään uusia parittelukumppaneita. Naaraiden kantoaika kestää kevään lopulle, jolloin ne synnyttävät 1-2 vasaa. 3
6 Tätä ennen naaras vieroittaa mahdolliset edellisvuoden vasat, jotka lähtevät etsimään omaa elinpiiriään (Nygren & Wallen 2001). Hirvet ovat tehokkaita lisääntymään, ja hirvikannan kasvu voi olla todella nopeaa olosuhteiden ollessa hyvät. Nygrenin (2009) mukaan kannan koostumus, ympäristö- ja ravintoolosuhteet sekä perimä yhdessä ovat hirvipopulaation lisääntymiseen olennaisimmin vaikuttavat tekijät. Hirvinaaras on hedelmällisyytensä huipussa n. 7 vuoden ikäisenä, mutta voi saavuttaa sukukypsyyden jo 1,5-vuotiaana (Nygren 2009). Myös urokset tulevat sukukypsiksi varsin varhaisessa iässä (Schwartz ym. 1982), mutta parhaassa lisääntymisiässä olevien sonnien riittävän määrän katsotaan olevan kuitenkin hyvin tärkeää sekä optimaalisen lisääntymisen että lajin evolutiivisen menestyksen kannalta (Bubenik 1987, Milner ym. 2007, Nygren 2009) Dispersaali, elinpiiri ja vaellus Dispersaalille on tieteellisessä kirjallisuudessa esitetty useita määritelmiä. Howard (1960, johon viitanneet Johnson & Gaines 1990) määritti termin yksilön pysyvänä muuttona syntymäalueeltaan paikkaan, jossa se lisääntyy tai olisi lisääntynyt, jos olisi selvinnyt elossa ja löytänyt kumppanin. On mahdollista myös erottaa ekologinen ja geneettinen dispersaali. Ekologisella dispersaalilla tarkoitetaan yksinkertaisesti yksilön liikkumista paikasta toiseen, kun taas geneettinen dispersaali viittaa erityisesti yksilöihin, jotka populaatiosta toiseen muutettua onnistuvat lisääntymään. Ekologinen dispersaali siis sisältyy geneettiseen dispersaaliin, mutta vastaavasti ei toisinpäin (Johnson & Gaines 1990). Tässä tutkielmassa dispersaali-termiä käytetään Howardin määritelmän mukaisesti. Hirvet ovat erittäin liikkuvia ja voivat edetä pitkiäkin matkoja suhteellisen lyhyessä ajassa (Hoffman ym. 2006). Tavallisin dispersoiva yksilö on emänsä vieroittama nuori hirvi, joka tekee parin ensimmäisen elinvuotensa aikana lyhyen vaelluksen tarkoituksenaan löytää sopiva elinympäristö (Nygren & Wallen 2001). Kuten nisäkkäille on tyypillistä, myös hirvillä urokset dispersoivat naaraita kauemmas synnyinseudultaan (Cederlund & Sand 1992). Vahvasta dispersaalipotentiaalista huolimatta hirvien on useissa tutkimuksissa havaittu olevan voimakkaasti paikkauskollisia (eli filopatrisia) elinpiireilleen ja elävän suhteellisen pienellä alueella (esim. Sweanor & Sandegren 1989, Cederlund & Sand 1992, Heikkinen 2000). 4
7 Hirviyksilöt eivät yleensä vietä kesää ja talvea samalla alueella, ja niiden vuosi rakentuukin erillisistä kesä- ja talvilaidunkausista, sekä niiden välisistä kevään ja syksyn liikkumisjaksoista. Vaelluskäyttäytymisen yksilökohtaisista eroista johtuen hirveä pidetään osittaisvaeltajalajina (partially migratory species, Nygren 2009). Lajin liikkumista sekä elinpiirin kokoa ja käyttöä on tutkittu suhteellisen paljon sekä Euroopassa että Pohjois-Amerikassa. Elinpiirin kokoon on havaittu vaikuttavan monia tekijöitä, kuten mm. yksilön sukupuoli ja ikä, paikallinen maasto, kasvillisuuden fenologia, hirvikannan rakenne ja tiheys sekä erilaiset häiriöt (Nygren & Wallen 2001). Tämän vuoksi on yhdellä alueella havaittujen käyttäytymispiirteiden yleistäminen toisenlaisiin olosuhteisiin hyvin vaikeaa. Esimerkiksi Ruotsissa kerättyjä tuloksia ei voida suoraa soveltaa Suomen oloihin (Heikkinen 2000). Kaivattua tarkempaa tietoa hirvien liikkumisesta ja elinpiirien suuruudesta Suomessa saatiin 90-luvulla Oulun seudulla tehdyssä mittavassa radiotelemetriatutkimuksessa (mm. Heikkinen 2000, Nikula ym. 2004). Tuossa tutkimuksessa hirvien kevätvaelluksien havaittiin olevan lyhyitä, ja tämän määrätietoisen kesälaidunalueelle siirtymisen keskipituus oli ainoastaan noin 15 km. Urokset vaelsivat tätä pidemmälle useammin kuin naaraat. Hirviyksilöiden todettiin palavaan hyvin uskollisesti vuodesta toiseen samalle kesälaidunalueelle, jonka keskimääräinen koko oli tutkimusalueella noin 1400 ha. Liikkumisen ja elinpiirin käytön havaittiin olevan siellä aktiivisempaa kuin talvilaidunalueella, jonka keskikooksi laskettiin 1200 ha. Talvella elinpiirin sijainnin vaihtelu on vuosien välillä suurempaa kuin kesällä, ja sen käytön voidaan kuvata olevan pienipiirteisestä siirtyilyä ravintolaikulta. Talvielinpiirin koon huomattiin olevan uroksilla suurempi naaraisiin verrattuna. Hirvien liikkuminen oli voimakkainta syksyllä, jolloin Heikkinen (2000) havaitsi elinalueiden laajenevan huomattavasti suuremmaksi kesäaikaisista, ja hirvien huomattiin vaeltelevan ennen talvilaitumelle asettumista kesä- ja talvielinpiirien välillä edestakaisin useiden kymmenien kilometrien etäisyydestä huolimatta. Syysliikkumisen laajuuteen ja aktiivisuuteen vaikuttavat elinpiirien vuodenaikaisvaihdon lisäksi hyvin mahdollisesti myös kiima-aika sekä metsästys (Heikkinen 2000) Hirvikannan historia ja nykytila Suomessa Hirvi on asuttanut Fennoskandiaa esihistorialliselta ajalta lähtien. Laji saapui tänne viimeisen jääkauden aikaisen mannerjäätikön vetäytymisen jälkeen vähintään 7500 vuotta sitten, ja jo mesoliittiselta kaudelta saakka on ihminen metsästänyt hirviä Suomen alueella (Nygren 1987, Pulliainen 1987). Kirjallista tietoa on maamme hirvistä olemassa 1500-luvulta alkaen. Tuol- 5
8 loin kanta lienee ollut kooltaan vaatimaton, mutta silti koko maan kattava (Nygren 1987, 2009). Tämän jälkeen 1600-luvulla alkaneiden, holtittomasta metsästyksestä johtuneiden paikallisten sukupuuttojen sarja johti koko maan laajuiseen hirvikannan romahtamiseen. Aikalaislähteiden mukaan 1800-luvun puoleenväliin tultaessa koko Suomen hirvipopulaatio oli sukupuuton partaalla (Nygren 2009). Kanta sai aikaa elpyä vain vuodesta 1868 vuosisadan loppuun kestäneen rauhoitusjakson ajan, jonka jälkeen yksilömäärät romahtivat jälleen luvun alun levottomina vuosina. Vuonna 1923 säädetyn rauhoituksen jälkeen hieman voimistunut kanta oli jakautunut koilliseen ja lounaiseen populaatioon. Näistä jälkimmäisen uskotaan syntyneen vain kuudesta Varsinais-Suomessa suojelun ansiosta säilyneestä hirviyksilöstä (Nygren 1987, 2009). Kanta-arvio vuodelta 1935 oli ainoastaan noin 3500 hirveä, minkä jälkeen sotavuodet vuosikymmenen vaihteessa tekivät lovensa hirvien määrään. Sotien jälkeen alkanut metsähakkuiden lisääntyminen ja suo-ojitukset kasvattivat suomalaismetsien taimikkoalaa ja näin paransivat hirvien ravinnonsaantia, ja 1950-luvulla hirvikanta alkoikin kasvaa ripeästi (Nygren 2009). Vuonna 1966 hirvien määräksi laskettiin jo noin yksilöksi (Koivisto 1966). Liian korkeiksi nostetut kaatokiintiöt kuitenkin saivat kannan taas laskemaan, mikä johti paikallisiin rauhoituksiin lukujen vaihteessa. Tämän jälkeen suomalaisen hirvikantojen säätelyssä tapahtui sen suurin muutos, eli siirtyminen vasaverotukseen, jonka seurauksena kanta alkoi nopeasti voimistua (Kuva 2). Kasvun vyöhyke eteni maan eteläosista pohjoiseen tiheyshuipun ajoittuessa etelässä 70-luvun lopulle ja pohjoisessa 80-luvun alkuun (Nygren 2009). Hirvikanta kuitenkin jälleen pieneni huippuvaiheesta vuoteen 1996 mennessä jyrkästi suunnitelmallisesta kannanverotuksesta johtuen. Tämä johti kaatomäärien reiluun vähentämiseen, mistä vastaavasti seurasi 2000-luvun alussa jälleen räjähdysmäinen kasvu ja toinen huippukausi (Nygren 2009): esimerkiksi talvella hirvien määräksi arvioitiin yksilöä (Ruusila ym. 2002). Ja kuten aikaisemminkin, liiaksi kasvanutta kantaa ruvettiin jälleen pienentämään voimakkaalla verotuksella. Syksyllä 2002 koko maassa kaadettiin yhteensä hirveä (Ruusila ym. 2003). Vuosikymmenen loppuun mennessä hirvikanta oli pienentynyt lähes yhtä rajusti kuin oli kasvanutkin. Jahtikauden 2008 jälkeen kannan kooksi arvioitiin hirveä (Pusenius 2009). 6
9 Kuva 2. Hirvien kaatomäärät ja jahtikauden jälkeiset kanta-arviot vuosina Tiilikaisen (2010) mukaan Hirvikannan säätely ja valikoiva verotus Suomen hirvikanta on nykyisin voimakkaasti säädelty. Sotien jälkeen on kaatolupiin perustuva metsästys ollut Suomessa merkittävin hirvien lukumäärää rajoittava tekijä (Nygren 2009) luvulta lähtien ovat säätelyn keskeisinä toimijoina olleet Maa- ja metsätalousministeriö, Metsästäjäin Keskusjärjestö (sisältäen riistahoitopiirit ja -yhdistykset) sekä Riista- ja kalatalouden tutkimuslaitos (Nikula ym. 2007). Tehtävän säätelyn päämäärät voidaan jakaa määrällisiin ja laadullisiin tavoitteisiin. Määrällisten, eli käytännössä tiheystavoitteiden taustana ovat hirvien aiheuttamat vahingot liikenteelle ja metsille sekä arviot Suomen luonnonolosuhteisiin soveltuvien hirvitiheyksien raja-arvoista. Laadullisina tavoitteina voidaan pitää kannan ikä- ja sukupuolirakenteen säätelyä halutuille tasoille (Nygren 2009). Sekä määrälliset että laadulliset tavoitteet ovat Suomessa muuttuneet ajan saatossa luvulta lähtien on säätelytyön keskeisimpänä periaatteena ollut voimakas vasaverotus, jolla on pyritty takaamaan erittäin tuottava hirvikanta, joka on vastaavasti ollut laadullisen säätelyn tärkein tavoite. Metsästettäessä paljon vasoja kantaan jää suhteessa enemmän aikuisia, mikä nostaa kannan keskiikää ja parantaa siten tuottavuutta. Tällä tavalla toimimalla on pyritty takamaan suuret saalismäärät (Nygren 2009). 7
10 Valikoiva verotus on Nygrenin (2009) mukaan yleistermi, jolla tarkoitetaan tapaa suunnitella kannan harvennus päättämällä ennakkoon, mitä sukupuolta ja ikäluokkaa metsästetään, taikka erityisesti suojellaan. Vuodesta 1978 vuoteen 1996 ulottuvalla aikajaksolla valikoivaa verotusta harrastettiin Suomessa säätämällä urosten, naaraiden ja vasojen kaatomäärät vuosittain kannanrakenteesta ennalta määrättyjen tavoitteiden mukaisesti päämääränä vakiintunut kanta luvulla siirryttiin kaavamaisempaan valikoivaan verotukseen, jossa saalista kaadetaan vuosittain vähintään puolet vasoina ja puolet aikuisina, joista vastaavasti noin puolet uroksia ja puolet naaraita (Nygren 2009). Nykyisin uroksia kaadetaan kuitenkin lähes poikkeuksetta naaraita enemmän osaksi vasallista emää suojaavasta lainsäädännöstä, mutta myös metsästäjien mieltymyksistä johtuen. Urosten osuudet ovatkin supistuneet kaikilla suuralueilla, ja esimerkiksi Etelä-Suomessa lehmä/sonnilukusuhde on noussut tasolta 1,2 jopa yli tason 2,0 naarasta jokaista urosta kohti, mikä on korkeampi kuin koskaan aikaisemmin (Nygren 2009). Lisäksi saaliiksi päätyvät useimmin parhaassa siitosiässä olevat suurisarviset urokset (Solberg ym. 2000) luvun suurten kaatovuosien jälkeen nousi huoli suurisarvisten urosten kadosta, minkä seurauksena alettiin antaa metsästäjille sarvipiikkisuosituksia luvulle tultaessa näistä suosituksista ja valikoivasta sarviverotuksesta on luovuttu (Nygren 2009). 1.2 Populaatiogenetiikka Mikrosatelliitit geenimerkkeinä Geenimerkeiksi (genetic marker) kutsutaan polymorfisia proteiinivariantteja (allotsyymit) tai DNA-sekvenssejä, joita voidaan käyttää koko genomin laajuisen geneettisen muuntelun indikaattorina (Beebe & Rowe 2004). Niiden avulla on mahdollista tutkia erilaisia biologisia ongelmia ja ilmiöitä, joiden selvittäminen muuten olisi hyvin työlästä. Geneettisen muuntelun määrän lisäksi geenimerkkien avulla pystytään tekemään johtopäätöksiä esimerkiksi populaatiorakenteesta ja jopa yksilöiden liikkumisesta populaatioiden välillä (Beebee & Rowe 2004). Tiedon ja tekniikan kehittymisen myötä tutkijoilla on nykyisin mahdollisuus valita tutkimusaiheensa mukaan parhaiten soveltuva geenimerkki useista vaihtoehdoista, joita ovat mm. allotsyymit, MHC-geenit (major histocompatibility complex), mitokondrion DNA (mtdna), AFLP (amplified fragment lenght polymorphism), SNP:t (single nucleotide polymorphism) ja mikrosatelliitit (Beebee & Rowe 2004). 8
11 Mikrosatelliitit, eli yksinkertaiset sekvenssitoistot (simple sequence repeats) tai lyhyet tandemtoistot (short tandem repeats), ovat runsaana genomissa esiintyviä, erittäin muuntelevia 1-6 emäsparin mittaisista sekvenssipätkistä koostuvia toistojaksoja (Litt & Luty 1989, Li ym. 2002). Tavallisimmat tyypit ovat di- (esim. CA), tri- (GTG) ja tetranukleotiditoistoiset (GA- TA/GACA) mikrosatelliitit (Jarne & Lagoda 1996). Toistojaksojen rakenteen perusteella voidaan tehdä jako täydellisiin (esim. CACACACA), yhdistelmätoistoihin (CACAGAGA) ja epätäydellisiin toistoihin (CACACATT) (Weber 1990, Jarne & Lagoda 1996). Tavallisesti mikrosatelliittilokuksen pituus on alle 40 toistoa (Beebee & Rowe 2004), ja mikrosatelliiteissa havaittava geneettinen muuntelu perustuu näiden toistojen määrän vaihteluun eikä niinkään variaatioon sekvenssissä itsessään. Mikrosatelliittien heterotsygotia-aste on tyypillisesti korkea ja niissä on runsaasti alleeleja (Ellegren 2004). Sukupuuanalyysit ovat osoittaneet mikrosatelliittien olevan kodominantteja (homo- ja heterotsygoottien erottaminen mahdollista) ja periytyvän mendelistisesti (Jarne & Lagoda 1996). Mikrosatelliitteja löytyy sekä genomin koodaavilta, ja siten valinnan alaisilta alueilta, että myös ei-koodaavilta, neutraaleilta alueilta (Jarne & Lagoda 1996, Ellegren 2004), joilla geenimerkkeinä käytettävät mikrosatelliitit usein sijaitsevat Mikrosatelliiteista on tullut erittäin suosittuja geenimerkkejä ja 2000-luvuilla mm. populaatiobiologiassa lukuisten hyvien ominaisuuksiensa ansiosta (Beebee & Rowe 2004, Ellegren 2004). Näitä ovat mm. jo edellä mainitut tekijät kuten suuri muuntelevuus, neutraalisuus ja kodominanssi. Mikrosatelliittien genotyypin määritys onnistuu suhteellisen helposti, koska niitä on mahdollista monistaa polymeraasiketjureaktion (polymerase chain reaction, PCR) avulla käyttäen spesifisiä oligonukleotidialukkeita, jotka usein ovat käyttökelpoisia lähisukuisten lajien välillä (Beebee & Rowe 2004). PCR-monistus ei kuitenkaan ole täysin ongelmatonta niin kutsuttujen nolla-alleelien vuoksi, joilla tarkoitetaan mikrosatelliittilokuksessa esiintyvää alleelia, jonka monistaminen ei onnistu syystä tai toisesta (Chapuis & Estoup 2007). Genotyyppiaineistossa nolla-alleelit voivat johtaa vääristymään hetero- ja homotsygoottien suhteellisissa osuuksissa ja siten vääriin johtopäätöksiin (Dakin & Avise 2004). Ongelmana voidaan pitää myös mikrosatelliittien mutaatiomekanismien monimutkaisuutta, joka mutkistaa aineistosta saatavan tiedon varmuutta (Ellegren 2004). 9
12 1.2.2 Populaatioiden geneettinen muuntelu Geneettisen muuntelun määrällä, eli geneettisellä diversiteetillä tarkoitetaan tutkivassa joukossa (populaatio, laji yms.) havaittavaa alleelien ja genotyyppien monimuotoisuutta. Geneettistä diversiteettiä tarvitaan, jotta populaatiot kehittyvät selviytyäkseen muuttavassa ympäristössä, ja sen menettäminen onkin usein yhteydessä heikentyneeseen lisääntymistehokkuuteen ja selviytymiseen (lisääntymiskelpoisuus, reproductive fitness). Populaation geneettinen diversiteetti siis kuvastaa sen evolutiivista potentiaalia (Frankham ym. 2002). Geneettistä muuntelua on mahdollista kuvata esimerkiksi genotyyppi- ja alleelifrekvenssien, alleelien lukumäärän tai heterotsygotian avulla. Uutta geneettistä materiaalia tulee populaatioon mutaatioiden ja immigraation kautta, ja geneettinen materiaali sekoittuu meioosin ja rekombinaatioiden kautta lisäten muuntelua. Vastaavasti neutraalia muuntelua vähentävät satunnainen geneettinen ajautuminen (alleelidiversiteetin väheneminen sattuman myötä) ja sukusiitos (Futuyama 1998). Populaatioiden välinen geenivirta vähentää valinnan ja sattuman aiheuttamaa populaatioiden välistä geneettistä erilaistumista, kun taas populaatioiden pirstaloituminen lisää sitä (Frankham ym. 2002). Mutaatioita syntyy enemmän suuressa populaatiossa kuin pienessä, mutta tasapainotilassa mutaatiofrekvenssi on aina sama kuin frekvenssi, jolla alleeleja katoaa geneettisen ajautumisen takia huolimatta populaatiokoosta. Keskimääräinen aika uuden alleelin syntymisen ja fiksaation välillä on kuitenkin verrannollinen efektiiviseen populaatiokokoon (Beebee & Rowe 2004). Pienessä populaatiossa keskimääräinen aika alleeleille fiksoitua on lyhyempi kuin suurissa, minkä lisäksi pienissä populaatioissa menetetään harvinaisia alleeleja satunnaisajautumisen takia nopeammin. Pienet populaatiot ovatkin herkkiä geneettiselle ajautumiselle (Futuyama 1998) Efektiivinen populaatiokoko Efektiivinen populaatiokoko (N e ) on alun perin Wrightin (1931) määrittämä käsite, jolla nykyisin tarkoitetaan yksilömäärää ideaalisessa, oletuksiltaan satunnaisesti pariutuvassa ja sukupuolijakaumaltaan tasaisessa populaatiossa, jossa heterotsygotian vähenemistahti ajan myötä on sama kuin varsinaisessa populaatiossa (Futuyama 1998). Yhdessä mutaation, valinnan, 10
13 migraation ja rekombinaation kanssa efektiivinen populaatiokoko määrittää populaatiossa havaittavan geneettisen muuntelun määrän ja jakautumisen (Wang 2005). N e :n avulla on mahdollista selittää nykyisin havaittavan geneettisen muuntelun määrää sekä siihen vaikuttuneita evolutiivisia mekanismeja. Lisäksi sitä voi käyttää ennustamaan neutraalin geneettisen muuntelun häviämistä ja jakautumista, hyödyllisten ja haitallisten alleelien fiksoitumistodennäköisyyttä sekä pienten populaatioiden kelpoisuutta ja selviytymistä (Wang 2005). Käytännössä N e arvioi niiden yksilöiden määrän, jotka lisääntyvät onnistuneesti jokaisessa sukupolvessa. N e on parempi mittari populaation elinkyvylle kuin kokonaisyksilömäärä, joka yleensä on suurempi kuin N e (Beebee & Rowe 2004). Tämä johtuu siitä, että poikkeamat perusolettamuksista, kuten epätasainen sukupuolijakauma, jälkeläismäärän korkea varianssi ja erityisesti sukupolvittainen populaatiokoon vaihtelu (fluktuaatio) pienentävät populaation efektiivistä kokoa. Syitä sukupuolijakauman vinoutumiseen voivat olla lajin pariutumissysteemi, sukupuolen määräävä mekanismi tai ihmistoiminta, kuten valikoiva metsästys. (Frankham ym. 2002, Allendorf ym. 2008). Populaatiokoon fluktuaatio voi vastaavasti olla seurausta ravintotilanteen muuttumisesta, ilmasto-oloista, tautiepidemioista tai esimerkiksi saalistuksesta (Frankham ym. 2002). Edellä kuvattujen demografisten tekijöiden avulla on mahdollista yrittää arvioida tutkittavan populaation efektiivistä kokoa. Koska näiden parametrien selvittäminen on luonnonpopulaatiosta hyvin vaikeaa, efektiivisen populaatiokoon arviointiin käytetäänkin nykyaikana usein geneettisiä menetelmiä (Wang 2005) Populaation pullonkaula Tilannetta, jossa populaation efektiivinen populaatiokoko tippuu syystä tai toisesta pitkäaikaisen keskiarvon alapuolelle, kutsutaan pullonkaulailmiöksi (bottleneck, Beebee & Rowe 2004). Populaation pullonkaula johtaa alleelimäärän laskemiseen ja ennen kaikkea harvinaisten alleelien menettämiseen. Se vaikuttaa negatiivisesti myös heterotsygotian määrään ja saa aikaan satunnaisia muutoksia alleelifrekvensseissä. Populaatiokoon pieneneminen taas altistaa sukusiitokselle ja satunnaiselle geneettiselle ajautumiselle, mikä voi johtaa geneettiseen erilaistumiseen muista populaatiosta (Frankham ym. 2002). Pullonkaulan vaikutus on sitä suu- 11
14 rempi, mitä voimakkaampaa on populaatiokoon pieneneminen ja mitä kauemmin se kestää (Beebee & Rowe 2004). Vakava pullonkaula voi tapahtua luonnossa myös tapauksissa, joissa uusi populaatio saa alkunsa ainoastaan muutamista yksilöistä. Tätä ilmiötä kutsutaan perustajavaikutukseksi (founder effect, Futuyama 1998) Sukusiitos pienessä populaatiossa Jos pieni populaatio jää sukupolvien ajaksi eristykseen muista populaatioista, eteen voi tulla erilaisia geneettisiä ongelmia. Pienestä populaatiosta seuraa ajan myötä väistämättä sukusiitos, jossa on kyse kahden keskenään sukua (yksi tai useampi yhteinen esivanhempi) olevan yksilön pariutumisesta (Frankham ym. 2002, Beebee & Rowe 2004). Sukusiitos johtaa populaatiossa heterotsygotian alenemiseen, homotsygoottien määrän kasvuun sekä lievästi haitallisten alleelien akkumuloitumiseen ja siten mm. selviytymisen ja lisääntymiskelpoisuuden heikkenemiseen, jota kutsutaan sukusiitosheikkoudeksi (inbreeding depression). Sukusiitos on kuitenkin usein vielä vakavampi ongelma suuressa populaatiossa, jossa hyvin haitalliset alleelit ovat voineet säilyä heterotsygootteina. Vaikka sukusiitoksen myötä syntyvät homotsygootit yksilöt voivat kärsiä suurestikin, niin populaatiotasolla vaikutukset ovat usein pieniä. Varsinkin pienessä populaatiossa tällaisten vakavasti haitallisten alleelien vaikutus ei ole niin suuri, koska valinta yleensä karsii homotsygootit pois. Vaikka sukusiitos muuttaa populaation genotyyppifrekvenssejä, se ei vaikuta alleelifrekvensseihin. Luonnossa useimmat eläinlajit välttävät sukusiitosta. Tätä edesauttavat useat erilaiset mekanismit kuten sukupuolipainotteinen dispersaali, parin/ryhmän ulkopuolinen hedelmöitys, sukulaisten tunnistaminen ja välttäminen parivalinnassa sekä myöhäinen sukukypsyys (Pusey & Wolf 1996) Populaatiorakenne ja geenivirta Luonnossa populaatioiden koon lisäksi populaatioiden rakenteet voivat vaihdella huomattavasti. Yhtenä ääripäänä voidaan pitää yhtenäistä, ns. panmiktista populaatiota, jossa kaikilla yksilöillä on yhtä suuri todennäköisyys pariutua minkä tahansa muun yksilön kanssa. Vaihtoehtoisesti populaatio voi olla sisäisesti jakautunut geneettiseltä muuntelultaan toisistaan eroaviin alapopulaatioihin, joissa yksilöt lisääntyvät todennäköisemmin keskenään kuin muista alapopulaatioista olevien yksilöiden kanssa (Beebee & Rowe 2004, Hedrick 2005). Syynä 12
15 tällaiseen jakoon voivat olla mitkä tahansa yksilöiden liikkumista haittaavat maantieteelliset esteet, mutta myös eliölajin omaan ekologiaan tai käyttäytymiseen liittyvät seikat (Hedrick 2005). Erilaisia populaatiorakenteen malleja on mahdollista kuvata alapopulaatioiden välisten yhteyksien mukaan. Manner-saari-malliksi kutsutaan tilannetta jossa migraatio on yksisuuntaista suuresta mannerpopulaatioista pieniin erillisiin saaripopulaatioihin, kun taas saarimallissa migraatio on tasaista samankokoisten saaripopulaatioiden välillä. Jos migraatiota tapahtuu ainoastaan vierekkäisten naapuripopulaatioiden välillä, on kyseessä astinkivimalli (steppingstone model), joka voi olla muodoltaan joko lineaarinen tai kaksiulotteinen (Frankham ym. 2002, Kuva 3). Populaation ollessa jakautunut, sen alapopulaatioiden geneettiset suhteet riippuvat niiden välillä vaikuttavasta tehollisesta geenivirrasta (gene flow). Tällä termillä tarkoitetaan ryhmissä (populaatio, alapopulaatio) tapahtuvaa geenien liikkumista ja vaihtuvuutta (Hedrick 2005, Neigel 1997). Migraatioksi taas kutsutaan populaatiogenetiikassa tavallisesti nimenomaan alapopulaatioiden välistä geenivirtaa (Neigel 1997), mutta joskus myös pelkkien yksilöiden liikkumista populaatioiden välillä, mikä ei syystä tai toisesta välttämättä edistä geenivirtaa (esim. Frankham ym. 2002). Tässä tutkielmassa termiä käytetään jatkossa Neigelin (1997) määrittämässä tarkoituksessa. 13
16 a) b) c) d) Kuva 3. Neljä erilaista populaatiorakennemallia: a) manner-saari-malli, b) saarimalli, c) lineaarinen astinkivimalli ja d) kaksiulotteinen astinkivimalli. Kun geenivirran määrä on korkea, vaikuttaa se homogenisoivasti populaatioiden väliseen geneettiseen variaatioon. Vastaavasti satunnainen geneettinen ajautuminen, sukusiitos, valinta ja jopa mutaatiot voivat johtaa populaatioiden erilaistumiseen toisistaan geenivirran ollessa vähäistä (Frankham ym. 2002, Hedrick 2005). Geenivirran määrään ja sitä kautta myös populaatioiden erilaistumisasteeseen vaikuttavat mm. eliölajin dispersaalikyky (määrä, etäisyys) ja populaatioiden välinen etäisyys. Rajallinen dispersaalikyky voi johtaa tilanteeseen, jossa yksilöiden geneettinen etäisyys kasvaa yhdessä niiden maantieteellisen etäisyyden kanssa. Tätä kutsutaan etäisyysisolaatioksi (isolation by distance), jonka takia jopa suuren yhtenäisen populaation eri osat voivat olla erilaistuneita toisistaan (Wright 1943, Frankham ym. 2002). Direktionaalisia alleelifrekvenssien muutoksia maantieteellisellä alueella kutsutaan kliineiksi (cline), joita voi syntyä populaation alarakenteen ja populaation eri osissa eri alleeleja suosivan valinnan (differential selection) yhteisseurauksena. Kliinit voivat olla ajallisesti joko vakaita tai väliaikaisia ja niissä alleelifrekvenssit voivat muuttua jyrkästi tai vähittäisesti (Hedrick 2005) 14
17 1.2.7 Geneettinen muuntelu hirvellä Kiinnostus hirven perimän monimuotoisuuteen on herännyt tutkijoiden keskuudessa varsin varhain, ja tutkimuksia aiheesta onkin kertynyt jo viideltä vuosikymmeneltä. Käytettäessä geenimerkkinä allotsyymejä oltiin pitkään siinä luulossa, että hirvi on maailmanlaajuisesti tarkasteltuna geneettisesti lähes monomorfinen laji. Sekä ensimmäisten 1960-luvulla tehtyjen että myöhempienkin tutkimusten tulokset hirven vähäisestä geneettisestä muuntelusta olivat hyvin samansuuntaisia (Hundermark & Bowyer 2004). Ryman ym. (1980) olivat ensimmäisiä, jotka havaitsivat laajempaa muuntelua Fennoskandian hirvipopulaatioissa. Tässä entsyymielektroforeesimenetelmällä tehdyssä pohjoismaisessa yhteistyötutkimuksessa 23 geenilokuksesta viisi havaittiin polymorfisiksi vähintään yhdessä Suomessa, Ruotsissa tai Norjassa tutkituista populaatioista. Osa havaituista alleeleista oli yhteisiä kaikkien kolmen maan hirvipopulaatioille, mutta osa taas selvästi tyypillisiä ainoastaan joko Suomelle tai Skandinavialle. Fosfoglukomutaasin PGM-1-alleelia löydettiin ainoastaan Lounais-Suomen alueelta, jossa 1920-luvun heikkojen vuosien aikana säilyi tiettävästi eteläisen Suomen ainoa hirvikanta (Nygren 1987). Heterotsygotia-asteen avulla mitattuna monimuotoisuus oli korkeinta Suomessa (ka=0,029), jossa korkeimmat lukemat löytyivät Etelä- Hämeen alueelta (0,047) (Ryman ym. 1980). Ryman ym. (1980) siis totesivat että hirvet eivät olleet monomorfisia mihin aikaisemmin tehdyt tutkimukset viittasivat. Heidän mukaansa niissä oli käytetty liian vähän lokuksia tai yksilöitä. Havaittu muuntelu oli joka tapauksessa jonkin verran alhaisempaa kuin monilla muilla nisäkäslajeilla, minkä Ryman ym. (1980) olettivat olevan seurausta pienestä historiallisesta populaatiokoosta johtuneesta geneettisestä ajautumisesta. Vuonna 1982 julkaistussa ruotsalaistutkimuksessa Chesser ym. vertailivat neljällä maantieteellisellä alueella yhteensä 1169 hirviyksilön yhden polymorfisen lokuksen muuntelua. Alueiden välisissä alleelifrekvensseissä todettiin eroavaisuuksia, minkä lisäksi myös yhden alueen sisäinen havaittu muuntelu oli aikaisempaan verrattuna huomattavaa. Tämä oli ensimmäinen kerta, kun muuntelua havaittiin näin pienellä, käytännössä yhden populaation käsittävällä maantieteellisellä mittakaavalla (Chesser ym. 1982, Hundertmark & Bowyer 2004). 15
18 Tekniikan ja uusien geenimerkkien kehittymisen myötä väite hirvien geneettisestä monomorfiasta romuttui lopullisesti, mutta havaitun muuntelun määrä oli edelleen alhainen (Hundertmark & Bowyer 2004). Yhden ensimmäisistä PCR-tekniikkaa hyödyntäneistä tutkimuksista tekivät Mikko ja Andersson (1995). Tuossa tutkimuksessa verrattiin MHC:n luokan II DRBlokuksen ja mtdna:n muuntelua ruotsalaisten ja kanadalaisten hirvien välillä. Molemmissa maissa havaittiin MHC-muuntelun olevan vähäistä, minkä oletettiin olevan seurausta historiallisesta pullonkaulasta ennen kanadalaisen ja eurooppalaisen linjan eroamista. Myös kahdessa hirven maailmanlaajuista mitokondrion DNA:n muuntelua tarkastelleessa tutkimuksessa tulokset olivat yhteneväisiä aikaisempien havaintojen kanssa viitaten muihin sorkkaeläimiin verrattuna suhteellisen alhaiseen geneettiseen diversiteettiin (Hundertmark ym. 2002a, 2002b). Mikrosatelliitteja käytettiin hirvillä ensimmäisen kerran vuonna 1997 julkaistussa neutraalia ja adaptiivista muuntelua vertailleessa kanadalaistutkimuksessa (Wilson ym. 1997). Tämän jälkeen niitä on hyödynnetty hirven geneettisen muuntelun ja populaatiorakenteen tutkimukseen Kanadan (esim. Broders ym. 1999) lisäksi viime vuosina mm. Alaskassa (Schmidt ym. 2009), Ruotsissa (Charlier ym. 2008) ja Norjassa (Solberg ym. 2009). Näiden tutkimuksien taustalla oli uudestaan herännyt huoli metsästyksen mahdollisista negatiivisista vaikutuksista hirvikantoihin. Sekä Ruotsissa että Norjassa, joissa lajin historia ja kannan kehitys on hyvin samankaltainen kuin Suomessa, hirvipopulaation on todettu jakautuneen geneettisesti erilaistuneisiin alapopulaatioihin ja geenivirran huomattu olevan rajoittunutta eri alueiden välillä. Suomessa hirvipopulaation geneettistä muuntelua on tutkittu todella vähän. 80-luvulla täälläkin herättiin tutkimaan voimistuvan metsästyksen mahdollisia haittavaikutuksia hirven perimään, mutta innostus loppui kuitenkin lyhyeen. Syinä tähän olivat varhaisin menetelmin havaittu vähäinen muuntelu ja ennen kaikkea yleinen käsitys siitä, että geneettisillä tekijöillä ei olisi merkitystä hirven kaltaisen eläinlajin menestymiselle (Nygren 2009). Suomalaisten hirvien DNA-näytteitä on ollut mukana mm. kansainvälisissä hirven fylogeografiaa selvittäneissä tutkimuksissa, minkä ansiosta on muodostunut jonkinlainen käsitys hirviemme geneettistä suhteista naapurimaiden kantoihin (esim. Hundertmark 2002a). Näissä tutkimuksissa on saatu myös lisätietoa esimerkiksi hirven kolonisaatiohistoriasta Fennoskandiassa. Molekyylimenetelmien myötä on saatu lisää varmuutta käsitykselle siitä, että laji on 16
19 saapunut Suomeen kahdesta suunnasta: sekä idästä Venäjältä että Keski-Euroopasta Skandinavian kautta pohjoisesta (Hundertmark ym. 2002a, Kholodova ym. 2005, johon viitannut Nygren 2009). Näytemäärät Suomen osalta ovat näissä tutkimuksissa kuitenkin olleet varsin pienet. 1.3 Tämän tutkimuksen tarkoitus Hirvi on saaliin määrän ja arvon perusteella selvästi Suomen tärkein riistaeläinlaji (Aarnio & Härkönen 2007) ja myös yksi tutkituimmista. Tietoa lajin ekologiasta ja kannan kehityksestä on vuosikymmenten aikana kertynyt runsaasti (Nygren 2009), mutta valitettavasti nykyaikaista tietämystä Suomen hirvipopulaation geneettisestä monimuotoisuudesta tai rakenteesta ei juuri ole olemassa. Näiden piirteiden tunteminen on kuitenkin tärkeää, sillä ne vaikuttavat sekä populaatioiden nykyiseen elinvoimaisuuteen että tulevaisuuden selviytymismahdollisuuksiin (Frankham ym. 2002). Erityisen merkittävää se on voimakkaasti verotettujen riistalajien kohdalla, koska ne altistuvat juuri metsästyksestä aiheutuville negatiivisille geneettisille seurauksille (Allendorf ym. 2008). Tässä pro gradu-tutkielmassa tarkoituksenani on tutkia mikrosatelliittien avulla Suomen hirvien geneettistä muuntelua (määrää ja alueellista jakautumista) sekä populaatiorakennetta, ja arvioida niihin vaikuttavia tekijöitä. Arvioita pyritään esittämään geneettisin menetelmin myös efektiivisestä populaatiokoosta ja populaation sisäisestä geenivirrasta valottamaan kannan tulevaisuuden näkymiä. Lisäksi etsitään todisteita siitä, onko hirven historiallinen populaatiokoon romahtaminen johtanut geneettiseen pullonkaulaan ja sitä myötä sukusiitokseen Suomessa. 17
20 2. AINEISTO JA MENETELMÄT 2.1 Näytteet Tutkimusta varten valittiin Elintarviketurvallisuusviraston (Evira) kala- ja riistaterveyden tutkimusyksikön satojen yksilöiden varastosta verinäytteet yhteensä 116 hirvestä. Näytteet olivat peräisin metsästyskaudella kaadetuista hirvistä. Yksilöt valittiin edustamaan populaatioita kuudelta eri alueelta: Lounais-Suomesta, Pohjanmaalta, Karjalasta, Kainuusta sekä Itä- ja Länsi-Lapista (Kuva 4). Lisäksi työhön saatiin mukaan 14 kappaletta talvella hirvieläinten satelliittipannoitusprojektin yhteydessä kerätyistä verinäytteistä, joista kahdeksan oli peräisin Porkkalanniemeltä ja kuusi Kuhmosta. Porkkala luokiteltiin omaksi alueekseen, mutta Kuhmon näytteet lisättiin täydentämään Kainuun populaatiota (Kuva 4). Tutkittavista yksilöistä oli tiedossa sukupuoli, näytteenottopaikkakunta sekä pantahirvistä myös näytteenottopaikan tarkat koordinaatit (Liite I). Käytettyjen näyteyksilöiden lukumäärä on esitetty alueittain ja sukupuolittain taulukossa 1 Taulukko 1. Hirvinäytteiden alueellinen jakautuminen sukupuolittain. Alue Uros Naaras Yhteensä Lounais-Suomi Porkkala Pohjanmaa Karjala Kainuu Itä-Lappi Länsi-Lappi Yhteensä DNA:n eristys DNA:n eristäminen verinäytteistä tapahtui käyttäen UltraClean TM BloodSpin TM Kiteristyskittiä (Mo Bio Laboratories) valmistajan ohjeita noudattaen. Eristetty DNA säilytettiin jääkaapissa (+4 C) liuotettuna 200 μl:aan eristyskitin puskuriliuosta. 18
21 Kuva 4. Tutkimuksessa käytettyjen hirven verinäytteiden keräyspaikat populaatioittain. 2.3 Mikrosatelliitit Tässä työssä tutkittiin 10 eri mikrosatelliittilokusta, joista yksi, CSSM003, on alun perin kehitetty nautaeläimille (Bovinae) ja loput yhdeksän peuralle (Rangifer tarandus). Käytetyt lokukset ja niiden alukkeet on esitetty taulukossa 2. Kaikkien lokusten toimivuus hirvellä on testattu aikaisemmissa tutkimuksissa (Wilson ym. 1997, Røed & Midthjell 1998). Mikrosatelliittien alukkeet tilattiin kahdelta valmistajalta: kaikki reverse-alukkeet sekä FAM-leimatut forwardalukkeet Oligomeriltä, ja kaikki muut forward-alukkeet Applied Biosystemsiltä. Kaikista alukkeista tehtiin 10 μm:n liuokset lisäämällä steriiliä vettä. 19
22 Taulukko 2. Mikrosatelliittilokusten toistojaksot ja alleelikoot (bp=emäsparia), alukkeiden sekvenssit ja forward-alukkeen leima sekä lähdeartikkelit. F= forward, R= reverse. Alleelikoko (bp) Sekvenssi (5-3 ) Leima Lähde PET Wilson ym. (1997) Lokus Toistojakso RT5 (GT) F: CAG CAT AAT TCT GAC AAG TG R: AAT TCC ATG AAC AGA GGA G RT9 (GT) F: TGA AGT TTA ATT TCC ACT CT R: CAG TCA CTT TCA TCC CAC AT RT23 (GT) F: GGC CAT TGG GTA GTC TCC R: AGC CTC CCT GAG TGC TCT RT24 (GT) 3 A(GT) F: TGT ATC CAT CTG GAA GAT TTC AG R: CAG TTT AAC CAG TCC TCT GTG RT27 (GT) F: CCA AAG ACC CAA CAG ATG R: TTG TAA CAC AGC AAA AGC ATT RT30 (GT) F: CAC TTG GCT TTT GGA CTT A R: CTG GTG TAT GTA TGC ACA CT NVHRT01 (GT) 7 GC(GT) F: GCA GTC TTC CCC TTT CTT R: GAT TGC AGA GTT GGA CAC TA NVHRT21 (GT) 16 (GC) 4 GT F: GCA GCG GAG AGG AAC AAA AG R: GGG GAG GAG CAG GGA AAT C NVHRT24 (TG) 2 (TA) 2 (CA) 16 (TA) F: TGT GGA CTA TAG GGA GC (CA) 3 (TA) 5 R: GTG TAC AAA AAG TGA TTG AGT CSSM003 GGGG.(TG) 4.TA.(TG) 9. (AG) 6.CN.(GA) 5.AA.GA F: GTA CCT TAA GGT CAA GGG CTT TCT R: TGG GTC CAA TTG AGA ATC TTC ATG VIC Wilson ym. (1997) FAM Wilson ym. (1997) NED Wilson ym. (1997) NED Wilson ym. (1997) FAM Wilson ym. (1997) PET Røed & Midthjell (1998) VIC Røed & Midthjell (1998) FAM Røed & Midthjell (1998) VIC Moore ym. (1994), Røed (1998) 2.4 PCR PCR-reaktiot toteutettiin 10 μl:n tilavuuksissa, joihin sisältyi: 1-2 μl templaatti-dna:ta, 1 μl 10 x PCR-puskuria (10 mm Tris-HCL, ph 8,0, 50 mm KCL, 1mM EDTA, 0,1 % Triton X- 100, 50 % glyseroli), 1 μl dntp-liuosta (2 mm / deoksinukleotidi), 0,4 μl molempia alukkeita (10 μm), 0,3-0,6 μl MgCl 2 (1,5-3,0 mm), 0,6 μl DNA-polymeraasia (Biotools) ja 4,74-5,74 steriiliä vettä lokuksesta riippuen. Reaktioseosten pipetoinnit suoritettiin aina jäähauteessa. Käytössä olleita PCR-laitteita oli kaksi: Applied Biosystems 2720 Thermal Cycler ja Techne TC-3000G. Tarkat PCR-olosuhteet on esitelty lokuksittain taulukossa 3. Taulukko 3. Mikrosatelliittilokusten PCR-olosuhteet. Alukkeiden kiinnittymislämpötila ( C) DNAtemplaatti (μl) Lokus MgCl 2 (mm) PCR-laite RT5, 9, 23, 24, 27 & 30 50/54 2,0 1 AB 2720 Thermal Cycler NVHRT ,0 1 2 AB 2720 Thermal Cycler NVHRT ,5 2 Techne TC-3000G NVHRT ,5 2 Techne TC-3000G CSSM ,0 1 AB 2720 Thermal Cycler Käytetty PCR-ohjelma rakentui seuraavista vaiheista: aloitus alkudenaturaatiolla (94 C, 5 min), jonka jälkeen käynnistyi 35 kertaa toistettava kolmivaiheinen sykli, joka alkoi denaturaatiosta (94 C, 30 s), jatkui alukkeiden kiinnittymisvaiheella (45-55 C, 30 s) ja loppui alukkeiden pidentymisvaiheeseen. (72 C, 30 s). Viimeisen syklin jälkeen ohjelma jatkui vielä 20
23 7 minuuttia 72 C:ssa, ja päättyi jäähdytykseen 4 C:een. Lokusten NHVRT21 ja NVHRT24 tapauksissa PCR-profiilia muokattiin niin, että alukkeiden pidentymisvaihe oli 45 s ja lopun jatkoaika 10 min. NVHRT01-lokuksen kohdalla monistus suoritettiin käyttäen ns. Touchdown-ohjelmaa, joka poikkesi edellä kuvatusta perusohjelmasta siten, että alukkeiden kiinnittymisvaiheen lämpötila oli ensimmäisen kahden syklin ajan 53 C, josta se laski 2 C:n verran kahden syklin välein 48 C:een, minkä jälkeen kiinnittymislämpötila oli 25 syklin ajan 45 C. Epäonnistumisten takia joidenkin lokusten ja näytteiden kohdalla monistus jouduttiin uusimaan tai olosuhteita säätämään. Valmiit PCR-tuotteet säilytettiin jääkaapissa agaroosielektroforeesiin asti, jonka jälkeen ne siirrettiin pakastimeen odottamaan kapillaarielektoforeesia. 2.5 Agaroosielektroforeesi Mikrosatelliittien monistuminen tarkistettiin elektroforeesin avulla ajamalla PCR-tuotteet 1,5 % agaroosigeelillä. Itse geelin valmistus aloitettiin lisäämällä 1,2 g agaroosia 85 ml:aan 0,5 x TBE-liuosta (10 mm EDTA, 0,45 M Tris, 0,45 M boorihappo, steriili H 2 O), ja kiehauttamalla sitten liuos mikroaaltouunissa. Kun liuos oli jäähdytetty tämän jälkeen noin 60 C:een, siihen lisättiin etidiumbromidia 3,5 μl, minkä jälkeen se kaadettiin ajokelkkaan (2-4 kampariviä). Geelin jähmetettyä kammat otettiin irti, ja geeli nostettiin ajolaitteeseen 0,5 x TBEajopuskuriliuokseen. Geelin kaivoihin pipetoitiin 4 μl jokaista näytettä sekoitettuna kahteen μl:aan DNA:n latauspuskuria (47 % steriili glyseroli, 0.26 M EDTA ja bromofenolisininen). Kunkin kaivorivin viimeiseen kaivoon pipetoitiin 100 bp molekyylikokostandardia (New England BioLabs). Geeliä ajettiin 30 minuuttia 100 voltin jännitteellä, minkä jälkeen ajon tulos tarkistettiin UV-pöydällä, jossa onnistuneet geelit myös valokuvattiin. 2.6 Kapillaarielektroforeesi ja näytteiden genotyyppien määritys Kapillaarielektroforeesia varten valmistettiin ensin ajoseos, johon pipetoitiin 800 μl formamidia ja 25 μl kokostandardia (GeneScan 500 Liz, Applied Biosystems). Tämä jaettiin 96- kuoppaiselle kuoppalevylle siten, että jokaiseen kuoppaan tuli seosta yhteensä 8,2 μl. PCRtuotetta pipetoitiin kuoppiin lokuksesta ja näytteestä riippuen 0,5/1,0/1,5/2,0 μl. Samaan kuoppaan voitiin lisätä useamman kuin yhden lokuksen PCR-tuotetta, mutta tällöin varmistettiin, että käytettävät forward-alukkeiden leimat olivat erilaiset (Taulukko 2). Pipetoinnit tehtiin jäähauteessa. Kapillaarielektroforeesiajo suoritettiin ABI PRISM 3730 DNA Analyzer laitteistolla (Applied Biosystems). 21
24 Kapillaarielektroforeesin tulokset tarkistettiin, ja näyteyksilöiden genotyypit määritettiin käyttämällä GeneMapper-ohjelmaa (GeneMapper Software, v. 4.0, Applied Biosystems). Genotyyppitulkinnan epäonnistuttua tai jäädessä jonkun näytteen kohdalla epävarmaksi suoritettiin PCR-monistus ja sitä seuraavat vaiheet tarvittaessa uudelleen. Valmis genotyyppidata koottiin taulukoksi, jossa ilmenee kunkin yksilön genotyyppi lokuksittain (ei esitetty). 2.7 Aineiston analysointi Genotyyppiaineisto tarkistettiin alueittain Micro-Checker-ohjelman (v , van Oosterhout 2004) avulla mahdollisten nolla-alleelien sekä alleelien määrityksessä tapahtuneiden virheiden havainnoimiseksi. Lisäksi aineisto muunnettiin tavallisimpien analyysiohjelmistojen tunnistamiin muotoihin käyttäen hyväksi Microsatellite analyzer-ohjelmaa (v.4.0, Dieringer & Schlötterer 2003) Hardy-Weinberg-tasapaino ja kytkentäepätasapaino Hardy-Weinbergin tasapainossa (HW-tasapaino) on kyse homo- ja heterotsygoottien odotetuista osuuksista suuressa, satunnaisesti pariutuvassa populaatiossa, kun alleefrekvenssit tunnetaan. Hardy-Weinbergin lain mukaan alleelifrekvenssien tulisi pysyä muuttumattomina sukupolvesta toiseen ilman migraatiota, mutaatioita tai valintaa (Hartl & Clark 1997, Beebee & Rowe 2004). HW-tasapainoa hirvillä tutkittiin Genepop v ohjelmalla (Raymond & Rousset 1995, Rousset 2008) käsittelemällä aineistoa sekä yhtenä kokonaisena populaationa että erillisinä seitsemänä populaationa. Ohjelma laskee harhattoman todennäköisyyden sille, että tietty lokus on HW-tasapainossa Markovin ketju-menetelmää käyttäen, ja tekee näillä arvoilla merkitsevyystestauksen Fisherin menetelmällä yli kaikkien lokusten. Markovin ketjuissa käytettiin ohjelman antamia oletusarvoja (Dememorization number=1000, Number of batches=100, Number of iterations per batch =1000). Populaatiogeneettisissä analyyseissä yleisenä perusolettamuksena on, että lokusten alleelit ovat kytkentätasapainossa eli periytyvät toisistaan riippumatta. Vastaavasti kytkentäepätasapainossa eri lokusten alleelit esiintyvät yhdessä useammin kuin on todennäköistä. Taustalla voi olla esimerkiksi valinta, geneettinen ajautuminen, sukusiitos, hybridisaatio tai pullonkaula (Hedrick 2005). Kytkentäepätasapainoa tarkasteltiin Genepop v ohjelmalla (Raymond & Rousset 1995, Rousset 2008) suorittamalla lokusten parittaiset vertailut koko aineistossa ja 22
25 vielä erillisinä populaatioina käsiteltynä. Analyysissä ohjelma laskee Markovin ketjumenetelmällä kytkentätasapainon todennäköisyyden kaikille lokuspareille populaatioiden sisällä käyttäen G-testiä, ja sen jälkeen merkitsevyystestauksen Fisherin menetelmällä yli kaikkien populaatioiden. Myös tässä analyysissä Markovin ketjuille käytettiin ohjelman oletusarvoja (Dememorization number=1000, Number of batches=100, Number of iterations per batch =1000) Populaatioiden geneettinen muuntelu ja sukusiitos Geneettistä muuntelua on mahdollista kuvata usean eri muuttujan avulla. Yleisin muuntelun mitta on odotettu heterotsygotia (H e ), eli geenidiversiteetti, joka on alleelifrekvensseistä laskettu heterotsygoottien osuus odotettu olettaen, että populaatio noudattaa Hardy-Weinbergin tasapainoa (Beebee & Rowe 2004). Odotettu heterotsygotia voidaan laskea käyttäen kaavaa: H n = (1 n 1 k E pi jossa n on alleelien lukumäärä ja pi on alleelin i frekvenssi. Tässä työssä H e laskettiin lokuksittain ja lokusten yli kullekin populaatiolle käyttäen Arlequin v.3.11-ohjelmaa (Excoffier ym. 2005). Lisäksi geenidiversiteetti laskettiin erikseen molemmille sukupuolille koko aineistosta FSTAT v ohjelmalla (Goudet 2001). i= 1 2 ) Muuntelua voidaan tarkastella myös yksinkertaisesti lokuksessa esiintyvien alleelien lukumäärää vertailemalla, mutta tällöin erilaiset näytekoot aiheuttavat ongelmia. Vertailukelpoisempi arviointi on mahdollista näytekoosta riippumattoman alleelirunsauden (A, allelic richness) avulla: 2N Ni n i 2n A = 1 i= 1 2N 2n jossa n on lokuksen pienin yksilömäärä näytteen sisällä ja Ni on alleelin i lukumäärä 2N yksilön joukossa. Hirville jokaisen lokuksen alleelien lukumäärä, sekä lokusten että populaation alleelirunsaudet laskettiin FSTAT v ohjelmalla (Goudet 2001), jossa erikokoisten näytemäärien aikaansaama virhe A:n estimaattiin korjattiin rarefaktio-menetelmällä. Yleensä n standardisoidaan pienimmän näytekoon mukaan. 23
26 Sukusiitoskerroin, eli F IS, mittaa alapopulaatioiden poikkeamaa HW-tasapainosta. Negatiiviset F IS :n arvot viittaavat heterotsygotian, ja positiiviset vastaavasti homotsygotian ylimäärään, mikä voi olla sukusiitoksesta johtuvaa. Sukusiitoskerroin voidaan laskea kaavalla F IS HO = 1, HS jossa H O on heterotsygoottien havaittu osuus ja H S populaation geenidiversiteetti. Myös F IS :n arvot laskettiin käyttämällä FSTAT v ohjelmaa (Goudet 2001). FSTAT v ohjelman laskemia alleelifrekvenssejä käytettiin laskettaessa ns. privaattialleeleja, eli alleeleja, jotka esiintyvät ainoastaan yhdessä populaatiossa Populaatiorakenne Suomen hirvipopulaation sisäistä geneettistä rakennetta lähdettiin tutkimaan etsimällä aineistosta geneettisiä alapopulaatioita bayesilaiseen ryhmittelymenetelmään perustuvalla Structure v ohjelmalla (Pritchard ym. 2000). Analyysi ei vaadi ennakkotietoja tutkittavan populaation rakenteesta, vaan ohjelma olettaa mallin, jonka mukaan aineistossa on K populaatiota (jossa K voi olla tuntematon), joita luonnehtii kussakin lokuksessa omanlaisensa alleelifrekvenssit. Yksilöt pyritään jakamaan genotyyppiensä mukaisesti populaatioihin, joita voi olla jopa kaksi tai useampia, jos yksilön perimä on sekoitus eri populaatioille tyypillisistä genotyypeistä. Samalla ohjelma arvioi näiden mahdollisten populaatioiden alleelifrekvenssejä (Pritchard ym. 2000). Tässä tutkimuksessa käytettiin Structure-mallia, jossa yksilön perimä voi olla sekoitus useammasta populaatiosta ja alleelifrekvenssit voivat korreloida keskenään populaatioiden välillä. Ohjelma ajettiin yhteensä kymmenellä K:n arvollä (K=1-10) suorittamalla kaksi iteraatiota per K. Markov Chain Monte Carlo- menetelmässä käytettiin alkuvaiheessa (burnin) ja loppuvaiheessa toistoa. Ohjelma laskee mallin mukaisesti K:n eri arvoilla logaritmiset uskottavuusarvot, joista korkeimman saaneen K:n pitäisi selittää populaatiorakennetta parhaiten. Vertailun vuoksi populaatioiden määrää arvioitiin Structure-ohjelman antamista tuloksista myös käyttämällä Evannon ym. (2005) esittämää menetelmää, joka perustuu ΔK:n laskemiseen eli uskottavuusarvofunktion toiseen derivaattaan suhteessa K:hon. Aluksi kunkin K:n arvon iteraatioiden uskottusvuusarvoista lasketaan keskiarvot (± keskihajonta), minkä jälkeen lasketaan uskottuusarvojakauman derivaatta (keskiarvo ± keskihajonta) kaavalla L (K) = L(K) 24
27 L(K 1). Sitten otetaan uskottavuusarvojakauman toisen derivaatan itseisarvot: L (K) = L (K + 1) L (K). ΔK saadaan lopuksi kaavalla ΔK = m L (K) /s[l(k)]. Se K:n arvoista, joka saa suurimman ΔK:n arvon, tulisi vastata parhaita todellista K:ta eli aineistosta löytyvien geneettisten ryhmien määrää (Evanno ym. 2005). Populaatioiden määrää arvioitiin myös toisella bayesilaista menetelmää käyttävällä ohjelmalla BAPS v.3.2 (Bayesian Analysis of Population Structure, Corander ym. 2003). Structureohjelman tavoin se pyrkii löytämään aineistosta K-ryhmää yksilöitä, jotka muistuttavat toisiaan niin paljon kuin mahdollista, mutta käyttää kuitenkin ryhmittelyssä erilaista algoritmiä kuin Structure-ohjelma. BAPS-ohjelmasta käytettiin Clustering of individuals -vaihtoehtoa, jossa alapopulaatioiden oletetaan olevan Hardy-Weinberg tasapainossa ja lokusten toisiinsa kytkeytymättömiä. Eri alueiden välisten maantieteellisten etäisyyksien mahdollista vaikutusta hirvien geneettiseen erilaistumiseen, eli etäisyysisolaatiota, tutkittiin käyttämällä ohjelmaa SPAGeDi (Spatial Pattern of Genetic Diversity, Hardy & Vekemans 2002). Sen avulla aineistosta laskettiin yksilöpareille mikrosatelliittialleeleihin perustuvat sukulaisuusasteet (kinship coefficient, Loiselle ym. 1995) ja koordinaattitietoihin pohjautuvien maantieteellisten etäisyyksien logaritmit, joiden välistä yhteyttä ohjelma tutki jakamassaan kymmenessä luokassa regression avulla. Etäisyyksien logaritmeja käytettiin siitä syystä, että dispersaalia tapahtuu luonnossa kaikkiin suuntiin. Ohjelma testasi lisäksi estimaattien tilastollista merkitsevyyttä permutaation avulla. Ainoastaan pantahirvistä oli saatavilla valmiiksi tarkat koordinaattipisteet näytteenottopaikoista. Tästä syystä muille 116 yksilölle sijainti päätettiin asettaa näytteenottokunnan keskuksen mukaan (Liite I). Koordinaatit saatiin Maanmittauslaitoksen Kansalaisen karttapaikkaverkkopalvelusta ( F-statistiikka on tärkeä tilastollinen metodi tarkasteltaessa populaatioiden erilaistumista. Siinä heterotsygotiaa mitataan kolmella hierarkkisella tasolla: yksilö, alapopulaatio ja populaatio. Näistä tasoista laskettavista erilaisista indekseistä F ST, eli fiksaatioindeksi, kuvaa satunnaisesta geneettisestä ajautumisesta johtuvaa alapopulaatioiden vähentynyttä heterotsygotiaa. Se siis kertoo, onko populaatiolla erilaistunut alapopulaatiorakenne vai onko se Hardy-Weinbergin tasapainossa (Hartl & Clark 1997). F ST saadaan käyttäen kaavaa: 25
28 F ( H H ) H T S ST =, T jossa H S on lokusten yli laskettu odotettu heterotsygotia alapopulaatioissa, ja H T on lokusten yli laskettu koko populaation heterotsygotia. F ST saa aina pelkästään positiivisia arvoja. F-statistiikassa geenimerkkien mutaatiomalliksi oletetaan IAM (infinite allele model), rajattoman alleelimäärän malli, jonka mukaan uusi mutaatio tuottaa joka kerta uuden alleelin. Sen sijaan R-statistiikkassa mutaatioiden oletetaan noudattavan vaiheittaista mutaatiomallia, SMM (stepwise mutation model), jossa mutaatio joko lisää tai poistaa yhden toistoyksikön alleeleista (Estoup 2002). F ST :n perustuessa eroihin alleelifrekvensseissä, R ST pohjautuu alleelikokojen vaihteluun: R ST ( S SW) =, S jossa S on keskimääräinen alleelikokojen erojen neliösumma koko populaation tasolla, ja S W on alapopulaatioiden sisäinen keskimääräinen alleelikokojen neliösumma. Tässä tutkimuksessa laskettiin sekä F ST - että R ST -arvot populaatioiden välille pareittain käyttämällä avuksi Arlequin v.3.11-ohjelmaa (Excoffier ym. 2005), joka myös testaa vertailut tilastollisesti permutaatioilla (110 kpl). Laskelmat suoritettiin sekä alkuperäiselle seitsemän populaation asettelulle että myös Structure v ohjelman tulosten perusteella muodostetuille populaatioille. Suomen hirvipopulaation geneettistä rakennetta tutkittiin myös molekyylivarianssianalyysillä, eli AMOVA:lla (Excoffier ym. 1992), joka mittaa geenifrekvenssien varianssia. AMOVA selvittää, kuinka tämän varianssin kokonaismäärä on jakautunut sekä populaatioiden sisälle että niiden välille, ja sillä on mahdollista testata erilaisia populaatiorakenteita ennalta määritetyissä ryhmissä. Analyysiin voi sisällyttää myös alleelien geneettisen etäisyyden, ja se laskee populaatiorakenteen tasoille fiksaatioindeksit, joiden merkitsevyys testataan permutaatioilla (Beebee & Rowe 2004). Hirvien mikrosatelliittiaineistolle AMOVA tehtiin Arlequin v ohjelmalla (Excoffier ym. 2005) kahdella eri tavalla: käyttämällä hyväksi pelkästään genotyyppifrekvenssejä (F ST ), sekä hyödyntämällä niiden lisäksi myös alleelien kokoeroja (R ST ). Analyysi tehtiin sekä alkuperäiselle seitsemän populaation asettelulle että myös Structure v ohjelman tulosten perusteella muodostetuille neljälle populaatiolle (ks.3.3). Permutaatioiden määrä kaikissa analyyseissä oli 1023 kappaletta. 26
29 Eri alueiden hirvipopulaatioiden geneettisiä suhteita kuvattiin sukupuulla, joka rakennettiin TFPGA v.1.3-ohjelmalla (Miller 1997) käyttäen UPGMA-ryhmittelyalgoritmia (unweighted pair group method using arithmetic averages) ja Nein (1978) geneettistä etäisyyttä. Ohjelma testaa rakentamansa puukaavion luotettavuutta uudelleenotannalla ns. saappaanraksimenetelmällä (bootstrap method, 1000 kpl). Tuloksen ohjelma ilmoittaa puun kunkin haarautumiskohdan saappaanraksiarvona eli osuutena niistä uudelleenotannoista, joissa esiintyvät samat haarat. UPGMA-puun lisäksi yksittäisten monilokusgenotyyppien geneettisiä etäisyyksiä visualisoitiin faktorikorrespondenssianalyysillä (factorial correspondence analysis) Genetix v.4.2- ohjelman (Belkhir ym. 1999) avulla. Tämä monimuuttujamenetelmä perustuu odotettuja ja havaittuja alleelifrekvenssejä vertaamalla laskettuun χ 2 -arvoon, jonka pohjalta muodostetaan sitä selittävät uudet, genotyypeille osoitettavat muuttujat eli faktorit. Muodostamalla pistediagrammi χ 2 -arvoa parhaiten selittävistä faktoreista voidaan kuvata geneettisen muuntelun jakautumista yksilöittäin kaksi- tai kolmiulotteisessa avaruudessa. Tämä analyysi suoritettiin Structure-ohjelman perusteella muodostetuille neljälle populaatiolle (ks. 3.3) Geenivirta Suomen eri alueiden välistä geenivirtaa arvioitiin Genepop v ohjelmalla (Raymond & Rousset 1995, Rousset 2008) Bartonin ja Slatkinin (1986) kehittämällä privaattialleelimenetelmällä, jossa arvioidaan vaikuttavien migranttien määrää (Nm, Slatkin 1985) sukupolvessa. Ohjelma ottaa huomioon näytekoon Slatkinin (1985), ja laskee Nm:n estimaatin Barton & Slatkinin (1986) esittämien privaattialleelien frekvenssin ja Nm:n välisten. regressiosuorien avulla. Populaatioiden välinen Nm on mahdollista laskea myös parittaisten F ST - arvojen perusteella. Arlequin ohjelma laskee näistä arvoista ns. M-arvomatriisin, jossa M on 2Nm (Excoffier ym. 2005). Aineistosta pyrittiin myös havainnoimaan ensimmäisen polven migrantteja, eli yksilöitä, jotka ovat syntyneet jossain muussa kuin siinä populaatiossa, jossa ne on havaittu. Tämä tehtiin GeneClass2 v. 2.0-ohjelmalla (Piry ym. 2004) käyttäen Rannalan & Mountainin (1997) luokittelumenetelmää, jossa yksilöille laskettiin kahden uskottavuusarvon välinen suhde (L_home/L_max), jonka avulla migrantti voidaan tunnistaa, kun potentiaalinen lähdepopulaa- 27
30 tio on edustettuna. Tunnistuksen todennäköisyyttä testattiin Monte Carlo-menetelmällä (Paetkau ym. 2004) yhteensä 1000 simulaatiolla alfan arvon ollessa 0,001. Arlequin ohjelmaa (Excoffier ym. 2005) käytettiin suoritettaessa aineistolle ns. kohdennustesti (assignment test), joka perustuu Paetkaun ym. (1995, 1997) ja Waser & Strobeckin (1998) esittämiin algoritmeihin. Tällä testillä aineistosta voidaan yrittää selvittää yksilöiden alkuperää, kun potentiaaliset lähdepopulaatiot ovat tiedossa. Testi määrittää alleelifrekvenssejä hyödyntäen monilokusgenotyypin log-uskottavuusarvot jokaiselle yksilölle jokaisessa populaatiossa olettaen, että kyseinen yksilö on oikeasti lähtöisin populaatiosta, johon se luokiteltu Efektiivinen populaatiokoko Tässä tutkielmassa efektiivisen populaatiokoon laskeminen suoritettiin LDNe v ohjelmalla (Waples & Do 2007), joka käyttää Waplesin (2006) korjattua menetelmää Hillin (1981) lokusten kytkentäepätasapainoon perustuvasta laskutavasta, eikä vaadi näytteitä peräkkäisistä sukupolvista. Structure v ohjelman tulosten mukaan muodostetuille neljälle populaatiolle (ks. 3.3) laskettiin kolme eri raja-arvioihin (alleeli lasketaan, jos sen frekvenssi on suurempi kuin 0,01/0,02/0,05) perustuvaa arviota N e :sta olettaen, että populaatiot ovat satunnaisesti lisääntyviä. Lisäksi ohjelma laskee arvioille 95 %:n luottamusvälin ns. linkkuveitsimenetelmällä (jack knife method) Pullonkaula Suomen hirvikannan romahtamisen aiheuttaman mahdollisen pullonkaulan merkkien havaitsemiseen käytettiin Bottleneck v ohjelmaa (Piry ym. 1999), joka analyysissään laskee näytekoon huomioiden jokaiselle populaatiolle ja lokukselle odotetun heterotsygotian jakauman sillä oletuksella, että populaatiot ovat mutaatioiden ja satunnaisajautumisen suhteen tasapainossa. Jakauma lasketaan simuloimalla koalesenssia kolmen mutaatiomallin mukaan: IAM, SMM ja TPM (Two-Phased Mutation Model) eli kaksivaiheinen mutaatiomalli, joka on mikrosatelliittien kanssa käytettäväksi suositeltu IAM- ja SMM-mallin sekoitus. Ohjelma laskee tästä jakaumasta heterotsygotian keskiarvon tasapainotilassa ja tätä verrataan Wilcoxonin testin avulla (1000 toistoa) havaittuun heterotsygotiaan selvittämään mahdollista geenidiversiteetin yli- tai alimäärää. Populaatio on todennäköisesti kokenut pullonkaulan, jos 28
31 havaittu heterotsygotia on tilastollisesti merkitsevästi suurempi kuin tasapainoheterotsygotia (Luikart & Cornuet. 1998). Tässä tutkimuksessa SMM:n osuudeksi TPM-mallissa asetettiin suosituksen mukaisesti 95 % (Piry ym.1999). Lisäksi Bottleneck v esittää alleelifrekvenssien jakauman alleelifrekvenssiluokissa ilmoittaen samalla noudattavatko ne tasapainopopulaatiossa esiintyvää L-muotoista jakaumaa (Luikart ym. 1998). Mahdollista pullonkaulaa voi myös tutkia laskemalla Garza-Williamsonin-indeksi (G-Windeksi): k G W =, R +1 jossa k on alleelien lukumäärä lokuksessa, ja R on alleelien toistojaksojen maksimi- ja miniarvojen välinen erotus (allelic range) (Garza & Williamson 2001, Excoffier ym. 2005). Jos populaation on kokenut pullonkaulan, tulisi k:n olla pienentynyt suhteessa enemmän kuin R, jolloin G-W saa matalia arvoja, ja vastaavasti vakaissa populaatioissa G-W:n arvo on lähellä ykköstä (Garza & Williamson 2001). G-W-indeksi laskettiin Arlequin v ohjelmalla (Excoffier ym. 2005). 29
32 3. TULOKSET MicroChecker-ohjelman avulla suoritetussa tarkastuksessa havaittiin useassa populaatiossa lokuksissa NVHRT01 ja RT30 yhteen tai useampaan nolla-alleeliin viittaavaa tilastollisesti merkitsevää homotsygotian ylimäärää (p < 0,001). Lokuksen NVHRT01 nollaalleelifrekvenssi Oosterhoutin algoritmin mukaan (van Oosterhout ym. 2004) oli Karjalassa 0,083, Kainuussa 0,242, Lounais-Suomessa 0,257 ja Itä-Lapissa 0,230. Vastaavasti RT30:n frekvenssi oli Pohjanmaalla 0,259, Lounais-Suomessa 0,186 ja Länsi-Lapissa 0,126. Nämä kaksi lokusta päätettiin jättää pois laskettaessa HW-tasapainoa, kytkentäepätasapainoa ja aluekohtaisia sukusiitoskertoimia. 3.1 Hardy-Weinberg-tasapaino ja kytkentätasapaino Kahdeksaa lokusta tutkittaessa Suomen hirvet poikkesivat yhtenä kokonaisena populaationa HW-tasapainosta tilastollisesti merkitsevästi (χ 2 =, df = 16, p < 0,001) viitaten heterotsygoottien ylimäärään. Erillisinä populaatioina käsiteltynä tasapainosta poikkesivat Pohjanmaan (χ 2 =, df=16, p < 0,001), Karjalan (χ 2 =, df=16, p < 0,001) ja Kainuun (χ 2 =26,547, df=16, p=0,047) populaatiot. Seitsemän erillisen populaation yli tehdyissä kahdeksan lokuksen parittaisissa vertailuissa tilastollisesti merkitseviä kytkentäepätasapainoja ei löytynyt. Jos taas aineistoa käsiteltiin yhtenä kokonaisena populaationa, merkitsevä kytkentäepätasapaino havaittiin lokusten RT9 ja RT23 (p=0,030) sekä CSSM003 ja NVHRT21 (p=0,012) välillä. 3.2 Lokusten ja populaatioiden geneettinen muuntelu Kaikki käytetyt kymmenen lokusta olivat varsin muuntelevia populaatioiden yli mitattuna H e :n, alleelien lukumäärän ja alleelirunsauden perusteella (Taulukko 4). Muuntelevimmissa lokuksissa RT5, RT24, RT27 ja RT30 H e :n arvot vaihtelivat välillä 0,768 0,828, alleelirunsauden ollessa välillä 6,2 7,1 ja alleelien lukumäärän yltäessä kappaleeseen. Matalimmat odotetun heterotsygotian ja alleelirunsauden arvot löytyivät molemmat lokuksesta RT23 (H e =0,522, A=2,5). Vähiten alleeleja havaittiin lokuksista CSSM003 ja NVHRT24; molemmista vain viisi kappaletta. Lokuskohtaiset alleelifrekvenssit on esitetty liitteessä II. 30
33 Taulukko 4. Lokusten muuntelu kolmella muuttujalla mitattuna. He = odotettu heterotsygotia (ka), A = alleelirunsaus (ka) pienimmän populaatiokoon mukaan suhteutettuna, F IS = sukusiitoskerroin. Lokus H e Alleelien lkm A F IS RT5 0, ,2-0,043 RT9 0, ,7 0,142 RT23 0, ,5-0,231 RT24 0, ,1-0,018 RT27 0, ,8 0,004 RT30 0, ,6 0,305 CSSM003 0, ,1 0,012 NVHRT01 0, ,6 0,47 NVHRT21 0, ,9-0,023 NVHRT24 0, ,4 0,059 Kaikkien kymmenen lokuksen yli tarkasteltuna eniten muuntelua havaittiin Karjalan populaatiossa sekä odotetussa heterotsygotiassa että alleelirunsaudessa (H e =0,747, A=5,1). Vastaavasti vähiten muuntelua löytyi Porkkalan populaatiosta (H e =0,682, A=4,3) Populaatiokohtaiset muuntelun määrää kuvaavat estimaatit on esitetty taulukossa 5. Sukupuolille erikseen lasketut H e :t eivät juuri poikenneet toisistaan; uroksilla (0,716) muuntelu oli aavistuksen pienempää kuin naarailla (0,732). Sukusiitoskertoimen arvot eri näytealueilla olivat hyvin lähellä nollaa (-0,046-0,0470). Koska lokuksissa RT30 ja NVHRT01 havaittiin mahdollisista nolla-alleeleista johtuvaa homotsygotian ylimäärää, laskettiin F IS -arvot populaatioille ainoastaan kahdeksan lokuksen yli (Taulukko 5). Privaattialleeleja löytyi eniten Kainuun populaatiosta: yhteensä neljä kappaletta. Lounais- Suomen sekä Itä- ja Länsi-Lapin populaatioista niitä havaittiin kustakin kolme. Itä-Suomessa privaattialleeleja oli kaksi kappaletta, Pohjanmaalla ainoastaan yksi ja Porkkalassa taas ei ainoatakaan. 31
34 Taulukko 5, Seitsemän tutkitun alueen geneettisen muuntelun määrä ja sukusiitos lokuksittain, He = odotettu heterotsygotia, A = alleelirunsaus, FIS = sukusiitoskerroin, Alimmilla riveillä odotetun heterotsygotian keskiarvo ja alleelirunsauden painotettu keskiarvo (10 lokusta) sekä sukusiitoskertoimen keskiarvo (8 lokusta). NE = ei estimoitu. Lounais- Suomi Porkkala Karjala Kainuu Pohjanmaa Itä- Lappi Länsi- Lappi RT5 H e 0,819 0,868 0,862 0,769 0,854 0,812 0,813 A 5,6 7,0 6,7 5,8 6,2 6,0 6,0 F IS -0,165-0,167 0,006-0,116-0,061 0,055 0,000 RT9 H e 0,745 0,775 0,729 0,723 0,712 0,816 0,740 A 4,5 4,0 4,1 4,6 4,6 5,5 4,78 F IS 0,156 0,371 0,021-0,139-0,003 0,188 0,246 RT23 H e 0,537 0,582 0,508 0,512 0,512 0,563 0,446 A 2,4 3,0 2,0 2,4 2,0 3,2 2,7 F IS -0,314-0,25-0,226-0,551-0,801 0,055 0,587 RT24 H e 0,765 0,667 0,869 0,741 0,855 0,921 0,810 A 5,8 4,7 7,8 6,0 6,6 8,7 6,3 F IS -0,017-0,135-0,155 0,102-0,060-0,009-0,082 RT27 H e 0,787 0,642 0,770 0,861 0,782 0,844 0,819 A 6,1 2,9 4,5 6,7 5,2 5,6 5,4 F IS 0,065 0,028-0,186 0,109 0,088-0,193 0,008 RT30 H e 0,892 0,692 0,871 0,782 0,721 0,630 0,788 A 7,6 6,4 7,3 5,8 4,6 3,7 5,6 F IS NE NE NE NE NE NE NE CSSM003 H e 0,537 0,567 0,556 0,645 0,542 0,508 0,558 A 3,3 2,9 2,9 3,4 3,2 2,0 2,9 F IS 0,071-0,355 0,234 0,003-0,055-0,189-0,124 NVHRT01 H e 0,683 0,567 0,765 0,749 0,417 0,591 0,702 A 3,7 2,9 4,7 4,4 2,5 3,6 4,4 F IS NE NE NE NE NE NE NE NVHRT21 H e 0,817 0,747 0,828 0,799 0,798 0,770 0,805 A 6,1 4,0 6,3 5,7 5,8 5,5 5,6 F IS -0,042 0,048 0,113-0,074-0,076 0,049-0,08 NVHRT24 H e 0,699 0,717 0,714 0,767 0,743 0,706 0,614 A 4,1 4,75 4,5 4,7 4,3 4,6 3,3 F IS 0,071 0,136-0,02 0,117 0,105-0,139-0,203 Kaikki H e 0,728 0,682 0,747 0,735 0,694 0,716 0,709 A 4,9 4,3 5,1 4,9 4,5 4,8 4,7 F IS -0,016-0,023 0,047-0,046 0,005-0,016 0, Populaatiorakenne Tutkittaessa aineistosta löytyvien geneettisten populaatioiden määrää antoi Structure-ohjelma suurimman tuen (uskottavuusarvon ka.= -4043,3) neljän populaation mallille (K=4, Kuva 4). Vastaavasti Evannon ym. (2005) menetelmää käyttäen suurimman tuen (korkein ΔK:n arvo, 17,423) sai kahden populaatio malli (Kuva 4). Ln-uskottavuusarvojen sekä yksilöiden alkuperästä piirretyn pylväsdiagrammin (Liite III) mukaisesti aineisto päätettiin jakaa uudelleen nel- 32
35 jään populaation: Etelä-Suomi (Lounais-Suomi + Porkkala), Pohjanmaa, Itä-Suomi (Karjala + Kainuu) ja Lappi (Itä-Lappi + Länsi-Lappi). ln-uskottavuusarvo ΔK ln-ua ΔK K Kuva 4. Havaitun genotyyppiaineiston ln-uskottavuusarvot (ln-ua) ja ΔK:t K:n eri arvoilla. Ln-ua:sta on kullekin K:n arvolle tehty kaksi iteraatiota, joista esitettynä keskiarvot. BAPS-ohjelman tulokset poikkesivat suuresti Structuren antamista: parhaimman tuen sai 10 alapopulaation malli (todennäköisyys 0,9565). Toiseksi suurimmalla, mutta huomattavasti ensimmäistä matalammalla todennäköisyydellä (0,04346) aineistoa selitti 11 alapopulaation malli. SpaGeDI-ohjelman tulosten mukaan hirviaineistosta on havaittavissa etäisyysisolaatiota (Kuva 5). Yksilöparien sukulaisuusasteen ja logaritmisten etäisyysluokkien välinen regressiosuoran kerroin oli negatiivinen (-0,0083) sekä tilastollisesti merkitsevä (p=0,000) kaksisuuntaisen (havaittu arvo odotettu arvo) permutaatiotestin mukaan. Toisin sanoen mitä suuremmaksi yksilöparien maantieteellinen etäisyys kasvoi, sitä suuremmaksi kasvoi niiden geneettinen etäisyys. Kolmessa ensimmäisessä etäisyysryhmässä (etäisyys alle 240 km) sukulaisuuskertoimen arvot olivat positiivisia, mutta neljännestä ryhmästä (etäisyys yli 300 km) eteenpäin kertoimet saivat negatiivisia arvoja. Ensimmäisessä luokassa, jossa yksilöt olivat muita luokkia keskimääräisesti geneettisesti samankaltaisempia (sukulaisuuskerroin=0,035, p=0,000), yksilöt sijaitsivat keskimäärin 38 kilometrin etäisyyden päässä toisistaan. Yksilöiden väliset etäisyydet olivat suurimmassa etäi- 33
36 syysluokassa keskimäärin 736 kilometriä ja sukulaisuuskerroin (-0.019) oli tilastollisesti merkitsevä (p=0,000). Tilastollisesti merkitsevä ero (p< 0,05) sukulaiskertoimista odotettuun arvoon löytyi myös ryhmistä 2, 5, 7 ja Sukulaisuuskerroin * * * * * * Maantieteellinen etäisyys (ln m) Kuva 5. Suomen hirvien keskimääräiset sukulaisuuskertoimet ja niiden keskivirheet kymmenessä maantieteellisessä etäisyysluokassa (logaritminen mitta-asteikko ln m). Tähdellä merkityissä ryhmissä sukulaiskertoimen arvo poikkesi merkitsevästi odotetusta. Seitsemän alueen hirvipopulaatioiden välillä havaittiin molekyylivarianssianalyysissä tilastollisesti merkitsevää (p=0,0000) erilaistumista sekä F ST :n (fiksaatioindeksi=0,041) että R ST :n (fiksaatioindeksi=0,050) arvojen perusteella laskettuna (Taulukot 6 ja 7). Populaatioiden välisen muuntelun osuus kokonaismuuntelusta oli R ST -arvoilla (4,95 %) laskettuna suurempaa kuin F ST -arvoilla (4,12 %), mutta molemmilla valtaosa havaitusta vaihtelusta löytyy populaatioiden sisältä. Taulukko 6. F ST -arvoihin perustuva populaatioiden välinen ja sisäinen muuntelu (AMOVA). Vaihtelun lähde Vapausasteet Neliösumma Varianssi Muuntelun osuus (%) Populaatioiden 6 53,086 0, ,12 välillä Populaatioiden ,026 3, ,88 sisällä Kokonaisvaihtelu ,112 3,59075 Fiksaatioindeksi (F ST ): 0,04121 (p = 0,0000) 34
37 Taulukko 7. R ST -arvoihin perustuva populaatioiden välinen ja sisäinen muuntelu (AMOVA). Vaihtelun lähde Vapausasteet Neliösumma Varianssi Muuntelun osuus (%) Populaatioiden ,831 17, ,95 välillä Populaatioiden , , ,05 sisällä Kokonaisvaihtelu , ,52776 Fiksaatioindeksi (F ST ): 0,04950 (p = 0,0000) Populaatioiden välille laskettujen parittaisten F ST - ja R ST -arvojen tulokset poikkesivat jossakin määrin toisistaan. Erilaistuminen oli F ST :n perusteella tilastollisesti merkitsevää yhteensä kahdeksassatoista kahdestakymmenestäyhdestä arvosta, ja vastaavasti R ST :n mukaan vain yhdessätoista (Taulukko 8). Taulukko 8. Seitsemän populaation parittaiset F ST - (diagonaalin yllä) ja R ST -arvot (diagonaalin alla) Lounais- Suomi Porkkala Pohjanmaa Karjala Kainuu Itä- Lappi Länsi- Lappi Lounais-Suomi 0,019 0,022 *** 0,002 0,023 *** 0,070 *** 0,080 *** Porkkala -0,01 0,010 0,037 ** 0,057 *** 0,045 *** 0,055 ** Pohjanmaa 0,007 0,010 0,020 *** 0,029 *** 0,044 *** 0,083 *** Karjala 0,013 0,041 0,044 0,015 *** 0,049 *** 0,054 *** Kainuu 0,014 0,053 ** 0,030 * 0,017 0,070 *** 0,073 *** Itä-Lappi 0,065 ** 0,025 0,083 ** 0,085 *** 0,133 *** 0,030 *** Länsi-Lappi 0,079 *** 0,082 * 0,093 *** 0,044 * 0,084 *** 0,044 * = p < 0,05, ** = p < 0,01, *** = p < 0,001 Structure-ohjelman tulosten mukaan tehty jako suurensi AMOVA-tulosten eroavaisuuksia, sillä fiksaatioindeksi F ST :n arvoilla laski (0,037), ja R ST -menetelmällä puolestaan nousi (0,047). Molemmat luvut olivat tilastollisesti merkitseviä (p=0,0000) viitaten populaatioiden erilaistumiseen (Taulukot 9 ja 10) Taulukko 9. FST-arvoihin perustuva populaatioiden välinen ja sisäinen muuntelu (AMOVA), Vaihtelun lähde Vapausasteet Neliösumma Varianssi Muuntelun osuus (%) Populaatioiden 3 35,506 0, ,70 välillä Populaatioiden ,606 3, ,30 sisällä Kokonaisvaihtelu ,112 3,59075 Fiksaatioindeksi (F ST ): 0,03705 (p = 0,0000) 35
38 Taulukko 10. RST-arvoihin perustuva populaatioiden välinen ja sisäinen muuntelu (AMOVA), Vaihtelun lähde Vapausasteet Neliösumma Varianssi Muuntelun osuus (%) Populaatioiden ,946 17, ,70 välillä Populaatioiden , , ,30 sisällä Kokonaisvaihtelu , ,46692 Fiksaatioindeksi (F ST ): 0,04697 (p = 0,0000) Parittaiset F ST -arvot olivat merkitseviä kaikkien Structure-ohjelman tulosten perusteella muodostetun neljän populaation välillä (Taulukko 11). Merkitsevää erilaistumista havaittiin myös R ST -arvoilla mitattuna kaikkien muiden kuin Etelä-Suomen ja Pohjanmaan populaatioiden välillä (Taulukko 11), Taulukko 11. Neljän populaation parittaiset F ST - (diagonaalin yllä) ja R ST -arvot (diagonaalin alla). Etelä-Suomi Pohjanmaa Itä-Suomi Lappi Etelä-Suomi 0,015 *** 0,017 *** 0,058 *** Pohjanmaa 0,008 0,021 ** 0,058 *** Itä-Suomi 0,021 * 0,032 ** 0,053 *** Lappi 0,059 *** 0,076 *** 0,075 *** * = p < 0,05, ** = p < 0,01, *** = p < 0,001. UPGMA-puussa (Kuva 6) Lapin alueen hirvet erottuivat muista selvänä omana haaranaan, joka myös esiintyi kaikissa (saappaanraksiarvo 100 %) uudelleenotantojen perusteella muodostetuissa puissa, jolloin tämän jaon voidaan tulkita olevan tilastollisesti merkitsevä (Nei & Kumar 2000). Muiden haarakohtien saappaanraksiarvot jäivät selvästi pienemmiksi resoluution ollessa riittämätön. 36
39 Kuva 6. UPGMA-puu, jonka haarakohdissa on esitetty kyseisen haaran saappaanraksiarvo. Ylhäällä on kuvattu mitta-asteikko haarakohtien geneettiselle etäisyydelle. Kuvassa 7 on esitetty yksilöiden jakautuminen faktorikorrespondenssianalyysin tuottamien faktoreiden selittämällä kaksiulotteisella tasolla. Alueet eivät muodostaneet selviä ryppäitä. Selvimmin pistejoukosta erottuvat Lapin populaatio, jonka sisällä myös yksilöiden väliset etäisyydet ovat suurimmat. (Kuva 7). Faktoreiden selitysasteet havaittavasta muuntelusta jäivät verrattain mataliksi; ensimmäinen faktori selitti siitä ainoastaan 4,67 % ja toinen vastaavasti 3,82 % Faktori 2 (3,82%) Faktori 1 (4,67%) Etelä-Suomi Itä-Suomi Pohjanmaa Lappi -1.5 Kuva 7. Genetix-ohjelman tuottama pistediagrammi korrespondenssianalyysin tuloksista. Ensimmäinen faktori selittää 4,67 % koko aineiston vaihtelusta ja toinen faktori 3,82 %. 37
40 3.4 Geenivirta Privaattialleelimenetelmällä arvioituna migranttien määrä oli korjauksen jälkeen noin 3,7 kappaletta per sukupolvi, privaattialleelien frekvenssin ollessa 0,043. Jakamalla populaatioiden parittaisista F ST -arvoista lasketut M-arvot (Taulukko 12) kahdella saadaan selville populaatioiden välinen Nm, eli vaikuttavien migranttien määrä.. Tällä menetelmällä laskettuna migraatio vaikuttaisi olevan voimakkainta Karjalan ja Lounais-Suomen populaatioiden välillä (230/2=115 yksilöä), ja vastaavasti heikointa Pohjanmaan ja Länsi-Lapin kesken (5/2=2,5 2 yksilöä). Taulukko 12. Populaatioiden välille laskettu M-arvomatriisi, jossa M=2Nm. Lounais-Suomi Porkkala Pohjanmaa Karjala Kainuu Itä-Lappi Porkkala 25,9 Pohjanmaa 22,0 47,4 Karjala 230,4 13,1 24,5 Kainuu 21,3 8,3 16,9 33,3 Itä-Lappi 6,7 10,6 10,7 9,7 6,7 Länsi-Lappi 5,7 8,6 5,5 8,7 6,3 16,4 Kohdennustesti löysi koko aineistosta yhteensä 17 yksilöä, jotka yksilölliseltä genotyypiltään poikkeavat siitä populaatiosta, johon ne maantieteellisen sijaintinsa puolesta kuuluvat (Taulukko 13). Vastaavasti GeneClass2-ohjelman mukaan mahdollisia ensimmäisen sukupolven immigranttiyksilöitä oli yhteensä kahdeksan (Kuva 8). Taulukko 13. Kohdennustestin luokittelemat lähdepopulaatiot 17 hirviyksilölle. Nro. Sijainti Luokiteltu lähdepopulaatio Kpl 1. Lounais-Suomi, Pohjanmaa 2 2. Lounais-Suomi Porkkala 1 3. Karjala Kainuu 1 4. Karjala Lounais-Suomi 1 5. Karjala Pohjanmaa 3 6. Kainuu Lounais-Suomi 1 7. Kainuu Karjala 2 8. Kainuu Länsi-Lappi 1 9. Kainuu Pohjanmaa Itä-Lappi Lounais-Suomi Itä-Lappi Länsi-Lappi Länsi-Lappi Porkkala 1 38
41 Kuva 8. GeneClass2-ohjelman luokittelemat kahdeksan ensimmäisen sukupolven migranttia. 3.5 Efektiivinen populaatiokoko Kytkentäepätasapainon perusteella lasketut N e -estimaatit ja niiden luottamusvälit on esitetty taulukossa 14. Pienin arvioitu efektiivinen populaatiokoko oli Etelä-Suomen populaatiossa, mutta myös Lapin populaatiolle lasketetut estimaatit jäivät alhaisiksi. Pohjanmaan populaation N e -estimaatit saivat negatiivisia lukemia jokaisella kolmella raja-arvolla mitkä viittaisivat äärettömään populaatiokokoon. Itä-Suomessa negatiivinen tulos saatiin ainoastaan matalimmalla raja-arvolla (0,01) lasketusta estimaatista (Taulukko 14). 39
42 Taulukko 14. Neljän populaation efektiivisen populaatiokoon estimaatit ja niiden 95 %:n luottamusvälit (lv) kolmella eri raja-arvolla (matalin käytetty alleelifrekvenssi) laskettuna. Matalin käytetty alleelifrekvenssi 0,05 0,02 0,01 Etelä-Suomi N e 79,2 53,4 102,8 95 %:n lv. 31,5-29,8-160,9 47,1 - Itä-Suomi N e 378,1 298,6-4507,2 95 %:n lv. 86,2-96,6 147,5 - Pohjanmaa N e -41,8-203,5-203,5 95 %:n lv. 286,1-95,1-95,1 - Lappi N e 77,4 73,4 176,6 95 %:n lv. 35,4-3027,4 38,3-331,8 68,9-3.6 Pullonkaula Geneettisen pullonkaulan merkkien havaitsemiseen käytetyt kaksi menetelmää antoivat erilaiset tulokset hirven historiallisen populaatiokoon muutoksen vaikutuksesta. Bottleneckohjelma ei havainnut selviä merkkejä pullonkaulasta: Wilcoxonin testin tulokset eivät olleet merkitseviä TPM-mutaatiomallilla yhdessäkään tutkitussa seitsemässä populaatiossa, joissa p- arvot vaihtelivat välillä 0,138 (Karjala) ja 0,920 (Länsi-Lappi). Myöskään alleelifrekvenssiluokkien frekvenssit eivät poikenneet normaalista L-jakaumasta (Kuva 9). Porkkalan populaation näytemäärä (8 yksilöä) oli liian pieni luotettavan testituloksen saamiseksi Alleelifrekvenssu Alleelifrekvenssu ,0 2,0 3,0 4,0 5,0 6,0 7,0 8,0 9,0 10,0 0 1,0 2,0 3,0 4,0 5,0 6,0 7,0 8,0 9,0 10,0 Alleelifrekvenssiluokka Alleelifrekvenssiluokka Kuva 9. Karjalan (vasen) ja Länsi-Lapin (oikea) populaatioiden alleelien frekvenssit jaoteltuna alleelifrekvenssiluokkiin. Sen sijaan G-W-indeksin arvot olivat jokaisessa populaatiossa varsin matalia viitaten geneettiseen pullonkaulaan. Keskiarvo kaikista populaatiosta oli vain 0,372 hajonnan ollessa varsin vähäistä (keskihajonta=0,024). Pienin G-W- arvo löytyi Porkkalasta (0,3181) ja korkein vastaavasti Länsi-Lapista (0,386). 40
43 4. POHDINTA 4.1 Aineiston luotettavuus Tutkimukseen pyrittiin valitsemaan maantieteelliset alueet ja yksilöt, jotka antaisivat mahdollisimman kattavan kuvan Suomen hirvikannasta joistakin alueellisista puutteista huolimatta. Pohjois-Suomen osalta Eviran näytevalikoima ei ollut lähellekään yhtä mittava kuin maan eteläosasta, ja mukaan otettiinkin kaikki käytettävissä olevat Lapin ja Kainuun näytteet. Molempien Lapin populaatioiden näytemäärä jäi muita pienemmäksi, minkä lisäksi Lapissa samasta kunnasta oli kerätty useita näytteitä muuta maata useammin. Tämän ei kuitenkaan uskottu haittaavan tulosten luotettavuutta ja vertailukelpoisuutta, koska pohjoisessa kuntia on muutenkin lukumääräisesti vähemmän, ja vastaavasti ne ovat pinta-alaltaan moninkertaisesta etelän pitäjiä suurempia. Tällä tavoin myös Pohjois-Suomen näytteiden välistä maantieteellistä etäisyyttä populaation sisällä saatiin hieman rajoitettua. Näytteiden puutteellisen paikkatiedon vuoksi tehty koordinaattipisteen kohdistaminen näytteenottopaikkakunnan keskukseen etäisyysisolaatioanalyysiä varten on voinut aiheuttaa tuloksiin jonkinasteista harhaa. Eteläisen Suomen näytteiden kohdalla ongelmaa ei ole kuntien pienten pinta-alojen ansioista, mutta Pohjois-Suomen suurissa kunnissa näytteenottopaikan ja kuntakeskuksen välimatka on saattanut olla pitkä. Pieni näytekoko oli ongelma erityisesti Porkkalan populaation kohdalla, mutta nämä yksilöt haluttiin ottaa analyyseihin mukaan alueen eteläisen maantieteellisen sijainnin vuoksi, sekä erityisesti johtuen asiantuntijoiden tekemistä havainnoista, jotka kertovat Porkkalan hirvien olevan heikommassa fyysisessä kunnossa kuin lajikumppaninsa muualla Suomessa (Sauli Laaksonen, suull.ilm.). DNA:n eristäminen verinäytteistä onnistui ongelmitta, mutta sen sijaan PCR-monistus vaatii joidenkin mikrosatelliittilokusten kohdalla olojen optimointia ennen positiivisia tuloksia. Genotyyppien määrityksessä ei havaittu virheitä, ja koottu data (ei esitetty) oli kattava, eikä minkään populaation tai lokuksen kohdalle jäänyt merkittäviä aukkoja. Lokuksissa RT30 ja NVHRT01 esiintyi todennäköisesti yksi (tai useampi) nolla-alleeli, mikä ilmeisesti sai aikaan molemmissa positiivisen F IS -arvon (Taulukko 4), minkä takia näitä lokuksia ei käytetty popu- 41
44 laatioiden sukusiitoskertoimen laskemisessa lainkaan. Muissa analyyseissä nolla-alleelien ei uskottu merkittävästi vaikuttavan tuloksiin. Koko aineiston merkitsevä poikkeaminen HW-tasapainosta on mitä todennäköisimmin tulosta ns. Wahlundin vaikutuksesta (Hedrick 2005); kun populaatiossa on alleelifrekvensseiltään toisistaan poikkeavia alapopulaatioita, tämä johtaa heterotsygotian alimäärään tarkastellessa niitä yhtenä kokonaisuutena, vaikka ne itsenäisinä yksikköinä olisivatkin tasapainossa. Kahden lokusparin välillä havaitun kytkentäepätasapainon ei uskottu vaikuttavan tuloksiin. 4.2 Suomalaisen hirven geneettinen muuntelu Varhaisissa hirvien geneettistä diversiteettiä selvittäneissä tutkimuksissa hirvien todettiin olevan muihin lajeihin verrattuna hyvin vähän muuntelevia (Hundertmark & Boweyer 2004). Se oli myös yksi tärkeimmistä syistä, miksi mielenkiinto lajin perinnöllisen muuntelun tutkimukselle loppui Suomessa 1980-luvulla (Nygren 2009). Harhakäsitys hirven maailmanlaajuisesta monomorfisuudesta on kumottu jo vuosia sitten, ja tämän tutkielman tulokset toivat lisätodisteita eurooppalaisen ja erityisesti suomalaisen hirvipopulaation geneettisestä monimuotoisuudesta Tutkielmassa käytetyt mikrosatelliittilokukset osoittautuivat hyvin muunteleviksi (Taulukko 4) ja seitsemän tutkitun populaation havaittu geneettisen muuntelun määrä oli yllättävän suurta: odotettu heterotsygotia vaihteli populaatioissa välillä 0,682-0,747 ja alleelirunsaus välillä 4,3-5,1, mikä kertoo, etteivät alueelliset erot muuntelun jakautumisessa olleet kovin suuria. Korkeinta muuntelu oli itäisissä populaatioissa Karjalassa ja Kainuussa, jotka ovat suorassa yhteydessä Venäjän populaatioon. Kainuun populaatioista löytyi lisäksi suurin määrä privaattialleeleja, mikä sopii hyvin yhteen sen kanssa, että 1900-luvun alussa tapahtuneen kannan romahduksen seuranneen nousun jälkeen Suomen vanhimmat populaatiot löytyvät juuri Kainuusta ja Lounais-Suomesta. Tässä tutkimuksessa hirvellä havaittu geneettinen muuntelu oli jossain määrin suurempaa kuin aikaisemmissa mikrosatelliitteja hyödyntäneissä töissä. Esimerkiksi Pohjois-Amerikassa alaskanhirven populaatiogenetiikkaa tarkastelleessa tutkimuksessa, jossa muuntelua tarkasteltiin kahdeksan mikrosatelliittilokuksen avulla näytekoon ollessa 141 yksilöä, muuntelu oli pienempää (H e : 0,483-0,616, A: 2,971-4,14) (Schmidt ym. 2009). Charlier ym. (2008) selvit- 42
45 tivät hirven geneettistä muuntelua ja rakennetta neljällä alueella Ruotsissa hyödyntäen yhteensä kuutta mikrosatelliittilokusta. Heidän havaitsemansa geenidiversiteetti vaihteli välillä ,656 ollen suurinta Keski-Ruotsissa Gälveborgin läänin alueen hirvillä. Myös tuoreessa norjalaistutkimuksessa (Solberg ym.2009) korkeimmat hirvillä havaitut H e :n (0,662) ja A:n (4,25) arvot jäivät Suomen tasoa matalammalle. Tämä tutkimus ylitti laajuudellaan ja kattavuudellaan kaikki muut, sillä käytetty näytemäärä oli jopa 616 hirviyksilöä ja muuntelua tarkasteltiin yhteensä 15 lokuksessa (Solberg ym. 2009). Havainto Suomen hirvien Skandinaviaa korkeammasta geneettisestä diversiteetistä on yhteneväinen aikaisemmasta allotsyymitutkimuksesta (Ryman ym. 1980) saatujen tulosten kanssa. Suomen hirvipopulaatio on Ruotsia ja Norjaa paremmin yhteydessä muun Euraasian hirvipopulaation kanssa (Kuva 1), mikä voi osaltaan selittää havaitun ilmiön, koska suurissa yhtenäisissä populaatioissa muuntelu yleensä on suurta (Frankham ym. 2002). Eri hirvieläimiä verrattaessa huomattiin, että suomalaishirvillä havaittu geneettinen muuntelu oli suurempaa kuin esimerkiksi peuran (Rangifer tarandus spp.) eri alalajeilla (H e : 0,441-0,502) (Cronin ym. 2006), mutta kuitenkin pienempää kuin mitä levinneisyydeltään eteläisemmällä isokauriilla (Cervus elaphus) on havaittu eri puolilla Eurooppaa (H e : 0,620-0,880, A: 4,9-8,0)(Zachos ym. 2006). Hirvien geneettinen muuntelu oli samaa tasoa kuin esimerkiksi sudella (Canis lupus) ja ilveksellä (Lynx lynx) Suomessa (Aspi ym. 2006, Pääkkönen 2008). Toiselta tai samaltakaan lajilta eri maista saatujen tulosten suora vertailu on hieman kyseenalaista, koska kussakin tutkimuksessa on käytetty eri määrä joko osittain tai kokonaan eri mikrosatelliitteja. Aikaisempien tutkimustulosten valossa Suomen hirvipopulaation havaittu kohtalaisen korkea geenidiversiteetti oli positiivinen yllätys. Geneettinen muuntelu on tärkeää lajien tulevaisuuden kehityksen kannalta, ja sen merkitys korostuu etenkin käynnissä olevan ilmastonmuutoksen takia. Hirvi on lumeen ja mataliin lämpötiloihin sopeutunut pohjoisten seutujen laji, mutta korkeat lämpötilat sen sijaan voivat olla sille ongelmallisia (Telfer 1984). Vuosittaisen keskilämpötilan nousu ja sekä eteläistä alkuperää olevien loisten ja tautien mahdollinen yleistyminen aiheuttavat hirvikannalle uusia haasteita, joihin vastaamiseen on riittävän korkea geneettisen muuntelun taso erittäin tärkeää. 43
46 Huolen aihetta kuitenkin nostaa sekä Etelä-Suomessa että Lapissa havaitut alhaiset efektiiviset populaatiokoot, jotka voivat altistaa populaatiot geneettisen ajautumisen aiheuttamalle geenidiversiteetin häviämiselle (esim. Allendorf ym. 2008). Etelä-Suomen N e -arvio vaihteli välillä yksilöä, kun esimerkiksi vuosina on Varsinais-Suomen riistahoitopiirin alueella maalaskentatulosten mukaan arvioitu olevan n hirveä ( Lapin riistahoitopiirin alueella on viime vuosina arvioitu olevan noin hirveä (Pusenius 2009), mutta tässä työssä arvioitu efektiivinen populaatiokoko oli vain noin Suomen hirvillä ei havaittu sukusiitoskertoimen perusteella merkkejä sukusiitoksesta, joten se tuskin on vaikuttanut havaitun ilmiön taustalla. Sen sijaan eteläisen Suomen hirvikannan sukupuolijakauma on maan vinoutunein, ja lehmä/sonni-lukusuhde onkin noussut siellä yli kahden naaraan jokaista urosta kohden (Nygren 2009). Populaation sukupuolijakauma on tärkeimpiä efektiiviseen populaatiokokoon vaikuttavia demografisia tekijöitä (Frankham ym. 2002) ja todennäköisimpiä selittäjiä Etelä-Suomen populaation tapauksessa. Arvioiden mukaan efektiivisen populaatiokoon tulisi olla yksilöä populaation evolutiivisen potentiaalin, ja vähintään 50 lisääntymismenestyksen säilyttämiseksi (Frankham ym. 2002). Pohjanmaan ja Itä-Suomen saamat negatiiviset N e -arvot viittaavat äärettömään populaatiokokoon, ja näillä alueilla geenivirta näyttäisikin olevan tässä tutkielmassa laskettujen estimaattien perusteella suurinta. Riski geenidiversiteetin menettämiseen geneettisen ajautumisen takia on siis noissa populaatioissa pieni, mutta varsinkin Etelä-Suomessa tilanne on toinen. 4.3 Hirven geneettinen populaatiorakenne ja geenivirta Suomessa Tämän tutkimuksen tulokset antavat todisteita sille, ettei Suomen hirvikanta ole geneettisestä yhtenäinen vaan, että eri alueiden hirvet ovat jossain määrin erilaistuneita toisistaan. Hirvipopulaation rakenteen tutkimisessa käytettyjen molempien bayesilaisten analyysien mukaan mikrosatelliittiaineistossa on havaittavissa useampi kuin yksi geneettinen ryhmä, mutta menetelmät tuottivat toisistaan hyvin erilaiset tulokset ryhmien määrästä. Lisäksi Structureohjelman antamat tulokset antamat tulokset poikkesivat kahdella eri menetelmällä tulkittaessa (uskottavuusarvo vs. Evannon menetelmä), joten selvien johtopäätösten tekeminen näiden pohjalta on hieman hankalaa. BAPS:in on aikaisemminkin todettu löytävän aineistoista 44
47 enemmän ryhmiä kuin Structure:n (mm. Aspi ym. 2006), mikä johtunee ohjelmien algoritmimenetelmien välisistä eroista. UPGMA-puu (Kuva 6) sekä korrespondenssianalyysin tuottama kuva (Kuva 7) geneettisen muuntelun jakautumisesta osoittivat Lapin ja eteläisemmän Suomen poikkeavan selvästi toisistaan., mikä edellä mainittujen menetelmien lisäksi oli havaittavissa Structure-ohjelman tuloksista (Liite III). Tämä eroavaisuus on todennäköisin selittäjä myös Evannon menetelmän ehdottamalla kahden alapopulaation mallille, mikä muiden tulosten ohessa voisi viitata hierarkkiseen populaatiorakenteeseen, jossa Structure-ohjelman mukaisesti on neljä alapopulaatiota, jotka edelleen jakautuvat kahteen ryhmään. Alapopulaatioiden määrän arvioimista vaikeuttaa kuitenkin tutkimuksessa käytetty otanta, jossa näytteet on poimittu keskitetysti eri alueilta (joiden maantieteelliset etäisyydet olivat tässä työssä kohtuullisen pitkiä), joten ne eivät varsinaisesti edusta mitään tiettyjä tunnettuja alapopulaatioita. Populaatioiden rajat eivät todellisuudessa todennäköisesti ole jyrkät vaan pikemminkin häilyvät. Tarkemman kuvan saamiseksi näytteiden tulisi sijoittua tasaisesti maantieteelliselle alueelle ilman liian pitkiä välimatkoja. Alueellisesti keskitetty näytteenotto kuitenkin sopii hyvin etäisyysisolaatioanalyysille, joka tässä tutkielmassa selittikin aineistoa käytetyistä menetelmistä parhaiten; maantieteellisen etäisyyden kasvaessa Suomen hirvillä myös geneettinen etäisyys populaatioiden välillä kasvoi. Vastaava ilmiö on havaittu hirvellä aikaisemmin myös Ruotsissa (Charlier ym. 2008) ja Alaskassa (Schmidt ym. 2009) sekä Suomessa sudella ja ilveksellä (Aspi ym. 2006, Pääkkönen 2008). Potentiaalisina selittäjinä tälle hirvelläkin havaittavalle geneettiselle rakenteelle pidetään esimerkiksi antropogeenisiä tekijöitä (tiet, kaupungit), alueittain vaihtelevia ravinto-oloja sekä myös intensiivistä metsästystä (Charlier ym. 2008, Schmidt ym. 2009). Ennen kaikkea on muistettava, että vaikka hirvi on potentiaalisesti hyvin liikkuva eläin, on sen paikoitellen todettu olevan käyttäytymiseltään hyvin filopatrinen ja pienellä alueella viihtyvä laji (esim. Cederlund & Sand 1992, Heikkinen 2000), mikä on myös yksi todennäköisimmistä geneettistä populaatiorakennetta selittävistä tekijöistä (Charlier ym. 2008). 45
48 Tässä työssä lasketut estimaatit osoittavat, että eri alueiden hirvipopulaatioiden välillä on geenivirtaa, mutta mitä ilmeisimmin Suomen kokoisessa maassa se ei riitä tasoittamaan havaittuja eroja alleelifrekvensseissä. Sekä M-arvojen että luokittelumenetelmien tulosten perusteella vaikuttaisi siltä, että eteläisissä populaatioissa alueiden välistä geenien vaihtoa on, mutta alueitten välille lasketut parittaiset F ST - ja R ST -arvot sekä AMOVA-tulokset kertovat silti geneettisestä erilaistumisesta. Saadut F ST -arvot olivat kuitenkin selvästi pienempiä kuin esimerkiksi Charlierin ym. (2008) Ruotsin eri alueiden hirvien välillä havaitsemat arvot. Migraatio Lapin ja muun Suomen välillä sen sijaan vaikuttaisi olevan melko heikkoa, minkä lisäksi sekä F ST - että R ST -arvot olivat suurimmillaan verrattaessa Lapin populaatioita muihin. Hirven mtdna:n muuntelua aikaisemmin tarkastelleessa venäläistutkimuksessa (Kholodova ym. 2005, johon viitannut Nygren 2009) on mukana olleista Suomen näytteistä huomattu, että erästä aikaisemmin Pohjois-Ruotsista ja Muurmanskin alueelta määritettyä haplotyyppiä (Hundertmark ym.2002a) esiintyi ainoastaan Lapin hirvissä. Yhteensä Pohjois-Suomessa tavattiin neljää erilaista haplotyyppiä, Väli-Suomessa kolmea ja eteläisimmässä Suomessa sen sijaan ainoastaan yhtä. Tässä tutkielmassa Lapin populaatioista löytyi yhteensä kuusi sellaista alleelia, joita ei havaittu muualla. Selityksenä Suomen pohjois- ja eteläosien hirvien geneettiselle eroavaisuudelle voisi siis olla skandinaavisia alleeleja Lapin hirvipopulaatioihin tuova geenivirta Ruotsista ja Norjasta. Kun pohjoisen ja eteläisen Suomen välinen etäisyys on liian suuri, voi etäisyysisolaatiosta seurata, että migraatio ei riitä sekoittamaan alleeleja, ja geneettinen ajautuminen ja valinta voivat edelleen lisätä eroa. Tosin tämän tutkielman aineistolla ei geenivirtaa Skandinaviasta sen enempää kuin Venäjältäkään Suomeen voida suoraan todistaa, koska se olisi vaatinut sikäläisten näytteiden mukanaoloa analyyseissä. Hirvi on mitä todennäköisimmin saapunut Fennoskandiaan kahdesta suunnasta (Hundertmark ym.2002a ja Kholodova ym. 2005, johon viitannut Nygren 2009), mistä pohjoisen ja etelän populaatioiden geneettiset eroavaisuudet mitä todennäköisimmin juontavat alkuperäiset juurensa. Viimeisimmän jääkauden jälkeen Ruotsin ja Manner-Euroopan yhdisti maasilta, jota pitkin eri eliölajit ovat päässeet leviämään pohjoiseen mannerjään vetäytyessä. Samaan aikaan nämä samat lajit ovat levinneet Fennoskandiaan idästä Karjalan kautta eri lähdepopulaatiosta. Tämän on uskottu olevan syynä esimerkiksi karhulla (Ursus arctos) havaittavalle geneettiselle eroavaisuudelle pohjoisen ja etelän populaatioiden välillä Skandinaviassa (Taberlet ym. 46
49 1998), minkä lisäksi Solberg ym. (2009) epäilivät saman ilmiön olevan taustalla Norjan hirvipopulaatiossa havaitsemassaan geneettisessä rakenteessa. Viimeisillä jääkausilla tiedetään olleen muutenkin todella voimakas vaikutus pohjoisten lajien geneettiseen rakenteeseen. Useissa tutkimuksissa on havaittu, että aiemmin mannerjäätiköiden peittämien alueiden eliöiden geneettisen muuntelun taso on vähäisempää kuin eteläisempien lajien (Hewitt 2004). Näiden pohjoisten lajien populaatiorakenteeseen vaikuttavia tekijöitä ovat habitaattien leveysasteen siirtyminen, maantieteelliset esteet, refugioiden määrä ja sijainti sekä eliön dispersaalikyky. Hirven fylogeografiaa lajin koko levinneisyysalueelta kerätyn mtdna:n muuntelun avulla selvittäneissä tutkimuksissa on pyritty selittämään hirvillä havaitun geneettistä muuntelua ja rakennetta sen kolonisaatiohistorialla (esim. Hundertmark ym.2002a). Boreaalisen vyöhykkeen siirtyminen pleistoseenikaudella etelään ilmaston kylmenemisen myötä on nykykäsityksen mukaan muokannut hirven levinneisyyttä rajusti (Hundertmark ym.2002a). Hirven alkukodin, Aasian, itä-länsisuuntaiset vuoristot ovat estäneet habitaattien siirtymistä ja ovatkin mitä todennäköisimmin pienentäneet niiden kokoa, toimien siis myös esteenä hirven liikkumiselle. Lämpimämpien ajanjaksojen aikana laji on taas voinut levitä pohjoiseen, jolloin populaatiotkin ovat voineet laajeta. Hundertmark ym. (2002a) havaitsivat analysoimansa geneettisen aineiston perusteella historiallisen hirvikannan kokeneen kaksi ajallisesti erillistä ekspansiota, jotka ajoittuvat arvioilta ja vuoden taakse. Tutkijat huomasivat näiden ekspansioiden korreloivan jääkauden kylmimpiä aikoja seuranneiden lämpimämpien ajanjaksojen kanssa. Tästä johtuen historiallisten, täyskäännöksiä tehneiden suurten ilmastonmuutosten on todettu hyvin todennäköisesti vaikuttaneen olennaisesti hirven evoluutioon ja maantieteelliseen levinneisyyteen jättäen samalla jälkensä modernin hirven perimään (Hundertmark & Bowyer 2004). 4.4 Pullonkaula Analysoimalla mtdna-aineistoa Hundertmark ym. (2002a) löysivät merkkejä siitä, että eurooppalainen hirvi on käynyt geneettisen pullonkaulan läpi. Vaikka aineisto Euroopan osalta onkin pieni, ovat tulokset yhteneväiset niiden tietojen kanssa, joita hirven historiasta mantereelta löytyy (Nygren 1987, 2009, Schmölcke & Zachos 2005). Tuoreissa ruotsalais- ja norja- 47
50 laistutkimuksissa ei kuitenkaan pystytty havaitsemaan mikrosatelliittiaineiston perusteella viitteitä pullonkaulasta Skandinaviassa, jossa hirvikannan historia on hyvin samankaltainen kuin Suomessa (Charlier ym. 2008, Solberg ym. 2009). Tässä tutkielmassa kahdella eri menetelmällä saadut tulokset olivat ristiriidassa keskenään. Bottleneck-ohjelma, jota myös Charlier ym. käyttivät (2008), ei havainnut Suomen hirvipopulaatiossa merkkejä geneettisestä pullonkaulasta. Ohjelman suorittaman testin heikkona puolena kuitenkin on, että aikajakso, jonka aikana pullonkaulan merkit ovat sen havaittavissa, on rajallinen. Cornuet ja Luikart (1996) arvioivat, että pullonkaula, jossa N e = 50 olisi havaittavissa heterotsygotian ylimäärä-menetelmällä sukupolvea (0,25 2,5*2Ne) siitä ajankohdasta kun populaatio alkoi pienetä. Garza-Williamson-menetelmällä pullonkaulat ovat havaittavissa vielä usean sadan sukupolven jälkeen (Garza & Williamson 2001), ja tämän menetelmän tuloksien perusteella Suomen hirvet näyttäisivät käyneet pullonkaulan läpi. Havaitut arvot olivat hyvin paljon pienempiä (0,32-0,39) kuin raja-arvo 0,7, jota pienempiä lukuja pidetään pullonkaulaan viittaavana (Garza & Williamson 2001). Eri menetelmiä vertailtaessa on havaittu, että G-W-indeksi ja heterotsygotian ylimäärään perustuva testi (kuten Bottleneck-ohjelman suorittama) eroavat kyvyssään havaita populaation pullonkauloja. Erot riippuvat tutkittavan populaation koosta sekä pullonkaulan kestosta ja vakavuudesta (Williamson-Natesan 2005). Tilanteessa, jossa populaatiokoko on alkujaan ollut suuri, pullonkaula on kestänyt useita sukupolvia tai populaatio on demografisesti toipunut, tulisi G-W indeksin toimia paremmin. Vastaavasti heterotsygotian ylimäärää tarkastelevat testit soveltuvat paremmin, jos pullonkaula on tapahtunut verrattain lyhyen ajan sisällä eikä ole ollut vakava ja populaatiokoko on ollut alun perin pieni (Williamson-Natesan 2005). Valitettavasti tarkkoja ja luotettavia arvioita siitä, kuinka paljon hirviä on Suomessa ollut ennen kannan romahtamista 1800-luvun puolessa välissä ja toistamiseen 1900-luvun alussa, ei ole olemassa, minkä lisäksi vanhoihin, aikalaisten tekemiin systemaattisiin laskentatapoihin perustumattomiin arvioihin on hyvin helppo suhtautua kriittisesti. Se kuitenkin tiedetään, että populaatiokoko on tippunut hyvin alas useamman kerran suhteellisen lyhyellä aikavälillä. Vastaavasti 1900-luvun lopulle tultaessa kanta toipui ja hirvien määrä kasvoi sekä Suomessa että naapurimaissa moninkertaisesti (esim. Nygren 2009). Nämä tekijät voivat selittää sen, 48
51 miksi Bottleneck-ohjelma ei kyennyt havaitsemaan merkkejä pullonkaulasta kun taas G-Windeksi siihen pystyi. Suomen hirvikannan geneettisessä koostumuksessa on siis tämän tutkielman tulosten valossa havaittavissa merkkejä aikaisemmasta populaation pullonkaulasta. Kuitenkin tutkimuksessa havaitusta kohtuullisen korkeasta geneettisen muuntelun määrästä ja sen suhteellisen tasaisesta maantieteellisestä jakautumisesta päätellen mahdollinen pullonkaula ei joko ole ollut vakava tai migraatio kantaa reunustavilta alueilta on ehtinyt palauttaa menetettyä muuntelua. Vaikka pieni populaatiokoko onkin joskus voinut altistaa hirvipopulaation sukusiitokselle, niin tämänhetkisessä kannassa ei sitä sukusiitoskertoimen perusteella ole havaittavissa. 4.5 Metsästyksen geneettiset vaikutukset Suomen hirvikanta on historiansa aikana romahtanut useita kertoja ja on ollut jopa vaarassa hävitä maasta kokonaan ihmisen toimien takia (Nygren 2009). Aallonpohjista on kuitenkin toivuttu, ja 2000-luvulla Suomen hirvipopulaatio on ollut määrällisesti suurempi kuin koskaan aikaisemmin. Kestämättömällä pohjalla oleva metsästys voi kuitenkin aiheuttaa suoran demografisen kannanromahduksen ohella vähemmän ilmeisiä uhkia: ihmisen suorittamalla verotuksella voi nimittäin olla riistaeläinkantoihin sekä ekologisia, geneettisiä että evolutiivisia vaikutuksia, joista aikaisemmin ei ole osattu tai haluttu olla kiinnostuneita saati huolestuneita (Milner ym. 2007, Allendorf ym. 2008, Coltman 2008, Nygren 2009). Suurin osa niistä on seuraamusta tiettyyn sukupuoleen, ikäluokkaan tai fenotyyppiin kohdistuvasta valikoivasta verotuksesta, jonka perinnöllisistä vaikutuksista toimivat esimerkkeinä populaation alarakenteen muuttuminen, geneettisen muuntelun menettäminen sekä selektiiviset geneettiset muutokset. Aikaisempia todisteita metsästyksen mahdollisista geneettisistä vaikutuksista Suomen hirvikantaan on saatu tutkimalla hirven sarvia, joita olemassa kahta tyyppiä, hankosarvia ja lapiosarvia (Nygren ym. 2007). Näitä säätelevät semidominantisti periytyvät ja penetranssiltaan uroksen iästä riippuvan geenin kaksi alleelia: lapiosarvialleeli ja hankosarvialleeli (Nygren 2009). Sarvien eri ominaisuudet vaihtelevat sekä ajallisesti että alueellisesti; pohjoista kohti mentäessä sarvien leveys, piikkimäärä sekä urosten paino lisääntyvät ja lapiosarvisten osuus kasvaa. Lisäksi lapiosarvisten osuuden on todettu pienentyneen tilastollisesti merkitsevästi kolmessa neljästä suuralueesta (Nygren ym. 2007), mikä siis on seurausta lapiosarvialleelin 49
52 suhteellisesta vähenemisestä. Syitä hankosarvien lisääntymiselle ei tunneta, mutta tällainen havainto suhteellisen lyhyellä aikavälillä tapahtuneesta muutoksesta geenifrekvensseissä voimakkaasti hyödynnetyssä riistaeläinpopulaatiossa on varsin merkittävä. Mahdollisina selityksinä havaitulle ilmiölle on esitetty 1) Etelä-Suomen metsien rakenteen muutoksesta johtuvaa luonnonvalintaa, 2) lapiosarvisiin yksilöihin kohdistuvaa voimakkaampaa saalisvalintaa ja 3) metsästyksen takia vinoutunutta sukupuolijakaumaa, joka lisääntymiskäyttäytymiseen vaikuttamalla ja efektiivistä populaatiokokoa pienentämällä on voimistanut sekä luonnonvalinnan että saalisvalinnan vaikutuksia (Nygren 2009). Tässä tutkielmassa joillakin alueilla todetun matalan efektiivisen populaatiokoon aiheuttajaa ei ole mahdollista suoraan osoittaa, mutta sukupuolijakaumaa vääristävän uroksiin painottuvan valikoivan metsästyksen on kuitenkin todettu vaikuttavan voimakkaasti sekä hirven efektiiviseen populaatiokokoon että sukupolven pituuteen (Ryman ym. 1981, Allendorf ym. 2008) Rymanin ym. (1981) laskeman simulaatiomallin mukaan urosten valikoiva metsästys voi laskea populaation efektiivisen koon jopa alle 20 prosenttiin sen todellisesta koosta. Lisäksi on olemassa viittauksia siitä, että kaadettaessa suhteessa paljon vanhoja yksilöitä geneettisen ajautumisen riski kasvaa enemmän kuin kaadettaessa vastaava määrä nuoria yksilöitä. Tämä johtuu siitä, että sukupolven pituus laskee merkittävästi, jos metsästys painottuu vanhoihin ikäryhmiin. Tästä syystä verotuksen mahdollisia vaikutuksia arvioidessa tulisikin sukupolven pituus ottaa efektiivisen populaatiokoon rinnalla huomioon yhtä tärkeänä tekijänä (Allendorf ym. 2008). Käytännössä geneettisen muuntelun säilyttämisen ja haitallisten muutosten välttämisen kannalta paras kannanhoidollinen menettelytapa olisi siis nuorten yksilöiden voimakkaampi verotus suhteessa vanhempiin ikäluokkiin. Vastaavasti uroksia ja naaraita tulisi kaataa tasapuolisesti. Nuorten, tuottamattomien ikäluokkien verottaminen on nykyisin suomalaisen kannansuunnittelun kenties olennaisin käytäntö, mutta valitettavasti eri sukupuolia metsästetään hyvin epätasaisesti (Nygren 2009). Vaikka tämän tutkielman tulosten perusteella nähtiinkin, että suomalaisen hirven geneettinen muuntelun määrä oli verrattain korkea, tulisi tämän hetkinen vinoutunut sukupuolijakauma saada tasoittumaan, jotta mahdollinen riski tuon muuntelun menettämiselle saataisiin pienenemään Sekä alapopulaatioiden sisäisen että niiden välisen geneettisen muuntelun säilyttäminen on tärkeää, jotta voidaan taata ja turvata sekä lajin pitkän aikavälin elinvoimaisuus että evoluutio 50
53 (Allendorf ym. 2008, Charlier ym. 2008). Populaatioiden välistä muuntelua tasoittaa geenivirta, joka vastaavasti ylläpitää populaatioiden sisäistä muuntelua. Hirvillä urokset dispersoivat naaraita kauemmas (esim. Cederlund & Sand 1992), mutta metsästyksestä johtuen juuri niiden kuolleisuus on Suomessa naaraita suurempaa (Nygren 2009). Tämän tutkimuksen tulosten mukaan Suomen hirvipopulaatiossa on havaittavissa geneettistä erilaistumista ja jakautuneisuutta eri alueitten välillä ja kannan harvetessa on mahdollista, että eri alueiden hirvipopulaatioiden väliset geneettiset erot kasvavat varsinkin, jos migraatio heikkenee dispersoivien nuorten urosten päätyessä muita todennäköisemmin saaliiksi luvulla Suomen hirvikannan perimän monimuotoisuuden säilyttämisestä ei ole juurikaan kannettu huolta kannan säätelystä päättävissä elimissä. Uhka geneettisen muuntelun häviämiselle onkin nyt suurempi kuin koskaan: aikuiskannan sukupuolijakauman raja-arvot on poistettu ja uroksia on kannassa nyt ennätyksellisen paljon. Kuluneen vuosikymmenen loppupuoliskolla on Suomen hirvikantaa leikattu rajusti kohti entistä pienempiä tiheystavoitteita: 2-4 yksilöä 1000 hehtaaria kohti. Lisäksi vuotuinen poisto on ollut valikoivaa parhaassa siitosiässä olevien urosten päätyessä hyvin usein saaliiksi (Nygren 2009). Toisin tulisi kuitenkin olla, jotta harjoitettava säätely olisi kestävää ja Suomen hirvikanta säilyisi koko maan laajuudelta elinvoimaisena ja valmiina tulevaisuuden haasteille. Kannanhoidon menetelmiä tulisi kehittää soveltamalla olennaisimpia genetiikan periaatteita, minkä lisäksi tulisi suorittaa geneettistä monitorointia hirvikannan perimässä tapahtuvien muutosten seuraamiseksi sekä mahdollisten riskien arvioimiseksi ja minimoimiseksi. 4.6 Jatkotutkimukset Tarkasteltujen näytteiden määrän ja alueellisen jakauman perusteella tätä tutkielmaa voidaan pitää ainoastaan pilottitutkimuksena Suomen hirvien populaatiogenetiikasta; näytteitä tarvitaan enemmän ja kattavammin koko maan laajuudelta selvittämään tarkemmin hirven geneettistä populaatiorakennetta. Ennen kaikkea tutkimusta tarvitaan kaikkien siihen mahdollisesti vaikuttavien tekijöiden tunnistamiseksi ja niiden merkityksen arvioimiseksi. Lisäksi Suomen, Skandinavian sekä myös Venäjän hirvikantojen väliset suhteet kaipaavat lisävaloa. Analysoimalla näytteitä kustakin maasta yhdessä voitaisiin tehdä arvioita maiden välisestä migraatiosta ja geneettisen erilaistumisen/samankaltaisuuden määrästä. 51
54 Eristämällä DNA:ta historiallisista ja jopa muinaisista hirven jäänteistä, kuten museoaineistosta, olisi mahdollista tarkastella geneettisen muuntelun ajallista vaihtelua ja kehittymistä sekä selvittää myös pullokaulan vaikutuksia entistä paremmin. Neutraalin muuntelun lisäksi tarvittaisiin tietoa myös adaptiivisesta muuntelusta sekä valikoivan verotuksen roolista siihen vaikuttavana valintatekijänä. Metsästyksestä jo aiheutuneiden ja mahdollisten tulevien geneettisten ja evolutiivisten muutosten tarkempi arvioiminen onkin yksi tulevaisuuden tärkeimmistä haasteista, ei ainoastaan hirven, vaan myös muiden riistaeläinten kohdalla 52
55 5. YHTEENVETO Hirvi (Alces alces) on Suomen tärkein riistaeläinlaji, ja ihminen on metsästänyt tätä suurnisäkästä jo tuhansien vuosien ajan ja 1900-luvuilla hirvikannat romahtivat koko Fennoskandiassa useita kertoja hyvin alas liiallisen verotuksen takia, mutta 2000-luvulle tultaessa hirvien määrät nousivat ennätyksellisen korkeiksi metsän rakenteessa ja kannansäätelyssä tapahtuneiden muutosten ansiosta. Nykyisin Suomen hirvikantaa säädellään voimakkaasti ajallisesti vaihtelevien määrällisten ja laadullisten tavoitteiden mukaisesti, mutta populaation geneettistä monimuotoisuutta ei nykyisessä verotussuunnittelussa oteta huomioon lainkaan. Tässä pro gradu-tutkielmassa tarkasteltiin Suomen hirvipopulaation geneettistä muuntelua ja populaatiorakennetta yhteensä kymmenen mikrosatelliittilokuksen avulla. Arvioita tehtiin myös kannan romahtamisesta seuranneesta mahdollisesta geneettisestä pullonkaulasta, efektiivisestä populaatiokoosta sekä eri alueiden välisestä geenivirrasta Kerätty DNA-aineisto koostui seitsemältä maantieteelliseltä alueelta kerätyistä yhteensä 130 hirven verinäytteestä. Havaittu geneettinen diversiteetti oli Suomen hirvillä oli kohtuullisen korkeaa (H e : 0,682-0,747 ja A: 4,3-5,1) ja alueellisesti tasaisesti jakautunutta. Saadut arvot olivat lisäksi hieman suurempia kuin aikaisemmissa ulkomaisissa tutkimuksissa on havaittu. Suomen hirvipopulaatio näyttäisi tämän tutkielman tulosten valossa käyneen geneettisen pullonkaulan läpi, mutta se ei joko ole ollut vakava tai migraatio on ehtinyt palauttaa menetettyä muuntelua. Sukusiitoksesta ei hirvillä havaittu merkkejä. Saatujen tulosten perusteella Suomen hirvikanta ei ole geneettisesti täysin yhtenäinen, mutta alapopulaatioiden määrää arvioivat menetelmät antoivat toisistaan poikkeavia tuloksia. Parhaiten aineistoa selitti etäisyysisolaatiomalli, jonka mukaan maantieteellisen etäisyyden kasvaessa myös hirvien geneettinen etäisyys kasvaa. Ryhmittelyanalyysit erottivat Lapin populaation erilliseksi ryhmäkseen, minkä lisäksi lasketut FST- ja RST-arvot olivat suurimmillaan verrattaessa Lapin hirviä eteläisimpien alueiden hirviin. Myös geenivirta noiden ryhmien välillä vaikutti olevan melko heikkoa. Arvioitu efektiivinen populaatiokoko oli pieni sekä eteläisessä Suomessa että Lapissa, minkä epäiltiin olevan seurasta valikoivasta verotuksesta johtuvasta vinoutuneesta sukupuolija- 53
56 kaumasta hirvillä. Toimenpiteet ovat tulevaisuudessa tarpeen efektiivisen populaatiokoon nostamiseksi, jotta riski geneettisen muuntelun vähenemiseksi saataisiin minimoitua. 6. KIITOKSET Tahdon esittää erittäin lämpimät kiitokseni tämän pro gradu-tutkielman ohjaamisesta dosentti Laura Kvistille sekä dosentti Jouni Aspille, jolle myös kuuluu kunnia alkuperäisideasta. Lauraa tahdon kiittää rennosta asenteesta ja kaikesta siitä avusta ja opastuksesta, jota olen saanut työn jokaisessa eri vaiheissa. Yhteistyöstä ja verinäytteiden toimittamisesta kiitokset Sauli Laaksoselle Eviraan. Työn taloudellisten kulujen kattamisesta kuuluu kiitos Jouni Aspille, ja henkilökohtaisista stipendeistä Suomen Riistanhoitosäätiölle, Suomen Biologian Seura Vanamolle ja Oulun luonnonystäville. Kiitän myös ystäviäni ja erityisesti perhettäni saamastani loppumattomasta tuesta ja kannustuksesta. Isälle ja muille Päivänniemen sankareille kiitos siitä, että tutustutitte minut jo pikkupoikana hirvien maailmaan. Lopuksi haluan omistaa tämän työn vastikään edesmenneen isoisäni Mauri Kankaan muistolle. Kiitos antamastasi esikuvasta, Ukko! 54
57 7. LÄHTEET Aarnio, J. & Härkönen, S. (2007) Hirvestä hyötyjä ja kustannuksia. Metsätieteen aikakausikirja. 2: Allendorf, F.W., England, P.R., Luikart, G., Ritchie, P.A. & Ryman, N. (2008) Genetic Effects of Harvest on Wild Animal Populations. Trends in Ecology and Evolution. 23: Aspi, J., Ruokonen, M., Kojola, I. & Vila, C. (2006) Genetic Diversity, Population Structure, Effective Population Size and Demographic History of the Finnish Wolf Population. Molecular Ecology. 15: Barton, N.H. & Slatkin, M (1986) A Quasi-Equilibrium Theory of the Distribution of Rare Alleles in a Subdivided Population. Heredity. 56: Beebee, T. & Rowe, G. (2004) An Introduction to Molecular Ecology. Oxford Univesity Press. 346 s. Belkhir, K., Borsa, P., Chikhi, L., Raufaste, N., Catch, F. (2004) GENETIX , Software under Windows for the genetics of the populations. Laboratory Genome, Populations, Interactions, CNRS UMR 5000, University of Montpellier II, Montpellier, France. Broders, H.G., Mahoney, S.P., Montevecchi, W.A. & Davidson, W.S. (1999) Population Genetic Structure and the Effect of Founder Events on the Genetic Variability of Moose, Alces alces in Canada. Molecular Ecology. 8: Bubenik, A.B. (1987) Behaviour of Moose (Alces alces SSP) of North America. Teoksessa Göransson, G. & Lavsund, S. (toim.) Swedish Wildlife Supplement, Proceedings Second International Moose Symposium. Svenska Jägareförbundet. 1: Cederlund, G.N. & Sand, H.K.G. (1992) Dispersal of Subadult Moose (Alces alces) in a Nonmigratory Population. Canadian Journal of Zoology. 70:
58 Chapuis, M.-P. & Estoup, A. (2007) Microsatellite Null Alleles and Estimation of Population Differentiation. Molecular Biology and Evolution. 24: Charlier, J., Laikre, L. & Ryman, N. (2008) Genetic Structure and Evidence of a Local Bottleneck in Moose in Sweden. Journal of Wildlife Management. 72: Chesser, R.K, Reuterwall, C. & Ryman, N. (1982) Genetic Differentiation of Scandinavian Moose Alces Alces Over Short Geographical Distances. Oikos. 39: Coltman, D.W. (2008) Molecular Ecological Approaches to Studying the Evolutionary Impact of Selective Harvesting in Wildlife. Molecular Ecology. 17: Corander, J., Waldmann, P. & Sillanpää, M.J. (2003) Bayesian Analysis of Genetic Differentiation Between Populations. Genetics. 163: Cornuet, J.-M. & Luikart, G. (1996) Description and Power Analysis of Two Tests for Detecting Recent Population Bottlenecks From Allele Frequency Data. Genetics. 144: Cronin, M.A., MacNeil, M.D. & Patton, J.C. (2006) Mitochondrial DNA and Microsatellite DNA Variation in Domestic Reindeer (Rangifer tarandus tarandus) and Relationships with Wild Caribou (Rangifer tarandus granti, Rangifer tarandus groenlandicus, and Rangifer tarandus caribou). Journal of Heredity. 97: Dakin, E.E. & Avise, J.C. (2004) Microsatellite Null Alleles in Parentage Analysis. Heredity 93: Dieringer, D. & Schlötterer, C. (2003) Microsatellite Analyser (MSA): a Platform Independent Analysis Tool for Large Microsatellite Data Sets. Molecular Ecology Notes. 3: Ellegren, H. (2004) Microsatellites: Simple Sequences with Complex Evolution. Nature Reviews. Genetics. 5:
59 Estoup, A., Jarne, P. & Cornuet, J.-M. (2002) Homoplasy and Mutation Model at Microsatellite Loci and Their Consequences for Population Genetics Analysis. Molecular Ecology. 11: Evanno, G.,Regnaut, S. & Goudet, J. (2005) Detecting the Number of Clusters of Individuals Using the Software STRUCTURE : A Simulation Study. Molecular Ecology. 14: Excoffier, L., Laval, G. & Schneider, S. (2005) Arlequin ver. 3.0: An Integrated Software Package for Population Genetics Data Analysis. Evolutionary Bioinformatics Online. 1: Excoffier, L., Smouse, P.E. & Quattro, J.M. (1992) Analysis of Molecular Variance Inferred From Metric Distances among DNA Haplotypes: Application to Human Mitochondrial DNA Restriction Data. Genetics. 131: Frankham, R., Ballou, J.D. & Briscoe, D.A. (2002) Introduction to Conservation Genetics. Cambridge University Press. 617s. Futuyma, D.F. (1998) Evolutionary Biology. Sinauer Associates, Inc. 763 s. Garza, J.C. & Williamson, E.G. (2001) Detection of Reduction in Population Size Using Data from Microsatellite Loci. Molecular Ecology 10: Geist, V. (1987) On the Evolution and Adaptations of Alces. Teoksessa Göransson, G. & Lavsund, S. (toim.) Swedish Wildlife Supplement, Proceedings Second International Moose Symposium. Svenska Jägareförbundet. 1: Goudet, J. (2001) FSTAT, a Program to Estimate and Test Gene Diversities and Fixation Indices (version 2.9.3). Hardy, O.J. & Vekemans, X. (2002) SPAGeDi: a Versatile Computer Program to Analyze Spatial Genetic Structure at Individual or Population Levels. Molecular Ecology Notes. 2:
60 Hartl, D.L. & Clark, A.G. (1997) Principles of Population Genetics. Sinauer Associates, Inc. 542 s. Hedrick, P.W. (2005) Genetics of Population. Jones and Bartlett Publishers. 737 s. Heikkinen, S. (2000) Hirven vuosi. Suomen Riista. 46: Hewitt, G.M. (2004) Genetic Consequences of Climatic Oscillations in the Quaternary. Philosofical Transactions of Royal Society B. 359: Hoffman, J.D. (2006) Long-Distance Dispersal and Population Trends of Moose in the Central United States. Alces. 42: Howard, W. E. (1960) Innate and Environmental Dispersal of Individual Vertebrates. The American Midland Naturalist. 63: Hundertmark, K.J. & Bowyer R.T. (2004) Genetics, Evolution and Phylogeography of Moose. Alces. 40: Hundertmark, K.J., Shields, G.F., Bowyer, R.T. & Schwartz, C.C. (2002b) Genetic Relationships Deduced from Cytochrome-b Sequences among Moose. Alces. 38: Hundertmark, K.J., Shields, G.F., Udina, I.G., Bowyer, R.T., Danilkin, A.A. & Schwartz, C.C. (2002a) Mitochondrial Phylogeography of Moose (Alces alces): Late Pleistocene Divergence and Population Expansion. Molecular Phylogenetics & Evolution. 22: Jarne, J. & Lagoda, P.J.L. (1996) Microsatellites, from Molecules to Populations and Back. Trends in Ecology & Evolution. 11: Jia, J. (1996) Moose Living Strategies: Biological Features in Manchuria, and Forage Selection in Fennoscandia. Akateeminen väitöskirja. Maatalous-metsätieteellinen tiedekunta. Helsingin yliopisto. 58
61 Johnson, M.L & Gaines, M.S. (1990) Evolution of Dispersal: Theoretical Models and Empirical Tests Using Birds and Mammals. Annual Review of Ecology and Systematics, 21: Kholodova, M. V., Davydov, A. V., Meshsherskii, I. G., Piskunov, O. D. & Rozhkov, Ju. I. (2005) Izuchenije molekuljarno-geneticheskogo raznoobrazija losja (Alces alces L.) centralnoi I severozapadnoi chasti Rossii: analiz mtdna. Vestnik Okhotovedenija. 2: Koivisto, I. (1966) Yhteenveto hirvilaskennan tuloksista vuonna Suomen Riista. 19: Li, Y.C., Korol, A.B., Fahima, T., Beiles, A., Nevo, E. (2002) Microsatellites: Genomic Distribution, Putative Functions and Mutational Mechanisms: a Review. 11: Litt, M & Luty, J.A. (1989) A Hypervariable Microsatellite Revealed by In Vitro Amplification of a Dinucleotide Repeat within the Cardiac Muscle Actin Gene. American Journal of Human Genetics. 44: Loiselle, B.A., Sork, V.L., Nason, J. & Graham, C. (1995) Spatial Genetic Structure of a Tropical Understory Shrub, Psychotria officinalis (Rubiaceae). American Journal of Botany. 82: Luikart, G., Allendorf, F.W., Sherwin, B. & Cornuet, J.-M. (1998) Distortion of Allele Frequency Distributions Provides a Test for Recent Population Bottlenecks. Journal of Heredity. 12: Luikart, G. & Cornuet, J.-M. (1998) Empirical Evaluation of a Test For Identifying Recently Bottlenecked Populations from Allele Frequence Data. Conservation Biology. 12: Mikko, S. & Andersson, L. (1995) Low Major Histocompatibility Complex Class II Diversity in European and North American Moose. Proceedings of the National Academy of Sciences. 92:
62 Miller, M.P. (1997) Tool for Population Genetic Analyses (TFPGA). A Windows Program for the Analysis of Allozyme and Molecular Population Genetic Data. Department of Biological Sciences. Nothern Arizona University. Milner, J. M., Nilsen, E. B. & Andreassen, H. P. (2007) Demographic Side Effects of Selective Hunting in Ungulates and Carnivores. Conservation Biology. 21: Moore, S.S., Byrne, K., Berger, K.T., Barendse, W., McCarthy, F., Womack, J.E. & Hetzel, D.J.S (1994) Characterization of 65 Bovine Microsatellites. Mammalian Genome. 5: Nei, M. (1978) Estimation of Average Heterotsygosity and Genetic Distance from a Small Number of Individuals. Genetics. 89: Nei, M. & Kumar, S. (2000) Molecular Evolution and Phylogenetics. Oxford University Press. 333 s. Neigel, J.E. (1997) A Comparison of Alternative Strategies for Estimating Gene Flow from Genetic Markers. Annual Review of Ecology and Systematics. 28: Nikula, A., Heikkinen, S. & Helle, E. (2004) Habitat Selection of Adult Moose Alces alces at Two Spatial Scales in Central Finland. Wildlife Biology. 10: Nikula, A., Härkönen, S., Leskine, P. & Kurttila, M. (2007) Hirvikannan säätelyn tasot ja päätöksenteko. Metsätieteen aikakausikirja. 2: Nygren, K. & Wallen, M.-L. (2001) Hirvi-tietosanakirja. Riistan- ja kalantutkimuslaitos. 162 s. Nygren, T. (1987) The History of Moose in Finland Teoksessa Göransson, G. & Lavsund, S. (toim.) Swedish Wildlife Supplement, Proceedings Second International Moose Symposium. Svenska Jägareförbundet. 1: Nygren, T. (2009) Suomen hirvikannan säätely biologiaa ja luonnonvarapolitiikkaa. Akateeminen väitöskirja. Biotieteiden tiedekunta. Joensuun yliopisto. 117 s. 60
63 Nygren, T., Pusenius, J., Tiilikainen, R. & Korpelainen, J. (2007) Moose Antler Type Polymorphism: Age and Weight Dependent Phenotypes and Phenotype Frequencies in Space and Time. Annales Zoologici Fennici. 44: Paetkau, D., Calvert, W., Stirling, I. & Strobeck, C. (1995) Microsatellite Analysis of Population Structure in Canadian Polar Bears. Molecular Ecology. 4: Paetkau, D., Slade, R., Burden, M., & Estoup, A. (2004) Genetic Assignment Methods for the Direct, Real-time Estimation of Migration Rate: a Simulation-Based Exploration of Accuracy and Power. Molecular Ecology. 13: Paetkau, D., Waits, L.P., Clarkson, P.L., Craighead, L. and Strobeck, C. (1997) An Empirical Evaluation of Genetic Distance Statistics Using Microsatellite Data from Bear (Ursidae) Populations. Genetics. 147: Piry, S., Alapetite, A., Cornuet, J.-M., Paetkau, D., Baudouin, L. & Estoup, A. (2004) GENECLASS2: A Software for Genetic Assignment and First-Generation Migrant Detection. Journal of Heredity. 95: Piry, S., Luikart, G. & Cornuet, J.-M. (1999) BOTTLENECK: A Computer Program for Detecting Recent Reductions in the Effective Population Size Using Allele Frequency Data. Journal of Heredity. 90: Pritchard, J.K, Stephens, M. & Donnelly, P. (2000) Inference of Population Structure Using Multilocus Genotype Data. Genetics. 155: Pulliainen, E. (1987) The Moose in the Early History of Man in Finland a Review. Teoksessa Göransson, G. & Lavsund, S. (toim.) Swedish Wildlife Supplement, Proceedings Second International Moose Symposium. Svenska Jägareförbundet. 1: Pusenius, J.,Tykkyläinen, R., Wallén, M., Karhapää, A., Kohvakka, J. & Murto, K. (2009) Teoksessa Wikman, M. (toim.) Riistakannat 2009 Riistaseurantojen tulokset. 18:
64 Pusey, A. & Wolf, M. (1996) Inbreeding Avoidance in Animals. Trends in Ecology and Evolution. 11: Pääkkönen, S. (2008) Fennoskandian ilveksen (Lynx lynx) geneettinen muuntelu ja populaatiorakenne. Pro gradu-tutkielma. Oulun yliopisto, biologian laitos. 99 s. Rannala, B. & Mountain, J.L. (1997) Detecting Immigration by Using Multilocus Genotypes. Proceedings of the National Academy of Sciences of the United States of America. 94: Raymond, M. & Rousset, F. (1995) GENEPOP (version 1.2): Population Genetics Software for Exact Tests and Ecumenicism. Journal of Heredity. 86: Rousset, F. (2008). Genepop'007: a Complete Reimplementation of the Genepop Software for Windows and Linux. Molecular Ecology Resources. 8: Ruusila, V., Pesonen, M., Tykkyläinen, R. & Wallen, M. (2002) Hirvikanta lähes ennallaan suurista kaatomääristä huolimatta. Riistantutkimuksen tiedote. 180: Ruusila, V., Aaltonen, I., Pesonen, M., Tykkyläinen, R. & Wallen, M. (2003) Etelä-Suomen hirvikanta pieneni Pohjois-Suomessa kasvu jatkui. Riistantutkimuksen tiedote. 187: Ruusila, V. & Malinen, J. (2006) Hirvi maailmalla Teoksessa Malinen, J. (toim.) Hirvenmetsästyksen käsikirja. Kustannusosakeyhtiö Otava. 263 s. Ryman, N. Baccus, R., Reuterwall, C. & Smith, M.H. (1981) Effective Population Size, Generation interval, and Potential Loss of Genetic Variability in Game Species Under Different Hunting Regimes. Oikos. 36: Ryman, N., Reuterwall, C., Nygren, K. & Nygren, T. (1980) Genetic Variation and Differentiation in Scandinavian Moose (Alces alces): Are Large Mammals Monomorfic?. Evolution. 34:
65 Røed, K.H. (1998) Microsatellite Variation in Scandinavian Cervidae Using Primers Derived from Bovidae. Hereditas. 129: Røed, K.H. & Midthjell, L. (1998) Microsatellites in Reindeer, Rangifer tarandus, and Their Use in Other Cervids. Molecular Ecology.7: Siivonen, L. & Sulkava, S. (1994) Pohjolan nisäkkäät. Kustannusosakeyhtiö Otava. 224 s. Schmölcke, U. & Zachos, F.E. (2005) Holocene Distribution and Etinction of the Moose (Alces alces, Cervidae) in Central Europe. Mammalian Biology. 70: Schwartz, C.C., Regelin, W.L, & Franzmann, A.W. (1982) Male Moose Succesfully Breed as Yearlings. Journal of Mammalogy. 63: Schmidt, J.I., Hundertmark, K.J., Bowyer, R.T. & McCracken, K.G (2009) Population Structure and Genetic Diversity of Moose in Alaska. Journal of Heredity. 100: Slatkin, M. (1985) Rare Alleles as Indicators of Gene Flow. Evolution. 39: Solberg, E. J., Loison, A., Sæther, B.-E. & Strand, O. ( 2000) Age-specific Harvest Mortality in a Norwegian Moose Alces alces Population. Wildlife Biology. 6: Solberg, E.J., Røed, K.H., Flagstad, Ø., Sæther, B.E., Heim, M., Andersen, R., Rolandsen, C.M. (2009) Elgens genetiske struktur i Norge. Nina Rapport s. Sweanor, P.Y. & Sandegren, F. (1989) Winter-Range Philopatry of Seasonally Migratory Moose. Journal of Applied Ecology. 26: Taberlet, P., Fumagalli, L., Wust-Saucy, A.G., Cosson, J.F. (1998) Comparative Phylogeography and Postglacial Colonization Routes in Europe. Molecular Ecology. 7: Telfer, E.S. (1984) Circumpolar Distribution and Habitat Requirements of Moose (Alces alces). Teoksessa Olson, R., Hastings, R. & Geddes, F. (toim.) Northern Ecology and Recource Management. The University of Alberta Press
66 Tiilikainen, R. (2010) The Effects of Selective Harvest on Life History of Moose Alces alces. Akateeminen väitöskirja. Luonnontieteiden ja metsätieteiden tiedekunta. Itä-Suomen yliopisto. 131 s. Van Oosterhout, C., Hutchinson, W.F., Wills, D.P.M. & Shipley, P (2004) MICRO- CHECKER: software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes. 4: Wang, J. (2005) Estimation of Effective Population Sizes from Data on Genetic Markers. Philosophical Transactions of the Royal Society B. 360: Waples, R.S. (2006) A Bias Correction for Estimates of Effective Population Size Based on Linkage Disequilibrium at Unlinked Gene Loci. Conservation Genetics. 7: Waples, R.S. & Do, C. (2008) LDNe: A Program for Estimating Effective Population Size from Data on Linkage Disequilibrium. Molecular Ecology Resources. 8: Waser, P.M. & Strobeck, C. (1998) Genetic Signatures of Interpopulation Dispersal. Trends in Ecology and Evolution. 13: Weber, J.L. (1990) Informativeness of Human (dc-da)n (dg-dt)n Polymorphisms. Genomics. 7: Williamson-Natesan, E.G. (2005) Comparison of Methods for Detecting Bottlenecks from Microsatellite Loci. Conservation Genetics. 6: Wilson, G.A., Strobeck, C., Wu, L., Coffin, J.W. (1997) Characterization of Microsatellite Loci in Caribou Rangifer tarandus, and Their Use in Other Artiodactyls. Molecular Ecology.6: Wilson, P.J., Grewal, S., Rodgers, A., Rempel, R., Saquet, J., Hristienko, H., Burrows, F., Peterson, R., & White, B.N. (2003) Genetic Variation and Population Structure of Moose (Alces alces) at Neutral and Functional DNA Loci. Canadian Journal of Zoology. 81:
67 Wright, S. (1931) Evolution in Mendelian Populations. Genetics. 16: Wright, S. (1943) Isolation by Distance. Genetics. 38: Zachos, F.E., Althoff, C., Steynitz, Y., Eckert, I. & Hartl, G.B. (2007) Genetic Analysis of an Isolated Red Deer (Cervus elaphus) Population Showing Signs of Inbreeding Depression. European Journal of Wildlife Research.53: Internet-lähteet: Kansalaisen karttapaikka (2001) Maanmittauslaitos. < > Riistaweb ( ) Metsästäjäin Keskusjärjestö & Riista- ja kalatalouden tutkimuslaitos. < >
68 8. LIITTEET Liite I. Hirviyksilöiden sukupuolet, verinäytteen keräyspaikkakunta sekä koordinaattisijainti. Nro. Sukupuoli Lounais-Suomi Leveysaste Pituusaste 1 uros Halikko uros Halikko naaras Halikko naaras Hanko naaras Kustavi uros Lieto naaras Lieto uros Marttila uros Marttila naaras Nauvo naaras Nauvo uros Perniö uros Perniö uros Pöytyä uros Pöytyä naaras Rymättylä uros Rymättylä naaras Vehmaa naaras Vahto naaras Vahto Nro. Sukupuoli Pohjanmaa Leveysaste Pituusaste 1 uros Alahärmä uros Alahärmä uros Isokyrö uros Isokyrö naaras Jurva naaras Kauhava uros Kauhava uros Kauhava naaras Mustasaari uros Laihia uros Laihia naaras Lappajärvi naaras Lappajärvi uros Purmo naaras Uusikaarlepyy naaras Vaasa uros Vaasa (Kvevlax) naaras Ylistaro uros Ylistaro naaras Maalahti naaras Maalahti
69 Nro. Sukupuoli Porkkala Leveysaste Pituusaste 1 uros Porkkalanniemi naaras Porkkalanniemi naaras Porkkalanniemi naaras Porkkalanniemi naaras Porkkalanniemi naaras Porkkalanniemi naaras Porkkalanniemi uros Porkkalanniemi Nro. Sukupuoli Karjala Leveysaste Pituusaste 1 naaras Enonkoski uros Enonkoski uros Enonkoski uros Kitee naaras Kitee uros Kitee uros Liperi naaras Liperi naaras Liperi uros Parikkala naaras Parikkala uros Parikkala uros Punkaharju naaras Pyhäselkä naaras Rautjärvi uros Rautjärvi uros Savonranta naaras Savonranta naaras Tohmajärvi uros Tohmajärvi naaras Tuupovaara naaras Tuupovaara Nro. Sukupuoli Kainuu Leveysaste Pituusaste 1 naaras Hyrynsalmi uros Hyrynsalmi naaras Kajaani naaras Kajaani uros Kajaani uros Kajaani naaras Kajaani naaras Paltamo uros Paltamo naaras Paltamo uros Ristijärvi uros Ristijärvi uros Sotkamo naaras Sotkamo naaras Sotkamo uros Sotkamo uros Suomussalmi naaras Suomussalmi
70 19 uros Suomussalmi uros Suomussalmi uros Suomussalmi uros Suomussalmi uros Kuhmo naaras Kuhmo uros Kuhmo naaras Kuhmo uros Kuhmo naaras Kuhmo Nro. Sukupuoli Itä-Lappi Leveysaste Pituusaste 1 naaras Kemijärvi naaras Kemijärvi naaras Kemijärvi naaras Kemijärvi uros Kuusamo naaras Kuusamo uros Posio uros Posio uros Salla uros Savukoski uros Savukoski uros Savukoski uros Savukoski naaras Savukoski uros Savukoski Nro. Sukupuoli Länsi-Lappi Leveysaste Pituusaste 1 uros Kemi uros Keminmaa naaras Keminmaa uros Keminmaa uros Kolari uros Kolari uros Kolari uros Kolari naaras Pello naaras Pello uros Pello naaras Pello naaras Tornio naaras Tornio naaras Ylitornio uros Ylitornio
71 Liite II. Käytettyjen kymmenen lokuksen alleelifrekvenssijakaumat. RT frekvenssi RT9 frekvenssi RT23 frekvenssi
72 RT frekvenssi RT27 frekvenssi RT30 frekvenssi CSSM001 frekvenssi
73 NVHRT frekvenssi NVHRT frekvenssi NVHRT24 frekvenssi
74 Liite III. Eri alueiden hirviksilöiden todennäköisyydet kuulua neljään eri Structure-ohjelman löytämään hirvipopulaatioon. Lounais-Suomi Porkkala Pohjanmaa Karjala Kainuu Itä-Lappi Länsi-Lappi 72
lpar1 IPB004065, IPB002277, and IPB Restriction Enyzme Differences from REBASE Gained in Variant Lost from Reference
 lpar1 IPB465, IPB2277, and IPB5385 Genomic Sequence Coding Sequence For help interpreting these results, view the PARSENP Introduction page. # View On Sequence Nucleotide Change Effect Restriction Enyzme
lpar1 IPB465, IPB2277, and IPB5385 Genomic Sequence Coding Sequence For help interpreting these results, view the PARSENP Introduction page. # View On Sequence Nucleotide Change Effect Restriction Enyzme
Hirvikannan koko ja vasatuotto pienenivät vuonna 2003
 Riistantutkimuksen tiedote :-. Helsinki 0..00 Hirvikannan koko ja vasatuotto pienenivät vuonna 00 Vesa Ruusila, Mauri Pesonen, Samuli Heikkinen, Arto Karhapää, Riitta Tykkyläinen ja Maija Wallén Suomen
Riistantutkimuksen tiedote :-. Helsinki 0..00 Hirvikannan koko ja vasatuotto pienenivät vuonna 00 Vesa Ruusila, Mauri Pesonen, Samuli Heikkinen, Arto Karhapää, Riitta Tykkyläinen ja Maija Wallén Suomen
Hirvikannan koko ja vasatuotto vuonna 2005
 Riistantutkimuksen tiedote :. Helsinki..00 Hirvikannan koko ja vasatuotto vuonna 00 Vesa Ruusila, Mauri Pesonen, Riitta Tykkyläinen, Arto Karhapää ja Maija Wallén Maamme hirvikannassa ei tapahtunut syksyn
Riistantutkimuksen tiedote :. Helsinki..00 Hirvikannan koko ja vasatuotto vuonna 00 Vesa Ruusila, Mauri Pesonen, Riitta Tykkyläinen, Arto Karhapää ja Maija Wallén Maamme hirvikannassa ei tapahtunut syksyn
Naudan perinnöllisen monimuotoisuuden tutkimus
 Naudan perinnöllisen monimuotoisuuden tutkimus Terhi Iso-Touru 25.5.2012 Emeritusprofessori Kalle Maijalan 85-vuotisjuhlaseminaari Naudan domestikaatio eli kesyttäminen yli 45 kiloa painavia kasvinsyöjälajeja
Naudan perinnöllisen monimuotoisuuden tutkimus Terhi Iso-Touru 25.5.2012 Emeritusprofessori Kalle Maijalan 85-vuotisjuhlaseminaari Naudan domestikaatio eli kesyttäminen yli 45 kiloa painavia kasvinsyöjälajeja
Dnro 269/301/2008. Maa- ja metsätalousministeriö Kala- ja riistaosasto PL VALTIONEUVOSTO
 9.3.2009 Dnro 269/301/2008 Maa- ja metsätalousministeriö Kala- ja riistaosasto PL 30 00023 VALTIONEUVOSTO Viite MMM 928/720/2008 Lausuntopyyntö 18.11.2008 Riista- ja kalatalouden tutkimuslaitoksen arvio
9.3.2009 Dnro 269/301/2008 Maa- ja metsätalousministeriö Kala- ja riistaosasto PL 30 00023 VALTIONEUVOSTO Viite MMM 928/720/2008 Lausuntopyyntö 18.11.2008 Riista- ja kalatalouden tutkimuslaitoksen arvio
Laboratorioanalyysit, vertailunäytteet ja tilastolliset menetelmät
 Jarmo Koskiniemi Maataloustieteiden laitos Helsingin yliopisto 0504151624 jarmo.koskiniemi@helsinki.fi 03.12.2015 Kolkunjoen taimenten geneettinen analyysi Näytteet Mika Oraluoma (Vesi-Visio osk) toimitti
Jarmo Koskiniemi Maataloustieteiden laitos Helsingin yliopisto 0504151624 jarmo.koskiniemi@helsinki.fi 03.12.2015 Kolkunjoen taimenten geneettinen analyysi Näytteet Mika Oraluoma (Vesi-Visio osk) toimitti
Hirvikannan koko ja vasatuotto vuonna 2004
 Riistantutkimuksen tiedote 0:-. Helsinki..00 Hirvikannan koko ja vasatuotto vuonna 00 Vesa Ruusila, Mauri Pesonen, Riitta Tykkyläinen, Arto Karhapää ja Maija Wallén Maamme hirvikanta pysyi syksyn 00 hirvijahdin
Riistantutkimuksen tiedote 0:-. Helsinki..00 Hirvikannan koko ja vasatuotto vuonna 00 Vesa Ruusila, Mauri Pesonen, Riitta Tykkyläinen, Arto Karhapää ja Maija Wallén Maamme hirvikanta pysyi syksyn 00 hirvijahdin
Dnro 269/301/2008. Maa- ja metsätalousministeriö Kala- ja riistaosasto PL VALTIONEUVOSTO
 31.12.2008 Dnro 269/301/2008 Maa- ja metsätalousministeriö Kala- ja riistaosasto PL 30 00023 VALTIONEUVOSTO Viite MMM 928/720/2008 Lausuntopyyntö 18.11.2008 Riista- ja kalatalouden tutkimuslaitoksen arvio
31.12.2008 Dnro 269/301/2008 Maa- ja metsätalousministeriö Kala- ja riistaosasto PL 30 00023 VALTIONEUVOSTO Viite MMM 928/720/2008 Lausuntopyyntö 18.11.2008 Riista- ja kalatalouden tutkimuslaitoksen arvio
Miten geenit elelevät populaatioissa, vieläpä pitkiä aikoja?
 Miten geenit elelevät populaatioissa, vieläpä pitkiä aikoja? Populaatio on lisääntymisyhteisö ja lisääntymisjatkumo Yksilöt ovat geenien tilapäisiä yhteenliittymiä, mutta populaatiossa geenit elelevät
Miten geenit elelevät populaatioissa, vieläpä pitkiä aikoja? Populaatio on lisääntymisyhteisö ja lisääntymisjatkumo Yksilöt ovat geenien tilapäisiä yhteenliittymiä, mutta populaatiossa geenit elelevät
S Laskennallinen systeemibiologia
 S-114.2510 Laskennallinen systeemibiologia 3. Harjoitus 1. Koska tilanne on Hardy-Weinbergin tasapainossa luonnonvalintaa lukuunottamatta, saadaan alleeleista muodostuvien eri tsygoottien genotyyppifrekvenssit
S-114.2510 Laskennallinen systeemibiologia 3. Harjoitus 1. Koska tilanne on Hardy-Weinbergin tasapainossa luonnonvalintaa lukuunottamatta, saadaan alleeleista muodostuvien eri tsygoottien genotyyppifrekvenssit
Villisikakanta-arvio tammikuussa
 Villisikakanta-arvio tammikuussa 2019 4.3.2019 Luonnonvarakeskus (Luke) Latokartanonkaari 9 00790 Helsinki Puhelin 0295 326 000 Y-tunnus 0244629-2 Tiivistelmä Suomessa oli tammikuun 2019 alussa arviolta
Villisikakanta-arvio tammikuussa 2019 4.3.2019 Luonnonvarakeskus (Luke) Latokartanonkaari 9 00790 Helsinki Puhelin 0295 326 000 Y-tunnus 0244629-2 Tiivistelmä Suomessa oli tammikuun 2019 alussa arviolta
Evoluutio. BI Elämä ja evoluutio Leena Kangas-Järviluoma
 Evoluutio BI Elämä ja evoluutio Leena Kangas-Järviluoma 1 Evoluutio lajinkehitystä, jossa eliölajit muuttuvat ja niistä voi kehittyä uusia lajeja on jatkunut elämän synnystä saakka, sillä ei ole päämäärää
Evoluutio BI Elämä ja evoluutio Leena Kangas-Järviluoma 1 Evoluutio lajinkehitystä, jossa eliölajit muuttuvat ja niistä voi kehittyä uusia lajeja on jatkunut elämän synnystä saakka, sillä ei ole päämäärää
111111111111111111 II IIII II II 11111111111111111111
 111111111111111111 II IIII II II 11111111111111111111 F10008 (12) PATENTTIJULKAISU PATENTSICRIFT (10) FI B (45) Patentti myönnetty - Patent beviljats 15.04.1999 (51) Kv.lk.6 - Int.k1.6 SUOMI-FINLAND (FI)
111111111111111111 II IIII II II 11111111111111111111 F10008 (12) PATENTTIJULKAISU PATENTSICRIFT (10) FI B (45) Patentti myönnetty - Patent beviljats 15.04.1999 (51) Kv.lk.6 - Int.k1.6 SUOMI-FINLAND (FI)
SISÄLLYSLUETTELO LYHENTEET JOHDANTO...
 SISÄLLYSLUETTELO LYHENTEET...4 1 JOHDANTO...5 1.1 Ilves (Lynx lynx)...5 1.1.1 Levinneisyys ja elinympäristö...5 1.1.2 Elinpiiri ja ravinto...6 1.1.3 Lisääntyminen, dispersaali ja kuolleisuus...8 1.1.4
SISÄLLYSLUETTELO LYHENTEET...4 1 JOHDANTO...5 1.1 Ilves (Lynx lynx)...5 1.1.1 Levinneisyys ja elinympäristö...5 1.1.2 Elinpiiri ja ravinto...6 1.1.3 Lisääntyminen, dispersaali ja kuolleisuus...8 1.1.4
LABORATORIOTYÖ: AGAROOSIGEELIELEKTROFOREESI
 LABORATORIOTYÖ: AGAROOSIGEELIELEKTROFOREESI Agaroosigeelielektroforeesi (AGE) on yksinkertainen ja tehokas menetelmä erikokoisten DNAjaksojen erottamiseen, tunnistamiseen ja puhdistamiseen. Eri valmistajien
LABORATORIOTYÖ: AGAROOSIGEELIELEKTROFOREESI Agaroosigeelielektroforeesi (AGE) on yksinkertainen ja tehokas menetelmä erikokoisten DNAjaksojen erottamiseen, tunnistamiseen ja puhdistamiseen. Eri valmistajien
Riista- ja kalatalouden tutkimuslaitoksen lausunto susitilanteesta
 15.2.2010 Dnro 85/301/2010 Maa- ja metsätalousministeriö Kala- ja riistaosasto PL 30 00023 VALTIONEUVOSTO Viite MMM 513/444/2010 Lausuntopyyntö 4.2.2010 Riista- ja kalatalouden tutkimuslaitoksen lausunto
15.2.2010 Dnro 85/301/2010 Maa- ja metsätalousministeriö Kala- ja riistaosasto PL 30 00023 VALTIONEUVOSTO Viite MMM 513/444/2010 Lausuntopyyntö 4.2.2010 Riista- ja kalatalouden tutkimuslaitoksen lausunto
Sukusiitoksesta sukulaistumiseen - jalostustietojärjestelmä työkaluna. Rovaniemi Susanna Back, Suomen Hippos ry
 Sukusiitoksesta sukulaistumiseen - jalostustietojärjestelmä työkaluna Rovaniemi 22.3.2018 Susanna Back, Suomen Hippos ry Sukulaisuussuhde Kahden yksilön yhteisten geenien todennäköinen osuus Riippuu eläinten
Sukusiitoksesta sukulaistumiseen - jalostustietojärjestelmä työkaluna Rovaniemi 22.3.2018 Susanna Back, Suomen Hippos ry Sukulaisuussuhde Kahden yksilön yhteisten geenien todennäköinen osuus Riippuu eläinten
III Perinnöllisyystieteen perusteita
 Perinnöllisyystieteen perusteita III Perinnöllisyystieteen perusteita 15. Populaatiogenetiikka ja evoluutio 1. Avainsanat 2. Evoluutio muuttaa geenipoolia 3. Mihin valinta kohdistuu? 4. Yksilön muuntelua
Perinnöllisyystieteen perusteita III Perinnöllisyystieteen perusteita 15. Populaatiogenetiikka ja evoluutio 1. Avainsanat 2. Evoluutio muuttaa geenipoolia 3. Mihin valinta kohdistuu? 4. Yksilön muuntelua
Molekyylipopulaatiogenetiikka
 Molekyylipopulaatiogenetiikka Hedrick 2005, kappale 8, pp. 452-462, 428-449 Demografian ja valinnan vaikutukset koalesenssipuihin Valinnan havaitseminen TajimanD Divergenssin ja polymorfismin vertailu
Molekyylipopulaatiogenetiikka Hedrick 2005, kappale 8, pp. 452-462, 428-449 Demografian ja valinnan vaikutukset koalesenssipuihin Valinnan havaitseminen TajimanD Divergenssin ja polymorfismin vertailu
Darwin nuorena. Darwinin syntymästä on 201 vuotta ja "Lajien synnystä" 151 vuotta.
 Darwin nuorena Darwinin syntymästä on 201 vuotta ja "Lajien synnystä" 151 vuotta. Evoluutio on dynaaminen prosessi, joka pohjautuu populaatiogenetiikkaan Darwinin suurin ansio oli luonnonvalinta adaptaation
Darwin nuorena Darwinin syntymästä on 201 vuotta ja "Lajien synnystä" 151 vuotta. Evoluutio on dynaaminen prosessi, joka pohjautuu populaatiogenetiikkaan Darwinin suurin ansio oli luonnonvalinta adaptaation
Evoluutiovoimat. Ydinkysymykset. Mikä on mutaation, valinnan ja sattuman merkitys evoluutiossa?
 Evoluutiovoimat Ydinkysymykset Mikä on mutaation, valinnan ja sattuman merkitys evoluutiossa? -sattuman sysäily: populaatiokoon vaikutus -valinta: positiivinen, tasapainottava ja negatiivinen -mutaatiot:
Evoluutiovoimat Ydinkysymykset Mikä on mutaation, valinnan ja sattuman merkitys evoluutiossa? -sattuman sysäily: populaatiokoon vaikutus -valinta: positiivinen, tasapainottava ja negatiivinen -mutaatiot:
Hirven biologia Mikael Wikström 2015
 Hirven biologia Mikael Wikström 2015 Sisältö Lisääntymiskäyttäytyminen Kannanvaihtelun perusmekanismit Kannan sääteleminen Lisääntymiskäyttäytyminen Kiima syyskuussa ja lokakuun alussa Kiimaan tulossa
Hirven biologia Mikael Wikström 2015 Sisältö Lisääntymiskäyttäytyminen Kannanvaihtelun perusmekanismit Kannan sääteleminen Lisääntymiskäyttäytyminen Kiima syyskuussa ja lokakuun alussa Kiimaan tulossa
Lisääntyminen. BI1 Elämä ja evoluutio Leena kangas-järviluoma
 Lisääntyminen BI1 Elämä ja evoluutio Leena kangas-järviluoma säilyä hengissä ja lisääntyä kaksi tapaa lisääntyä suvuton suvullinen suvuttomassa lisääntymisessä uusi yksilö syntyy ilman sukusoluja suvullisessa
Lisääntyminen BI1 Elämä ja evoluutio Leena kangas-järviluoma säilyä hengissä ja lisääntyä kaksi tapaa lisääntyä suvuton suvullinen suvuttomassa lisääntymisessä uusi yksilö syntyy ilman sukusoluja suvullisessa
Suurpetojen lukumäärä ja lisääntyminen vuonna 2000
 1 Riistantutkimuksen tiedote 175:1-6. Helsinki, 15.8.2001. Suurpetojen lukumäärä ja lisääntyminen vuonna 2000 Ilpo Kojola Karhukannan kasvu näyttää olevan tasaantumassa. Karhun vähimmäiskanta oli vuoden
1 Riistantutkimuksen tiedote 175:1-6. Helsinki, 15.8.2001. Suurpetojen lukumäärä ja lisääntyminen vuonna 2000 Ilpo Kojola Karhukannan kasvu näyttää olevan tasaantumassa. Karhun vähimmäiskanta oli vuoden
Metsäpeuran paluu Metsäpeuran EU LIFE hankkeen kuulumisia. Hanna-Maija Lahtinen
 Metsäpeuran paluu Metsäpeuran EU LIFE hankkeen kuulumisia Hanna-Maija Lahtinen 6.4.2017 Mikä metsäpeura? Mistä peurasta on kyse? www.suomenluonto.fi Valkohäntäpeura eli -kauris www.maaseutumedia.fi Kuusipeura
Metsäpeuran paluu Metsäpeuran EU LIFE hankkeen kuulumisia Hanna-Maija Lahtinen 6.4.2017 Mikä metsäpeura? Mistä peurasta on kyse? www.suomenluonto.fi Valkohäntäpeura eli -kauris www.maaseutumedia.fi Kuusipeura
Saimaannorppanaaraiden ( Phoca hispida saimensis)
 Saimaannorppanaaraiden (Phoca hispida saimensis) yksilöntunnistus ja pesäpaikkauskollisuuden selvittäminen geneettisin menetelmin pesäpaikoilta kerätyistä istukkanäytteistä Hanna Buuri Pro gradu tutkimus
Saimaannorppanaaraiden (Phoca hispida saimensis) yksilöntunnistus ja pesäpaikkauskollisuuden selvittäminen geneettisin menetelmin pesäpaikoilta kerätyistä istukkanäytteistä Hanna Buuri Pro gradu tutkimus
Uhanalaisuusarvioinnin keskeiset käsitteet. Annika Uddström, Suomen ympäristökeskus,
 Uhanalaisuusarvioinnin keskeiset käsitteet Annika Uddström, Suomen ympäristökeskus, 2.2.2017 Populaatio ja populaatiokoko (kriteerit A, C ja D) Populaatiolla tarkoitetaan lajin tarkastelualueella elävää
Uhanalaisuusarvioinnin keskeiset käsitteet Annika Uddström, Suomen ympäristökeskus, 2.2.2017 Populaatio ja populaatiokoko (kriteerit A, C ja D) Populaatiolla tarkoitetaan lajin tarkastelualueella elävää
Avainsanat. populaatio yksilöiden levintätyypit ikärakenne sukupuolijakauma populaation kasvumallit ympäristön vastus elinkiertostrategiat
 Avainsanat populaatio yksilöiden levintätyypit ikärakenne sukupuolijakauma populaation kasvumallit ympäristön vastus elinkiertostrategiat Populaatio Populaatiolla tarkoitetaan tietyllä alueella tiettynä
Avainsanat populaatio yksilöiden levintätyypit ikärakenne sukupuolijakauma populaation kasvumallit ympäristön vastus elinkiertostrategiat Populaatio Populaatiolla tarkoitetaan tietyllä alueella tiettynä
Kemijoen Sihtuunan ja Rautuojan taimenten geneettinen analyysi Jarmo Koskiniemi, Helsingin yliopisto, maataloustieteiden osasto
 21.12.2018 Kemijoen Sihtuunan ja Rautuojan taimenten geneettinen analyysi Jarmo Koskiniemi, Helsingin yliopisto, maataloustieteiden osasto Näytteet Jarmo Huhtala toimitti syksyllä 2018 Helsingin yliopiston
21.12.2018 Kemijoen Sihtuunan ja Rautuojan taimenten geneettinen analyysi Jarmo Koskiniemi, Helsingin yliopisto, maataloustieteiden osasto Näytteet Jarmo Huhtala toimitti syksyllä 2018 Helsingin yliopiston
PCR - tekniikka elintarvikeanalytiikassa
 PCR - tekniikka elintarvikeanalytiikassa Listerian, Salmonellan ja kampylobakteerien tunnistus elintarvikkeista ja rehuista 29.11.2012 Eva Fredriksson-Lidsle Listeria monocytogenes Salmonella (spp) Campylobacter
PCR - tekniikka elintarvikeanalytiikassa Listerian, Salmonellan ja kampylobakteerien tunnistus elintarvikkeista ja rehuista 29.11.2012 Eva Fredriksson-Lidsle Listeria monocytogenes Salmonella (spp) Campylobacter
Riistapäivät 2015 Markus Melin Itä Suomen Yliopisto Metsätieteiden osasto markus.melin@uef.fi
 Riistapäivät 2015 Markus Melin Itä Suomen Yliopisto Metsätieteiden osasto markus.melin@uef.fi Laserkeilaus pähkinänkuoressa Aktiivista kaukokartoitusta, joka tuottaa 3D aineistoa (vrt. satelliitti- ja
Riistapäivät 2015 Markus Melin Itä Suomen Yliopisto Metsätieteiden osasto markus.melin@uef.fi Laserkeilaus pähkinänkuoressa Aktiivista kaukokartoitusta, joka tuottaa 3D aineistoa (vrt. satelliitti- ja
Riista- ja kalatalouden tutkimuslaitos 12.12.2001 Marja-Liisa Koljonen. 1.1. Yleistä
 Geneettinen monimuotoisuus Suomen luonnonvaraisissa ja viljellyissä lohikannoissa ja tehollisten populaatiokokojen osuus todellisista emokalamääristä DNA:n mikrosatelliittimuuntelun avulla arvioituna Riista-
Geneettinen monimuotoisuus Suomen luonnonvaraisissa ja viljellyissä lohikannoissa ja tehollisten populaatiokokojen osuus todellisista emokalamääristä DNA:n mikrosatelliittimuuntelun avulla arvioituna Riista-
Etelä-Suomen hirvikanta pieneni Pohjois- Suomessa kasvu jatkui
 1 Riistantutkimuksen tiedote 187:1-11. Helsinki 8.8.2003 Etelä-Suomen hirvikanta pieneni Pohjois- Suomessa kasvu jatkui Vesa Ruusila, Ilkka Aaltonen, Mauri Pesonen, Riitta Tykkyläinen ja Maija Wallén Hirvien
1 Riistantutkimuksen tiedote 187:1-11. Helsinki 8.8.2003 Etelä-Suomen hirvikanta pieneni Pohjois- Suomessa kasvu jatkui Vesa Ruusila, Ilkka Aaltonen, Mauri Pesonen, Riitta Tykkyläinen ja Maija Wallén Hirvien
loppuraportti Japo Jussila, Itä-Suomen yliopisto ja Raputietokeskus ry. Vesa Tiitinen, Etelä-Karjalan kalatalouskeskus
 1/5 loppuraportti SUOLEN TYHJENEMINEN RAPUSUIHKUSSA Japo Jussila, Itä-Suomen yliopisto ja Raputietokeskus ry. Vesa Tiitinen, Etelä-Karjalan kalatalouskeskus 1 Testin työt Täpläravut sijoitettiin RapuSuihkuun
1/5 loppuraportti SUOLEN TYHJENEMINEN RAPUSUIHKUSSA Japo Jussila, Itä-Suomen yliopisto ja Raputietokeskus ry. Vesa Tiitinen, Etelä-Karjalan kalatalouskeskus 1 Testin työt Täpläravut sijoitettiin RapuSuihkuun
Perinnöllisyyden perusteita
 Perinnöllisyyden perusteita Eero Lukkari Tämä artikkeli kertoo perinnöllisyyden perusmekanismeista johdantona muille jalostus- ja terveysaiheisille artikkeleille. Koirien, kuten muidenkin eliöiden, perimä
Perinnöllisyyden perusteita Eero Lukkari Tämä artikkeli kertoo perinnöllisyyden perusmekanismeista johdantona muille jalostus- ja terveysaiheisille artikkeleille. Koirien, kuten muidenkin eliöiden, perimä
Metsähanhen hoitosuunnitelmat. Photo Asko Kettunen
 Metsähanhen hoitosuunnitelmat Photo Asko Kettunen Metsähanhen kaksi alalajia Suomessa liikkuu syksyllä metsähanhen kahta eri alalajia. Suomessa pesii taigametsähanhi, joka taantui merkittävästi viime vuosikymmenien
Metsähanhen hoitosuunnitelmat Photo Asko Kettunen Metsähanhen kaksi alalajia Suomessa liikkuu syksyllä metsähanhen kahta eri alalajia. Suomessa pesii taigametsähanhi, joka taantui merkittävästi viime vuosikymmenien
Suden ekologiaa Ilpo Kojola, Luonnonvarakeskus (Luke), Rovaniemi
 Suden ekologiaa Ilpo Kojola, Luonnonvarakeskus (Luke), Rovaniemi Suden levinneisyys esiintymisalue v. 2000 hävinnyt - alkuperäisestä esiintymisalueesta jäljellä 67 % vuonna 2000 Ihmisasutuksen tiheys asukkaita/km
Suden ekologiaa Ilpo Kojola, Luonnonvarakeskus (Luke), Rovaniemi Suden levinneisyys esiintymisalue v. 2000 hävinnyt - alkuperäisestä esiintymisalueesta jäljellä 67 % vuonna 2000 Ihmisasutuksen tiheys asukkaita/km
Molekyylievoluutio. (753327A, S), 4 op syksy Photo Marie-Hélène Müller
 Molekyylievoluutio (753327A, 753627S), 4 op syksy 2011 Photo Marie-Hélène Müller Molekyylievoluutio (753327A, 753627S), 4 op, syksy 2011 -Helmi Kuittinen p. 553 1803, PT312, helmi.kuittinen@oulu.fi harj.
Molekyylievoluutio (753327A, 753627S), 4 op syksy 2011 Photo Marie-Hélène Müller Molekyylievoluutio (753327A, 753627S), 4 op, syksy 2011 -Helmi Kuittinen p. 553 1803, PT312, helmi.kuittinen@oulu.fi harj.
pitkittäisaineistoissa
 Puuttuvan tiedon ongelma p. 1/18 Puuttuvan tiedon ongelma pitkittäisaineistoissa Tapio Nummi tan@uta.fi Matematiikan, tilastotieteen ja filosofian laitos Tampereen yliopisto mtl.uta.fi/tilasto/sekamallit/puupitkit.pdf
Puuttuvan tiedon ongelma p. 1/18 Puuttuvan tiedon ongelma pitkittäisaineistoissa Tapio Nummi tan@uta.fi Matematiikan, tilastotieteen ja filosofian laitos Tampereen yliopisto mtl.uta.fi/tilasto/sekamallit/puupitkit.pdf
Liito-orava kartoitus Nouvanlahden ulkoilualueelle sekä eteläisen Kilpijärven länsirannalle.
 Liito-orava kartoitus Nouvanlahden ulkoilualueelle sekä eteläisen Kilpijärven länsirannalle. Tarmo Saastamoinen 2010. Kuva.1 Kaatunut kuusenrunko Nouvanlahdesta. LIITO-ORAVA: Liito-orava (pteromys volans)on
Liito-orava kartoitus Nouvanlahden ulkoilualueelle sekä eteläisen Kilpijärven länsirannalle. Tarmo Saastamoinen 2010. Kuva.1 Kaatunut kuusenrunko Nouvanlahdesta. LIITO-ORAVA: Liito-orava (pteromys volans)on
VARESJÄRVI KOEKALASTUS
 Varsinais-Suomen Kalavesien Hoito Oy Puutarhakatu 19 A 20100 TURKU www.silakka.info VARESJÄRVI KOEKALASTUS 2012 Chris Karppinen Varsinais-suomen kalavesien Hoito Oy 1. Johdanto Maataloustuottajain säätiö
Varsinais-Suomen Kalavesien Hoito Oy Puutarhakatu 19 A 20100 TURKU www.silakka.info VARESJÄRVI KOEKALASTUS 2012 Chris Karppinen Varsinais-suomen kalavesien Hoito Oy 1. Johdanto Maataloustuottajain säätiö
Hiiltä varastoituu ekosysteemeihin
 Hiiltä varastoituu ekosysteemeihin BIOS 3 jakso 3 Hiili esiintyy ilmakehässä epäorgaanisena hiilidioksidina ja eliöissä orgaanisena hiiliyhdisteinä. Hiili siirtyy ilmakehästä eliöihin ja eliöistä ilmakehään:
Hiiltä varastoituu ekosysteemeihin BIOS 3 jakso 3 Hiili esiintyy ilmakehässä epäorgaanisena hiilidioksidina ja eliöissä orgaanisena hiiliyhdisteinä. Hiili siirtyy ilmakehästä eliöihin ja eliöistä ilmakehään:
Populaatiosimulaattori. Petteri Hintsanen HIIT perustutkimusyksikkö Helsingin yliopisto
 Populaatiosimulaattori Petteri Hintsanen HIIT perustutkimusyksikkö Helsingin yliopisto Kromosomit Ihmisen perimä (genomi) on jakaantunut 23 kromosomipariin Jokaisen parin toinen kromosomi on peritty isältä
Populaatiosimulaattori Petteri Hintsanen HIIT perustutkimusyksikkö Helsingin yliopisto Kromosomit Ihmisen perimä (genomi) on jakaantunut 23 kromosomipariin Jokaisen parin toinen kromosomi on peritty isältä
pitkittäisaineistoissa
 Puuttuvan tiedon käsittelystä p. 1/18 Puuttuvan tiedon käsittelystä pitkittäisaineistoissa Tapio Nummi tan@uta.fi Matematiikan, tilastotieteen ja filosofian laitos Tampereen yliopisto Puuttuvan tiedon
Puuttuvan tiedon käsittelystä p. 1/18 Puuttuvan tiedon käsittelystä pitkittäisaineistoissa Tapio Nummi tan@uta.fi Matematiikan, tilastotieteen ja filosofian laitos Tampereen yliopisto Puuttuvan tiedon
Hirvikannan kasvu pysähtyi, mutta naaraita säästävä verotus pitänyt vasatuoton korkeana
 1 Riistantutkimuksen tiedote 173:1-12. Helsinki, 12.6.2001. Hirvikannan kasvu pysähtyi, mutta naaraita säästävä verotus pitänyt vasatuoton korkeana Vesa Ruusila, Mauri Pesonen, Riitta Tykkyläinen ja Maija
1 Riistantutkimuksen tiedote 173:1-12. Helsinki, 12.6.2001. Hirvikannan kasvu pysähtyi, mutta naaraita säästävä verotus pitänyt vasatuoton korkeana Vesa Ruusila, Mauri Pesonen, Riitta Tykkyläinen ja Maija
Koko maan ilveskanta-arvion taustasta ja erityisesti Etelä-Hämeen arviosta. Tiedosta ratkaisuja kestäviin valintoihin
 Koko maan ilveskanta-arvion taustasta ja erityisesti Etelä-Hämeen arviosta Tiedosta ratkaisuja kestäviin valintoihin Erillislaskentojen pentuetulos talvi 2012/2013 Ensimmäinen tieto lehdistössä Pentueet
Koko maan ilveskanta-arvion taustasta ja erityisesti Etelä-Hämeen arviosta Tiedosta ratkaisuja kestäviin valintoihin Erillislaskentojen pentuetulos talvi 2012/2013 Ensimmäinen tieto lehdistössä Pentueet
HIRVIKANNAN SÄÄTELY Vakauden esteet ja mahdollisuudet Tuire Nygrén, RKTL
 HIRVIKANNAN SÄÄTELY Vakauden esteet ja mahdollisuudet Tuire Nygrén, RKTL Riistabiologian kurssi Mekrijärven tutkimusasemalla 12.4.2010 Tiivistetysti Suomen hirvikannan säätelystä: Viime sotien jälkeen
HIRVIKANNAN SÄÄTELY Vakauden esteet ja mahdollisuudet Tuire Nygrén, RKTL Riistabiologian kurssi Mekrijärven tutkimusasemalla 12.4.2010 Tiivistetysti Suomen hirvikannan säätelystä: Viime sotien jälkeen
T Luonnollisen kielen tilastollinen käsittely Vastaukset 3, ti , 8:30-10:00 Kollokaatiot, Versio 1.1
 T-61.281 Luonnollisen kielen tilastollinen käsittely Vastaukset 3, ti 10.2.2004, 8:30-10:00 Kollokaatiot, Versio 1.1 1. Lasketaan ensin tulokset sanaparille valkoinen, talo käsin: Frekvenssimenetelmä:
T-61.281 Luonnollisen kielen tilastollinen käsittely Vastaukset 3, ti 10.2.2004, 8:30-10:00 Kollokaatiot, Versio 1.1 1. Lasketaan ensin tulokset sanaparille valkoinen, talo käsin: Frekvenssimenetelmä:
Algoritmit lyhyiden sekvenssien rinnastamiseen referenssigenomia vasten. Krista Longi
 Algoritmit lyhyiden sekvenssien rinnastamiseen referenssigenomia vasten. Krista Longi 19.05.2014 DNA:n sekvensointi DNA:n pilkotaan lyhyiksi mallipalasiksi, templaateiksi, joiden emäsjärjestys selvitetään.
Algoritmit lyhyiden sekvenssien rinnastamiseen referenssigenomia vasten. Krista Longi 19.05.2014 DNA:n sekvensointi DNA:n pilkotaan lyhyiksi mallipalasiksi, templaateiksi, joiden emäsjärjestys selvitetään.
Suurpetojen lukumäärä ja lisääntyminen vuonna 2001
 1 Riistantutkimuksen tiedote 182: 1-7. Helsinki 3.9.2 Suurpetojen lukumäärä ja lisääntyminen vuonna 1 Ilpo Kojola Riista- ja kalatalouden tutkimuslaitoksen suurpetoseurannan mukaan maamme karhukanta pysyi
1 Riistantutkimuksen tiedote 182: 1-7. Helsinki 3.9.2 Suurpetojen lukumäärä ja lisääntyminen vuonna 1 Ilpo Kojola Riista- ja kalatalouden tutkimuslaitoksen suurpetoseurannan mukaan maamme karhukanta pysyi
Biologia. Pakolliset kurssit. 1. Eliömaailma (BI1)
 Biologia Pakolliset kurssit 1. Eliömaailma (BI1) tuntee elämän tunnusmerkit ja perusedellytykset sekä tietää, miten elämän ilmiöitä tutkitaan ymmärtää, mitä luonnon monimuotoisuus biosysteemien eri tasoilla
Biologia Pakolliset kurssit 1. Eliömaailma (BI1) tuntee elämän tunnusmerkit ja perusedellytykset sekä tietää, miten elämän ilmiöitä tutkitaan ymmärtää, mitä luonnon monimuotoisuus biosysteemien eri tasoilla
Suuria eroja eri rotujen tehollisessa populaatiokoossa
 1 / 7 Suuria eroja eri rotujen tehollisessa populaatiokoossa Elina Paakala Koirarotujen perinnöllinen monimuotoisuus on ajankohtainen puheenaihe. Koiramme-lehdessä on aiheesta juttuja jokseenkin joka numerossa.
1 / 7 Suuria eroja eri rotujen tehollisessa populaatiokoossa Elina Paakala Koirarotujen perinnöllinen monimuotoisuus on ajankohtainen puheenaihe. Koiramme-lehdessä on aiheesta juttuja jokseenkin joka numerossa.
Metsähanhen metsästyksen EETTISET OHJEET
 Metsähanhen metsästyksen EETTISET OHJEET 2 Suomessa esiintyy kaksi metsähanhen alalajia; taigametsähanhi (Anser fabalis fabalis) ja tundrametsähanhi (Anser fabalis rossicus). Taigametsähanhi esiintyy meillä
Metsähanhen metsästyksen EETTISET OHJEET 2 Suomessa esiintyy kaksi metsähanhen alalajia; taigametsähanhi (Anser fabalis fabalis) ja tundrametsähanhi (Anser fabalis rossicus). Taigametsähanhi esiintyy meillä
Vesilintujen runsaus ja poikastuotto vuonna 2006
 1 Riistantutkimuksen tiedote 209:1-5. Helsinki 16.8.6 Vesilintujen runsaus ja poikastuotto vuonna 6 Hannu Pöysä, Marcus Wikman, Esa Lammi ja Risto A. Väisänen Vesilinnuston kokonaiskanta pysyi viime vuoden
1 Riistantutkimuksen tiedote 209:1-5. Helsinki 16.8.6 Vesilintujen runsaus ja poikastuotto vuonna 6 Hannu Pöysä, Marcus Wikman, Esa Lammi ja Risto A. Väisänen Vesilinnuston kokonaiskanta pysyi viime vuoden
HIRVIKANNAN SÄÄTELY Perusbiologiaa
 HIRVIKANNAN SÄÄTELY Perusbiologiaa Tuire Nygrén, RKTL Riistabiologian kurssi Mekrijärven tutkimusasemalla 12.4.2010 Kuva: Jari K. 020210 Kannansäätelyn biologista tietopohjaa Hirvisaaliit Valikoiva verotus
HIRVIKANNAN SÄÄTELY Perusbiologiaa Tuire Nygrén, RKTL Riistabiologian kurssi Mekrijärven tutkimusasemalla 12.4.2010 Kuva: Jari K. 020210 Kannansäätelyn biologista tietopohjaa Hirvisaaliit Valikoiva verotus
RIIHIMÄKI AROLAMPI 1 JA HERAJOKI ETELÄINEN LIITO-ORAVASELVITYS 2017
 TUTKIMUSRAPORTTI 5.4.2017 RIIHIMÄKI AROLAMPI 1 JA HERAJOKI ETELÄINEN LIITO-ORAVASELVITYS 2017 Riihimäen kaupunki Tekijä: Laura Ahopelto SISÄLLYS 1 Johdanto... 4 2 Menetelmä... 5 3 Tulokset... 5 4 Muita
TUTKIMUSRAPORTTI 5.4.2017 RIIHIMÄKI AROLAMPI 1 JA HERAJOKI ETELÄINEN LIITO-ORAVASELVITYS 2017 Riihimäen kaupunki Tekijä: Laura Ahopelto SISÄLLYS 1 Johdanto... 4 2 Menetelmä... 5 3 Tulokset... 5 4 Muita
Biodiversiteetti Luonnon monimuotoisuus Naturens mångfald Biodiversity
 Biodiversiteetti Luonnon monimuotoisuus Naturens mångfald Biodiversity Esa Koskenniemi, Etelä-Pohjanmaan ELY-keskus/ ELY-centralen i Södra Österbotten 19.4.2012 1 Miksi tästä nyt puhutaan. Biodiversiteettistrategian
Biodiversiteetti Luonnon monimuotoisuus Naturens mångfald Biodiversity Esa Koskenniemi, Etelä-Pohjanmaan ELY-keskus/ ELY-centralen i Södra Österbotten 19.4.2012 1 Miksi tästä nyt puhutaan. Biodiversiteettistrategian
SAIMAANNORPPA Kannan koon arvioinnista Tero Sipilä & Tuomo Kokkonen Metsähallitus, Etelä-Suomen Luontopalvelut Akselinkatu 8, 57130, Savonlinna
 Saimaannorppa, ilmastonmuutos ja kalastus seminaari. Rantasalmi 28.5.21 SAIMAANNORPPA Kannan koon arvioinnista Tero Sipilä & Tuomo Kokkonen Metsähallitus, Etelä-Suomen Luontopalvelut Akselinkatu 8, 5713,
Saimaannorppa, ilmastonmuutos ja kalastus seminaari. Rantasalmi 28.5.21 SAIMAANNORPPA Kannan koon arvioinnista Tero Sipilä & Tuomo Kokkonen Metsähallitus, Etelä-Suomen Luontopalvelut Akselinkatu 8, 5713,
¼ ¼ joten tulokset ovat muuttuneet ja nimenomaan huontontuneet eivätkä tulleet paremmiksi.
 10.11.2006 1. Pituushyppääjä on edellisenä vuonna hypännyt keskimäärin tuloksen. Valmentaja poimii tämän vuoden harjoitusten yhteydessä tehdyistä muistiinpanoista satunnaisesti kymmenen harjoitushypyn
10.11.2006 1. Pituushyppääjä on edellisenä vuonna hypännyt keskimäärin tuloksen. Valmentaja poimii tämän vuoden harjoitusten yhteydessä tehdyistä muistiinpanoista satunnaisesti kymmenen harjoitushypyn
Vaeltavan taviokuurnapopulaation (Pinicola enucleator) alkuperän ja sukupuolen määritys DNA:n ja vakaiden isotooppien avulla.
 Vaeltavan taviokuurnapopulaation (Pinicola enucleator) alkuperän ja sukupuolen määritys DNA:n ja vakaiden isotooppien avulla. Konsta Isometsä Pro Gradu -tutkielma Oulun yliopisto Biologian laitos Genetiikan
Vaeltavan taviokuurnapopulaation (Pinicola enucleator) alkuperän ja sukupuolen määritys DNA:n ja vakaiden isotooppien avulla. Konsta Isometsä Pro Gradu -tutkielma Oulun yliopisto Biologian laitos Genetiikan
Miltä näyttää kotimaisten rotujemme perinnöllinen monimuotoisuus? Jalostusvalinta on merkittävin koirarotujen monimuotoisuutta vähentävä tekijä
 Miltä näyttää kotimaisten rotujemme perinnöllinen monimuotoisuus? Katariina Mäki ja Mauri Kumpulainen Kennelliitolla on meneillään tilaustutkimus kotimaisten rotujen DLA-monimuotoisuudesta. Mukana ovat
Miltä näyttää kotimaisten rotujemme perinnöllinen monimuotoisuus? Katariina Mäki ja Mauri Kumpulainen Kennelliitolla on meneillään tilaustutkimus kotimaisten rotujen DLA-monimuotoisuudesta. Mukana ovat
Saarijärven rhy Hirvikanta
 Saarijärven rhy Hirvikanta 1995 2012 Välissä parikymmentä vuotta ja useita kaatoja SAARIJÄRVEN RHY HIRVIHAVAINTOKORTTIEN YHTEENVETO SEUROITTAIN/SEURUEITTAIN 1995 2012 Tulokset eivät ole ns. tarkkoja lukuarvoja
Saarijärven rhy Hirvikanta 1995 2012 Välissä parikymmentä vuotta ja useita kaatoja SAARIJÄRVEN RHY HIRVIHAVAINTOKORTTIEN YHTEENVETO SEUROITTAIN/SEURUEITTAIN 1995 2012 Tulokset eivät ole ns. tarkkoja lukuarvoja
DNA sukututkimuksen tukena
 Järvenpää 12,2,2019 Teuvo Ikonen teuvo.ikonen@welho.com DNA sukututkimuksen tukena DNA sukututkimuksessa (Peter Sjölund: Släktforska med DNA) tiesitkö, että olet kävelevä sukukirja? on kuin lukisit kirjaa
Järvenpää 12,2,2019 Teuvo Ikonen teuvo.ikonen@welho.com DNA sukututkimuksen tukena DNA sukututkimuksessa (Peter Sjölund: Släktforska med DNA) tiesitkö, että olet kävelevä sukukirja? on kuin lukisit kirjaa
Riistatiedon merkitys vieraslajitilanteen. esimerkkinä lajipari euroopanmajava - kanadanmajava. Kaarina Kauhala Luke
 Riistatiedon merkitys vieraslajitilanteen hallinnassa: esimerkkinä lajipari euroopanmajava - kanadanmajava Kaarina Kauhala Luke Euroopanmajava Metsästettiin sukupuuttoon 1800-luvulla (1868). Takaisinistutettu
Riistatiedon merkitys vieraslajitilanteen hallinnassa: esimerkkinä lajipari euroopanmajava - kanadanmajava Kaarina Kauhala Luke Euroopanmajava Metsästettiin sukupuuttoon 1800-luvulla (1868). Takaisinistutettu
Matemaatikot ja tilastotieteilijät
 Matemaatikot ja tilastotieteilijät Matematiikka/tilastotiede ammattina Tilastotiede on matematiikan osa-alue, lähinnä todennäköisyyslaskentaa, mutta se on myös itsenäinen tieteenala. Tilastotieteen tutkijat
Matemaatikot ja tilastotieteilijät Matematiikka/tilastotiede ammattina Tilastotiede on matematiikan osa-alue, lähinnä todennäköisyyslaskentaa, mutta se on myös itsenäinen tieteenala. Tilastotieteen tutkijat
Methods S1. Sequences relevant to the constructed strains, Related to Figures 1-6.
 Methods S1. Sequences relevant to the constructed strains, Related to Figures 1-6. A. Promoter Sequences Gal4 binding sites are highlighted in the color referenced in Figure 1A when possible. Site 1: red,
Methods S1. Sequences relevant to the constructed strains, Related to Figures 1-6. A. Promoter Sequences Gal4 binding sites are highlighted in the color referenced in Figure 1A when possible. Site 1: red,
r = 0.221 n = 121 Tilastollista testausta varten määritetään aluksi hypoteesit.
 A. r = 0. n = Tilastollista testausta varten määritetään aluksi hypoteesit. H 0 : Korrelaatiokerroin on nolla. H : Korrelaatiokerroin on nollasta poikkeava. Tarkastetaan oletukset: - Kirjoittavat väittävät
A. r = 0. n = Tilastollista testausta varten määritetään aluksi hypoteesit. H 0 : Korrelaatiokerroin on nolla. H : Korrelaatiokerroin on nollasta poikkeava. Tarkastetaan oletukset: - Kirjoittavat väittävät
Geenitutkimusta: evoluutiosta kohti geenivarojen suojelua ja jalostusta
 Geenitutkimusta: evoluutiosta kohti geenivarojen suojelua ja jalostusta 19.10.2016 Metsätieteen päivä Outi Savolainen, Oulun yliopisto, Genetiikan ja fysiologian tutkimusyksikkö Käyttölisenssi: CC BY 4.0
Geenitutkimusta: evoluutiosta kohti geenivarojen suojelua ja jalostusta 19.10.2016 Metsätieteen päivä Outi Savolainen, Oulun yliopisto, Genetiikan ja fysiologian tutkimusyksikkö Käyttölisenssi: CC BY 4.0
Miltä näyttää kotimaisten rotujemme perinnöllinen monimuotoisuus?
 1 / 6 Miltä näyttää kotimaisten rotujemme perinnöllinen monimuotoisuus? Katariina Mäki ja Mauri Kumpulainen Kennelliitolla on meneillään tilaustutkimus kotimaisten rotujen perinnöllisestä monimuotoisuudesta.
1 / 6 Miltä näyttää kotimaisten rotujemme perinnöllinen monimuotoisuus? Katariina Mäki ja Mauri Kumpulainen Kennelliitolla on meneillään tilaustutkimus kotimaisten rotujen perinnöllisestä monimuotoisuudesta.
Merkkausvärin kehittäminen
 Merkkausvärin kehittäminen Heikki Juhe, 26.1.2011 1. Johdanto JL-tuotteet aloitti keväällä 2010 tutkimus- ja kehitysprojektin, jonka tarkoituksena oli tutkia käytössä olevien merkkausvärien imeytyvyyttä
Merkkausvärin kehittäminen Heikki Juhe, 26.1.2011 1. Johdanto JL-tuotteet aloitti keväällä 2010 tutkimus- ja kehitysprojektin, jonka tarkoituksena oli tutkia käytössä olevien merkkausvärien imeytyvyyttä
VILLISIKAKANNAN HALLINTA/METSÄSTYS
 VILLISIKAKANNAN HALLINTA/METSÄSTYS VILLISIKA RIISTALAJINA Maitse liikkuva riistalaji kohti vettä ja ravintoa Lisääntymispotentiaali laumakäyttäytyminen Villisika tunnista poliittisia rajoja ei viisumia
VILLISIKAKANNAN HALLINTA/METSÄSTYS VILLISIKA RIISTALAJINA Maitse liikkuva riistalaji kohti vettä ja ravintoa Lisääntymispotentiaali laumakäyttäytyminen Villisika tunnista poliittisia rajoja ei viisumia
Suomalaisen maatiaiskanan säilytysohjelman koulutuspäivä, Riihimäki, 25.10.2014 Pasi Hellstén
 Suomalaisen maatiaiskanan säilytysohjelman koulutuspäivä, Riihimäki, 25.10.2014 Pasi Hellstén Sisäsiittoisuudella tarkoitetaan perinnöllisyystieteessä lisääntymistä, jossa pariutuvat yksilöt ovat enemmän
Suomalaisen maatiaiskanan säilytysohjelman koulutuspäivä, Riihimäki, 25.10.2014 Pasi Hellstén Sisäsiittoisuudella tarkoitetaan perinnöllisyystieteessä lisääntymistä, jossa pariutuvat yksilöt ovat enemmän
Kanalintupoikueet metsämaisemassa; Metla-RKTL yhteistyötutkimus. Esa Huhta Pekka Helle Ari Nikula & Vesa Nivala
 Kanalintupoikueet metsämaisemassa; Metla-RKTL yhteistyötutkimus Esa Huhta Pekka Helle Ari Nikula & Vesa Nivala Taustaa Maisemaekologinen tutkimus metsähabitaattien ja maisemarakenteen vaikutuksista metsäkanalintupoikueiden
Kanalintupoikueet metsämaisemassa; Metla-RKTL yhteistyötutkimus Esa Huhta Pekka Helle Ari Nikula & Vesa Nivala Taustaa Maisemaekologinen tutkimus metsähabitaattien ja maisemarakenteen vaikutuksista metsäkanalintupoikueiden
raakku - eläinkuntamme kiehtova ikänestori Panu Oulasvirta Metsähallitus
 raakku - eläinkuntamme kiehtova ikänestori Panu Oulasvirta Metsähallitus Raakkuseminaari Pudasjärvellä 19.11.2011 Makean veden suursimpukka Suomessa esiintyy 7 eri lajia makean veden suursimpukoita Raakku
raakku - eläinkuntamme kiehtova ikänestori Panu Oulasvirta Metsähallitus Raakkuseminaari Pudasjärvellä 19.11.2011 Makean veden suursimpukka Suomessa esiintyy 7 eri lajia makean veden suursimpukoita Raakku
Elintarvikepetokset Annikki Welling Kemian ja toksikologian tutkimusyksikkö Evira
 Elintarvikepetokset Annikki Welling Kemian ja toksikologian tutkimusyksikkö Evira Elintarvikepetokset EU:ssa ei ole yleisesti hyväksyttyä elintarvikepetosten määritelmää. Yleinen ohjeistus löytyy elintarvikelainsäädäntöä
Elintarvikepetokset Annikki Welling Kemian ja toksikologian tutkimusyksikkö Evira Elintarvikepetokset EU:ssa ei ole yleisesti hyväksyttyä elintarvikepetosten määritelmää. Yleinen ohjeistus löytyy elintarvikelainsäädäntöä
Metsägenetiikan sovellukset: Metsägenetiikan haasteet: geenit, geenivarat ja metsänjalostus
 Katri Kärkkäinen Matti Haapanen Metsägenetiikan sovellukset: Metsägenetiikan haasteet: geenit, geenivarat ja metsänjalostus Katri Kärkkäinen ja Matti Haapanen Metsäntutkimuslaitos Vantaan tutkimuskeskus
Katri Kärkkäinen Matti Haapanen Metsägenetiikan sovellukset: Metsägenetiikan haasteet: geenit, geenivarat ja metsänjalostus Katri Kärkkäinen ja Matti Haapanen Metsäntutkimuslaitos Vantaan tutkimuskeskus
Otoskoko 107 kpl. a) 27 b) 2654
 1. Tietyllä koneella valmistettavien tiivisterenkaiden halkaisijan keskihajonnan tiedetään olevan 0.04 tuumaa. Kyseisellä koneella valmistettujen 100 renkaan halkaisijoiden keskiarvo oli 0.60 tuumaa. Määrää
1. Tietyllä koneella valmistettavien tiivisterenkaiden halkaisijan keskihajonnan tiedetään olevan 0.04 tuumaa. Kyseisellä koneella valmistettujen 100 renkaan halkaisijoiden keskiarvo oli 0.60 tuumaa. Määrää
Näsijärven muikkututkimus
 1 Näsijärven muikkututkimus Markku Nieminen iktyonomi 1.1.1 NÄSIJÄRVEN KALASTUSALUEEN TUTKIMUKSIA MUIKKUTUTKIMUS VUONNA 1 1. Johdanto Näsijärven muikkukannan tilaa on seurattu kalastusalueen saaliskirjanpitäjien
1 Näsijärven muikkututkimus Markku Nieminen iktyonomi 1.1.1 NÄSIJÄRVEN KALASTUSALUEEN TUTKIMUKSIA MUIKKUTUTKIMUS VUONNA 1 1. Johdanto Näsijärven muikkukannan tilaa on seurattu kalastusalueen saaliskirjanpitäjien
EKOLOGISET YHTEYDET ALUEELLISEN YMPÄRISTÖHALLINNON
 EKOLOGISET YHTEYDET ALUEELLISEN YMPÄRISTÖHALLINNON TARPEET JA ROOLI 27.3.2013 SYKE Noora Raasakka ELINYMPÄRISTÖJEN PIRSTALOITUMINEN SUURIN UHKA EKOLOGISISTEN YHTEYKSIEN SÄILYMISELLE Alueiden käytön ja
EKOLOGISET YHTEYDET ALUEELLISEN YMPÄRISTÖHALLINNON TARPEET JA ROOLI 27.3.2013 SYKE Noora Raasakka ELINYMPÄRISTÖJEN PIRSTALOITUMINEN SUURIN UHKA EKOLOGISISTEN YHTEYKSIEN SÄILYMISELLE Alueiden käytön ja
Biodiversiteetti. Biodiversiteetin tasot
 Biodiversiteetti Johdatus biologian opetukseen-opetusmateriaali Ronja Hyppölä, Petteri Saarela, Matias Järvinen Biodiversiteetin tasot Biodiversiteetti eli luonnon monimuotoisuus tarkoittaa elollisen luonnon
Biodiversiteetti Johdatus biologian opetukseen-opetusmateriaali Ronja Hyppölä, Petteri Saarela, Matias Järvinen Biodiversiteetin tasot Biodiversiteetti eli luonnon monimuotoisuus tarkoittaa elollisen luonnon
Harjoitus 7: NCSS - Tilastollinen analyysi
 Harjoitus 7: NCSS - Tilastollinen analyysi Mat-2.2107 Sovelletun matematiikan tietokonetyöt Syksy 2006 Mat-2.2107 Sovelletun matematiikan tietokonetyöt 1 Harjoituksen aiheita Tilastollinen testaus Testaukseen
Harjoitus 7: NCSS - Tilastollinen analyysi Mat-2.2107 Sovelletun matematiikan tietokonetyöt Syksy 2006 Mat-2.2107 Sovelletun matematiikan tietokonetyöt 1 Harjoituksen aiheita Tilastollinen testaus Testaukseen
YLÄ-LAPIN AIKAISTETTU HIRVIJAHTI - tuloksia vuosilta 2010 ja 2011
 YLÄ-LAPIN AIKAISTETTU HIRVIJAHTI - tuloksia vuosilta 2 ja 211 Tuire Nygrén, Maija Wallén ja Riitta Tykkyläinen RIISTA- JA KALATALOUDEN TUTKIMUSLAITOS Utsjoki, Inari, Enontekiö ja Muonio 27. 3.8.212 Tästä
YLÄ-LAPIN AIKAISTETTU HIRVIJAHTI - tuloksia vuosilta 2 ja 211 Tuire Nygrén, Maija Wallén ja Riitta Tykkyläinen RIISTA- JA KALATALOUDEN TUTKIMUSLAITOS Utsjoki, Inari, Enontekiö ja Muonio 27. 3.8.212 Tästä
HISTORIA 5: RYHMÄTEHTÄVÄT SUOMEN ESIHISTORIASTA
 HISTORIA 5: RYHMÄTEHTÄVÄT SUOMEN ESIHISTORIASTA 1. Suomusjärven kulttuuri PEPPI, JANNA, LOVIISA, MINNA 2. Kampakeraaminen kulttuuri JONNA, SALLA, ESSI, JUHANI 3. Vasarakirveskulttuuri (nuorakeraaminen
HISTORIA 5: RYHMÄTEHTÄVÄT SUOMEN ESIHISTORIASTA 1. Suomusjärven kulttuuri PEPPI, JANNA, LOVIISA, MINNA 2. Kampakeraaminen kulttuuri JONNA, SALLA, ESSI, JUHANI 3. Vasarakirveskulttuuri (nuorakeraaminen
Huhtasuon keskustan liito-oravaselvitys
 Huhtasuon keskustan liito-oravaselvitys Taru Heikkinen 19.12.2008 Kaupunkisuunnitteluosasto Jyväskylän kaupunki 1. Tehtävän kuvaus ja tutkimusmenetelmät Työn tarkoituksena oli selvittää liito-oravan esiintyminen
Huhtasuon keskustan liito-oravaselvitys Taru Heikkinen 19.12.2008 Kaupunkisuunnitteluosasto Jyväskylän kaupunki 1. Tehtävän kuvaus ja tutkimusmenetelmät Työn tarkoituksena oli selvittää liito-oravan esiintyminen
Bioteknologian perustyökaluja
 Bioteknologian perustyökaluja DNAn ja RNAn eristäminen helppoa. Puhdistaminen työlästä (DNA pestään lukuisilla liuottimilla). Myös lähetti-rnat voidaan eristää ja muuntaa virusten käänteiskopioijaentsyymin
Bioteknologian perustyökaluja DNAn ja RNAn eristäminen helppoa. Puhdistaminen työlästä (DNA pestään lukuisilla liuottimilla). Myös lähetti-rnat voidaan eristää ja muuntaa virusten käänteiskopioijaentsyymin
ASPIRIININ MÄÄRÄN MITTAUS VALOKUVAAMALLA
 ASPIRIININ MÄÄRÄN MITTAUS VALOKUVAAMALLA Jaakko Lohenoja 2009 Johdanto Asetyylisalisyylihapon määrä voidaan mitata spektrofotometrisesti hydrolysoimalla asetyylisalisyylihappo salisyylihapoksi ja muodostamalla
ASPIRIININ MÄÄRÄN MITTAUS VALOKUVAAMALLA Jaakko Lohenoja 2009 Johdanto Asetyylisalisyylihapon määrä voidaan mitata spektrofotometrisesti hydrolysoimalla asetyylisalisyylihappo salisyylihapoksi ja muodostamalla
Skenaariot suurpetokantojen verotuksen suunnittelussa
 Skenaariot suurpetokantojen verotuksen suunnittelussa Katja Holmala Riistapäivät 19.1.2016 Esityksen rakenne Tausta Mallit ilveksen populaatiokehityksestä Malli 1: populaatiomalli Malli 2: skenaario- eli
Skenaariot suurpetokantojen verotuksen suunnittelussa Katja Holmala Riistapäivät 19.1.2016 Esityksen rakenne Tausta Mallit ilveksen populaatiokehityksestä Malli 1: populaatiomalli Malli 2: skenaario- eli
Suomenhevosten kasvuhäiriötutkimus Susanna Back
 Suomenhevosten kasvuhäiriötutkimus Susanna Back 1 Tutkimuksen tavoite Kartoittaa suomenhevospopulaatiossa osteokondroosin (OD) esiintyvyyttä ja periytyvyyttä (heritabiliteetti) Osa suomenhevosten jalostusohjesäännön
Suomenhevosten kasvuhäiriötutkimus Susanna Back 1 Tutkimuksen tavoite Kartoittaa suomenhevospopulaatiossa osteokondroosin (OD) esiintyvyyttä ja periytyvyyttä (heritabiliteetti) Osa suomenhevosten jalostusohjesäännön
Jokaisesta sairausgeenistä saa lisätietoa klikkaamalla kyseisen sairauden kohtaa ohjelmassa.
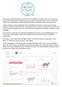 Genoscoopper julkaisi MyCatDNA-testin 13.06.2018. MyCatDNA-testi voidaan ottaa niin rodullisesta kuin roduttomasta kissasta ja kaiken ikäisistä kissoista. Testi tarkistaa jokaisesta kissasta yli 40 eri
Genoscoopper julkaisi MyCatDNA-testin 13.06.2018. MyCatDNA-testi voidaan ottaa niin rodullisesta kuin roduttomasta kissasta ja kaiken ikäisistä kissoista. Testi tarkistaa jokaisesta kissasta yli 40 eri
Panu Oulasvirta Alleco Oy
 Panu Oulasvirta Alleco Oy Raakku elää meillä pitkälti yli 100 vuotiaaksi Ruotsissa mitattu raakkuyksilön iäksi 280 vuotta! Kasvaa koko ikänsä Sukukypsyyden saavutettuaan lisääntymiskykyinen kuolemaansa
Panu Oulasvirta Alleco Oy Raakku elää meillä pitkälti yli 100 vuotiaaksi Ruotsissa mitattu raakkuyksilön iäksi 280 vuotta! Kasvaa koko ikänsä Sukukypsyyden saavutettuaan lisääntymiskykyinen kuolemaansa
Hirvieläinten verotussuunnittelmat 2017
 Hirvieläinten verotussuunnittelmat 2017 Tilannetieto 8.4.2017 10.4.2017 Suomen riistakeskus 1 10.4.2017 Suomen riistakeskus 2 Voimassa olevat hirvitavoitteet Tavoitteet on asetettu vuosille 2015-2017 PH1,
Hirvieläinten verotussuunnittelmat 2017 Tilannetieto 8.4.2017 10.4.2017 Suomen riistakeskus 1 10.4.2017 Suomen riistakeskus 2 Voimassa olevat hirvitavoitteet Tavoitteet on asetettu vuosille 2015-2017 PH1,
Säkylän Pyhäjärven kalataloudellinen kannattavuus tulevaisuudessa
 Säkylän Pyhäjärven kalataloudellinen kannattavuus tulevaisuudessa EMMI NIEMINEN, TOHTORIKOULUTETTAVA EMMI.E.NIEMINEN@HELSINKI.FI TALOUSTIETEEN LAITOS, MAATALOUS-METSÄTIETEELLINEN TIEDEKUNTA HELSINGIN YLIOPISTO
Säkylän Pyhäjärven kalataloudellinen kannattavuus tulevaisuudessa EMMI NIEMINEN, TOHTORIKOULUTETTAVA EMMI.E.NIEMINEN@HELSINKI.FI TALOUSTIETEEN LAITOS, MAATALOUS-METSÄTIETEELLINEN TIEDEKUNTA HELSINGIN YLIOPISTO
Kemiönsaaren Nordanån merikotkatarkkailu kesällä 2017
 Kemiönsaaren Nordanån merikotkatarkkailu kesällä 2017 Tmi Vespertilio 11.8.2017 Tiivistelmä Kemiönsaaren Nordanå-Lövbölen alueelle suunnitellaan tuulivoimapuistoa. Varsinais-Suomen ELYkeskus on vuonna
Kemiönsaaren Nordanån merikotkatarkkailu kesällä 2017 Tmi Vespertilio 11.8.2017 Tiivistelmä Kemiönsaaren Nordanå-Lövbölen alueelle suunnitellaan tuulivoimapuistoa. Varsinais-Suomen ELYkeskus on vuonna
KONEen yhtiökokous helmikuuta 2015 Henrik Ehrnrooth, toimitusjohtaja
 KONEen yhtiökokous 2015 23. helmikuuta 2015 Henrik Ehrnrooth, toimitusjohtaja 2014: Kannattava kasvu jatkui 2014 2013 Historiallinen muutos Vertailukelpoinen muutos Saadut tilaukset Me 6 812,6 6 151,0
KONEen yhtiökokous 2015 23. helmikuuta 2015 Henrik Ehrnrooth, toimitusjohtaja 2014: Kannattava kasvu jatkui 2014 2013 Historiallinen muutos Vertailukelpoinen muutos Saadut tilaukset Me 6 812,6 6 151,0
Geenikartoitusmenetelmät. Kytkentäanalyysin teoriaa. Suurimman uskottavuuden menetelmä ML (maximum likelihood) Uskottavuusfunktio: koko aineisto
 Kytkentäanalyysin teoriaa Pyritään selvittämään tiettyyn ominaisuuteen vaikuttavien eenien paikka enomissa Perustavoite: löytää markkerilokus jonka alleelit ja tutkittava ominaisuus (esim. sairaus) periytyvät
Kytkentäanalyysin teoriaa Pyritään selvittämään tiettyyn ominaisuuteen vaikuttavien eenien paikka enomissa Perustavoite: löytää markkerilokus jonka alleelit ja tutkittava ominaisuus (esim. sairaus) periytyvät
806109P TILASTOTIETEEN PERUSMENETELMÄT I Hanna Heikkinen Esimerkkejä estimoinnista ja merkitsevyystestauksesta, syksy (1 α) = 99 1 α = 0.
 806109P TILASTOTIETEEN PERUSMENETELMÄT I Hanna Heikkinen Esimerkkejä estimoinnista ja merkitsevyystestauksesta, syksy 2012 1. Olkoon (X 1,X 2,...,X 25 ) satunnaisotos normaalijakaumasta N(µ,3 2 ) eli µ
806109P TILASTOTIETEEN PERUSMENETELMÄT I Hanna Heikkinen Esimerkkejä estimoinnista ja merkitsevyystestauksesta, syksy 2012 1. Olkoon (X 1,X 2,...,X 25 ) satunnaisotos normaalijakaumasta N(µ,3 2 ) eli µ
Tilastollinen testaaminen tai Tilastollinen päättely. Geneettinen analyysi
 Tilastollinen testaaminen tai Tilastollinen päättely Geneettinen analyysi Tilastollisen testaamisen tarkoitus Tilastollisten testien avulla voidaan tutkia otantapopulaatiota (perusjoukkoa) koskevien väittämien
Tilastollinen testaaminen tai Tilastollinen päättely Geneettinen analyysi Tilastollisen testaamisen tarkoitus Tilastollisten testien avulla voidaan tutkia otantapopulaatiota (perusjoukkoa) koskevien väittämien
Muikkukannat ja ilmastonmuutos Hannu Lehtonen Helsingin yliopisto
 Muikkukannat ja ilmastonmuutos Hannu Lehtonen Helsingin yliopisto 100 vuotta suomalaista muikkututkimustaseminaari Jyväskylä 2.12.2008 LÄMPÖTILA SADANTA Erotus (%) vuosien 1961-1990 keskiarvosta Erotus
Muikkukannat ja ilmastonmuutos Hannu Lehtonen Helsingin yliopisto 100 vuotta suomalaista muikkututkimustaseminaari Jyväskylä 2.12.2008 LÄMPÖTILA SADANTA Erotus (%) vuosien 1961-1990 keskiarvosta Erotus
Tarkastelen suomalaisen taloustieteen tutkimuksen tilaa erilaisten julkaisutietokantojen avulla. Käytän myös kerättyjä tietoja yliopistojen
 1 2 3 Tarkastelen suomalaisen taloustieteen tutkimuksen tilaa erilaisten julkaisutietokantojen avulla. Käytän myös kerättyjä tietoja yliopistojen opettajien tutkimusalueista. 4 Kuviossa 1 esitetään kansantaloustieteen
1 2 3 Tarkastelen suomalaisen taloustieteen tutkimuksen tilaa erilaisten julkaisutietokantojen avulla. Käytän myös kerättyjä tietoja yliopistojen opettajien tutkimusalueista. 4 Kuviossa 1 esitetään kansantaloustieteen
TIIVISTELMÄ. Työstä eläkkeelle tulokehitys ja korvaussuhteet. Eläketurvakeskuksen raportteja 2010:3. Juha Rantala ja Ilpo Suoniemi
 R RAPORTTEJA Eläketurvakeskuksen raportteja 2010:3 TIIVISTELMÄ Juha Rantala ja Ilpo Suoniemi Työstä eläkkeelle tulokehitys ja korvaussuhteet Tutkimuksessa arvioitiin, mitä muutoksia henkilön tuloissa ja
R RAPORTTEJA Eläketurvakeskuksen raportteja 2010:3 TIIVISTELMÄ Juha Rantala ja Ilpo Suoniemi Työstä eläkkeelle tulokehitys ja korvaussuhteet Tutkimuksessa arvioitiin, mitä muutoksia henkilön tuloissa ja
