Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratory infection
|
|
|
- Johannes Laine
- 4 vuotta sitten
- Katselukertoja:
Transkriptio
1 SUPPLEMENTARY INFORMATION Articles In the format provided by the authors and unedited. Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratory infection Themoula Charalampous 1,8, Gemma L. Kay 1,2,8, Hollian Richardson 1,8, Alp Aydin 2, Rossella Baldan 1,3, Christopher Jeanes 4, Duncan Rae 4, Sara Grundy 4, Daniel J. Turner 5, John Wain 1,2, Richard M. Leggett 6, David M. Livermore 1,7 and Justin O Grady 1,2 * 1 Bob Champion Research and Educational Building, University of East Anglia, Norwich Research Park, Norwich, UK. 2 Quadram Institute Bioscience, Norwich Research Park, Norwich, UK. 3 CIDR, King s College London, St Thomas Hospital, London, UK. 4 Microbiology Department, Norwich and Norfolk University Hospital, Norwich, UK. 5 Oxford Nanopore Technologies, Gosling Building, Oxford Science Park, Oxford, UK. 6 Earlham Institute, Norwich Research Park, Norwich, UK. 7 AMRHAI, Public Health England, London, UK. 8 These authors contributed equally: Themoula Charalampous, Gemma L. Kay, Hollian Richardson. * justin.ogrady@quadram.ac.uk Nature Biotechnology
2 Supplementary Information (A) TTGTTGCTTCGTTCAGTCGGGTGGGTGTTTATGGGACCGTAAGCCTACCGTGACGTTACCAGTAGGAATGAATCCTTCGTT TTTCGTGCGCCGCTTCACTCTATAACTTGTTCCACAGCCATTTTTTGAAGATCTGGTGGACCATATTTCTTTAATAGGCGTCT AACTTGAACTTCTCATTTTAGCTTTAGCACTGTCTCGTTTGAACCAGTCAACACTCATGTTATCATAACTGTTTTTAGTTAACT CATGTGCAATGGCTCTTAACTCTTTATCGCCAAAGCTTCTTTAGCTGTTCATGTGAAGCTCGATACTACATACAATATACGC AACAATGCGCTTGTTTCGATTAGGTTTAGGCATTGAATCATAAAGTGCAATATATACTTAACACGTTCTTTAATATTTGAATTA AGACCTAGTAAATATTGTGCCATTATTTATGTGCTGAGATTTTAAAATAGAACATTTTAATATCCCGATTCTACGCAGTATAT GTTCATGGCGATTCAAAATTTTTTAAAACGTTTCTATCACATACCTTTTCACAGTCATTGTCCATCATTGTTTTTACTTATATAT AGTGCAGGTTAAACCAGAATATCATTTTAAAAACAACATATAAAGCTGGTTGAATATTAAGTCATGACTTTTCGCATGTGATA GTTCATGTAGAATAATATATTCAATTTCTTTGTCATTCATGGTTTCATTACGACAGTTGGTATTGGTACAATTGGGATTTCACT AAACCAAATACCATCGGATTATCAATGTTTTGAACTATAACTAATTGTTATATGCTTTTTTGTGCGTTACATCTTACTTTGACA TACTTTAAGTCGTTCATTAAGATATGACGATTCCAATGACAGACTTTAATAACATCCAATTTGTCGGAATGCCTTAATCATAT AAAAATAAGCACAACAAACTACCGAATACCATATCAAAAGAATCATATACGTTATATTTGAGGTCTCGGCAGTTAACATTAAT TTTCAAGTCTTTCGTAACAGATGATTGTTGACCATCTAACATATGACTAACCAGAAAGAAGTCGTGTCAGATACATTTCGATT CATCATATCTTTGAAAATGGAATTCGATATTTTGTAAAATGGGTATTAATGGAATTAACGTGGAGACGAGCACCTAATAACCA AATCTTATGTGACATAATATTTTGAGTATATTTTATATAGAACATTCTCACTAAAAAATTACATATCGTGAGCAATGAACTGAT TATACTTAACATTAAAAAGGTATTAACATATTCTACACCTCCATATCACAAAATTGTAACATTATTTTGACATAAATACTACATT TGTAATATACTACAAATGTGGTCTTATATAAGAGTATATTGATGAAAAGAAGATAAAAATTGTTCACACTTATTTTTAATAGT TGTAGTTGTCGGGTTTGGTATATATTTTATGCTTCAAAGATAAAGAAATTAATAATACTATTGATGCAATTGAAGATAAAA TTTCAAACAAGTTTATAAGATAACAGTTATATTTCTAAAAGCGATAATGGTGAAGTAGAAATAATGACTGAACGTCCGAT AAAAATATATATAATAGTTAGGCGTTAAAGATATAAACATTCAGGATCCGTAAAATAAAAAAGTATCTAAAATAAAAACA GTAGATGCTCAATATAAAATTAAAACAAACTGGTAGCATTGATCCGCAGCGTTCCGTTTAATTTTGTTAAAAGATGGTAT GTGGAAGTTAGGTGGGATCATAGCGTCATTATTCAGAATGCAGAAAGACCAAAGCATACATATTGAAAATTTAAAATCA GAACGTGGTAAAATTTTAGACCGAAACAATGTGGAATTGGCCAATACAGGAACAGCATATGAGATAGGCATCGTTCCAA AGAATGTATCTAAAAGATTATAAAACAATCGCTAAAGAACTAAGTATTTCTGAAGACTATATCAAACCAACAAATGGATC AAAATTGGGTACAAAGATGATACCTTCGTTCTTTTAAAACCGTTAAAAATGGATGAATATTTAAGTGATTTCGCAAAATTT CATCTTACAACTAATGAAACAGAAAGTCGTAACTATCCTCTAGAAAAACGACTTCACATCTATTAGGTTATGTTGGTCCC ATTAACTCTGAAGAATTAAAACAAAAGAATATAAAGGCTATAAAGATGATGCAGTTATTGGTAAAAAGGGACTCGAAAA ACTTTACGATAAAAAACCAACATGAAGATGGCTATCGTGTCACAATCGTTGACGATAATAGCAATACAATCGCACATAC ATTAATAGAGAAAAAGAAAAAGATGGCAAAGATATTCAATAACTATTGATGCTGAAGTTCAAAAGTGACTATAACAACA TGAAAAATGATTATGGCTCAGGTACTGCTATCCACCCTCAAACAGGTGAATTATTATTAGCACTTGTAAGCACACCTTCA TATGACGTCTATCCATTTTATGTATAGCATGAGTAACGAAAAGAATATAATATAAATTAACCGAAGATGAAAGAAAAGAA CCTCTGCTCCAACAAGTTCCAGATTACAACTTCACCGAGTTCAACTCAAAAATATTAACAGCAATGATTGGGTTAAATAA CAAAACATTAGACGATAAAACAAGTTATAAAATCGATGGTAAGGTTGGCAAAAAGATAAATCTTGGGGTGGTTACAACG CGTTACAAGATATATGAAGTGGTAAATGGTAATATCGACTTAAAACAAGCAATAGAATCATCAGATAACATTTTCTTTGC TAGAGTAGCACTCAGATTAGGCAGTGAAATTTGAAAAGGCATGAAAAACTAGGTGTTGGTGAAGATATACCAAGTGATT ATCCATTTTATAATGCTCAGAATTTCAAACAAAATTTAGATAATGAAATATTGATAGCTGATTCAGGTTACGGACAAGGT GAAATACTGATTAACCCAGTACAGATCCTTTCAATCTATAGCGCATTAGAAAGAAATAATAGCAATATTAACGCACCTCA TGATAAAAGAACTTACGAAAAACAAAGTTTGGAAGAAAAATATTTCAATGAAGCGGCGCACGAAAACAGAGGATTCATTC CACGGTAACACGTCACC (B)
3 (C) ATGGTTCCGTGGTGTTTAACAGTCATGTAAATGACCTTGTCCAAGTTTGTAACCTTCGTTTTTCGTGCGCCGCTTCCAATCG CGTCCTTTGTTTACACGAATGTCTTTTGCTCTTAATATTAATTCCTGCTTCCTCATCTATAATCGCATGTGCTTTCATTTTGCT TTGTATGCGGGAAAACCCTGCGATTTTAGCAGGAAGAGTTGTAATTAAAATATAATATTCCTCCAATATTGTTTATTTTAAAT CCCTATTTTCTAACCTCAGTTATCATTTTGGCAAACTATAGCAATGTAATGAGTCTAAAATACCTGATAGTAAAACCAATG ATTTACACCGATTTTATCGGCAAAAGAATCCAAGAAGAATTATTAATTGTATGAAGTGACATGATACTTTAAATCAAGAA AATACACGTCCCTAAATATTCTAGACTTAATTTTCTCCTGAAAAAGAGCTGTTTACACCATATAAATATACCAGCCCAATG TGCACAACAAACTACGAATATTATAATCCCAGCTGGAGGAAGTATTCCGAAACGGCTAAACTGGTCCCCATAAAAAATG AACTTGTTATTGGTAATACATGCTTTTCAGAAGCCCCTAATCTTCAACCTAATAAGCCTCCTGCTGGCATCCCAAATACTG AGGAAATTTCCATGTAGCAAAGTAATATACGTTCCATTAAGAGTGTTCCATGCTTATTAAAGAAAATAATTGCATTGATTG GCATATAAACAAAGTATATAGTGTTCTAAAAGAGTATAAGGCAAACAAATCCCTTTGTTTTGTCTCCAGAACCACAACTC CTTCTTTCATCTCCCTTGTGAAATTTAGTTCAAACTTTGCATGATTACCCAGCTTGGTATACGGTACAATTTACCGTATAA ATCACAGTCTGGCACCCATACGTCGATGGCAATAATAGCATTTAAATCCCAAACGGAGGTATAAGAGTGCTGCAACTGC CGGACTATTTCATATAACATAGACTGCAGACTGACTATAGCTCGCATTTCGTTAATAATTTCTTCTGGTACTAAAATTGGT GTAACCGCATTGAGAGTGCTGGGGTGGCTGTTCAATGCTACGGATAACAATACTATCGCAAATCATCAGACAGGTAGCT CCCGTCAGCAACCGAAACACTGCACTGCTTCTGCGATATTAAATCGGCACCAATCATTCCTCTTCTTCCTATCGTGATCC ACTAACTTTTCAATGGCGGTCAAATCGCATGGGGTAAAAACCTACTAATGAAGCCATAGACAGGACAAAAATGCAGATC CTGTTTTTCTGTAGGTAAAAATAATCATAATTTACGGGATGGCACTGGTCATGATACTGCTTTGCCCACTTATGTGCATAG GTACTTCGTTTCCAATTGTTTGTATTTCCATTTATATTTATCTCACCATATTCTTTATGAATTTCTAATGGCCTGAATAGCATT CCAGGCAATAAAAATGCGAATGACCAAGTAAGCTTTTGGTCTTACCTTACGCTGTTAGGTATTCATATTAAAGACCTTGGAT TGAAATTAAAGGGTATGGTTAAATAACCAATGTCCTCACCGTAACTATGAATGCTCAATATCGTATAAATAAGCACAACAAAA AGCCTATCATCGGGTATAGATTCCTTTGTTGCCAGCCAAACGCATTAGAGGCTGTCATAGTTTCGATATCTATCTTTGTTTAT ATCAATTTATGAGTAACTTTAACAAGATGATATGTTCAATAAAGGATTATGGAATGGACTCATACTTTAATTCGATTAGTTTAA AGGATATGACAGAGTTTTCTTCATGAACCGTAACGCTCTAGTTATCTTCCTTAGTGAGCAAGCGATTAGCTTTCTAAAATCTT CTGTTTGTAGTTTAGAATTCATCAGGATTTTCTAACTCAACCTCAAATATTCATTTTACATTCCTATTTCATTTATTGATAAATT AGTTTGCAAAAAATTGGACAATTTTTGTCCTCTTAACCTTTAAATAAGTTTTTTTAATCGATTTAGGTTGCCTAAATTATTGCT TATTCGGTAAATGAAGTATTGCTTTCAACAGATTTCCTTCAACTACACTTCTTGATTCAAACAAGTTGTTTATTCTTCCACAAA CTCCTTGTCAATGGAATAAAACACATGCCACAGGTAAATGAATAGAACTGATGATTTCTAAACTATCGCTACCTCATTCCAG AATTTTCTCACTCTCTAGATGCCCACCCTGTAAAGGCAAAACTGCAATCAAACTCCATTATCCTTTCGTTCAAGGACCACAA GCTTTACAAACCGGGCGACCCACACTCAGCGGTTCGCTATCTCCGTTCTGTCACGCGTGCTAACTTGTCGTCACGGACGG CTATCGCATGGAGCGGAGTAAATGTAATCTTCGTCGTTTTGCTCCTTGACCGCCAAACTACCCGCCTCAGCATGCTAAATG CATCTCCTATATTAAAACGAAAGTAACTGAGTTGTTGTTTAAATTAAAATAGAATGAAACTATTTTATAGTTACCATTTTTCAA CACCAGCTGATATTCATCCGCCAAAAATTTACCCTTTAACTTACGGCAAACTTGTTGAGTCACATAGCACTTTTAACGTCCA ACTTCACCATTCTTCTTTAATCAACTCTTTTATTATTCAGCTTTTCTCAATCAAATGCCAACTAGGAATGGCCGTTGCTTTAAT ATCCATCGTACCATTCTCCATTCCCTGCGCTTGATTGCATTGCTGATATTTCTTCAGCTCCGTAGGAACGGGCCATGGAGC ACCAACTATGTTGTTGAAAGAATATAGTGAAGCGGCCGCACGAGAAAAACGAAGGTTACGCAAACCCTGGACAGGTGGG (D)
4 Supplementary Figure 1: The metagenomic pipeline with a turnaround time of approx. six hours (optimised) and approx. eight hours (pilot) from sample collection to sample result inferring antibiotic resistance gene/organism association using resistance gene flanking sequence. A: An example MinION read mapping to the meca gene (gene sequence in bold)* B: Identification of Staphylococcus aureus chromosome sequence using the meca gene 5 flanking sequence of the read*. C: An example of a MinION read mapping to the gene (gene sequence in bold)*. D: Identification of Streptococcus gordonii chromosome sequence using the gene 3 flanking sequence of the read*. *Three independent clinical samples were analysed (an example of a pathogen and a lung commensal are represented).
5 Supplementary Table 1a: Comparison of bacterial DNA extraction methods using qpcr. Sample Sample type Microbiology result O1 Sputum S. aureus O2 Sputum P. aeruginosa Condition 16S rrna gene V3-V4 fragment qpcr assay (Cq) Bacterial gain to standard depletion (DCq) Human qpcr assay (Cq) New DNA depletion compared to standard depletion (DCq) Standard depletion New depletion (enzyme cocktail) (3 fold) New depletion (bead beating) (21 fold) (2 fold) Standard depletion New depletion (enzyme cocktail) New depletion (bead beating)
6 Supplementary Table 1b: Comparison of the microbial community with altered PCR extension times for MinION sequencing. Sample Sample type Microbiology result PCR extension time (min) Average read length (Kb) Bacterial species from WIMP analysis ( 0.5% of classified reads) O1 Sputum S. aureus S. aureus Streptococcus agalactiae Streptococcus anginosus group Veillonella parvula S. aureus Streptococcus agalactiae Streptococcus anginosus group O2 Sputum P. aeruginosa P. aeruginosa group Pseudomonas P. stutzeri group P. aeruginosa group Pseudomonas Percentage of total classified reads (%)
7 Supplementary Table 2a: Limit of detection for Gram-positive and Gram-negative organisms using the optimised method in a high bacterial background. Sample Replicate Pathogen Approx. number of pathogen cells (CFU) DNA yield (ng/µl) Total raw read count Reads mapping to hg38 Nonhuman reads Classified from nonhuman reads Unclassified from non-human reads Total number of pathogen reads from total classified reads , (0.9%) 65,947 (99.1%) 50,922 15,015 12,658 (24.9%) SA , , (0.8%) 54,158 (99.2%) 42,612 11,524 4,286 (10.1%) , (0.6%) 36,064 (99.4%) 29,303 6,749 3,882 (13.2%) 1 S. aureus , (0.7%) 39,739 (99.3%) 29,311 10, ** (0.6%) SA , , (0.9%) 36,104 (99.1%) 27,233 8, (1.4%) , (0.9%) 44,428 (99.1%) 32,656 11, (2.1%) , (0.7%) 36,963 (99.3%) 28,559 8,398 1,346 (4.7%) EC , , (0.5%) 50,581 (99.5%) 38,924 11,637 2,030 (5.2%) , (0.5%) 44,959 (99.5%) 34,140 10,798 1,092 (3.2%) 1 E. coli , (0.4%) 27,916 (99.6%) 22,324 5,586 90** (0.4%) EC , , (0.7%) 44,469 (99.3%) 33,525 10, ** (0.5%) , (0.7%) 46,589 (99.3%) 34,319 12, ** (0.4%) *22Cq bacterial 16S rrna gene V3-V4 fragment qpcr assay **number of reads detected was below the ³1% of classified microbial reads and WIMP assignment q-score ³20 cut-off required for a sample to be considered positive.
8 Supplementary Table 2b: Limit of detection for Gram-positive and Gram-negative organisms using the optimised method in a low bacterial background*. Sample Replicate Pathogen Approx. number of pathogen cells (CFU) DNA yield (ng/µl) Total raw read count Reads mapping to hg38 Nonhuman reads Classified from nonhuman reads Unclassified from non-human reads Total number of pathogen reads from total classified reads** , (1.5%) 37,624 (98.5%) 36,271 1,343 23,514 (64.8%) SA , , (1.1%) 8,384 (98.9%) 8, ,928 (72.7%) , (4.1%) 13,719 (95.9%) 12,642 1,074 3,129 (24.8%) 1 S. aureus ,889 19,276 (31.1%) 42,613 (68.9%) 38,796 3,805 7,222 (18.6%) SA ,360 9,337 (29.8%) 22,023 (70.2%) 19,905 2,115 3,037 (15.3%) ,346 2,531 (10.0%) 22,815 (90.0%) 20,6802 2,120 2,499 (12.1%) , (3.0%) 28,443 (97.0%) 26,452 1,983 10,609 (40.1%) EC , , (2.5%) 7,139 (97.5%) 6, ,756 (41.5%) , (2.3%) 3,965 (97.7%) 3, ,072 (29.5%) 1 E. coli 0.8 2, (3.3%) 2,557 (96.7%) 2, (4.8%) EC , , (2.7%) 10,099 (97.3%) 8,736 1, (4.4%) , (3.6%) 14,473 (96.4%) 12,663 1, (4.4%) *27Cq bacterial 16S rrna gene V3-V4 fragment qpcr assay **The number of reads detected for all samples was above the 1% of classified reads and WIMP assignment q-score 20 required for a sample to be considered positive.
9 Supplementary Table 3: Mock community qpcr results in triplicate for spiked NRF samples with and without the optimised method. qpcr assay Sample Triplicate 1 (Cq) Triplicate 2 (Cq) Triplicate 3 (Cq) Average (Cq) Bacterial loss (DCq) Human Undepleted Depleted (10 3 fold) E. coli Undepleted Depleted H. influenzae Undepleted Depleted K. pneumoniae Undepleted Depleted P. aeruginosa Undepleted Depleted S. aureus Undepleted Depleted S. maltophilia Undepleted Depleted S. pneumoniae Undepleted Depleted (5.7 fold)
10 Supplementary Table 4: Sequencing metadata for all respiratory samples processed with the optimised method. Sample number Number of raw reads from 2hrs Number of reads minus hg38 Human reads Classified reads Unclassified reads Metagenomics output S E. coli S K. pneumoniae 1692 S P. aeruginosa S S. marcescens 3900 S K. oxytoca 3254 K. pneumoniae 906 S S. aureus Process extraction negative E. coli S. aureus S. pneumoniae S P. aeruginosa H. influenzae 232 S M. catarrhalis 7078 S. pneumoniae 504 S E. coli S H. influenzae S. pneumoniae 571 S S. pneumoniae 693 S M. catarrhalis H. influenzae Process extraction negative S. aureus H. influenzae E. coli S S. marcescens S S. aureus 9465 Number of pathogen reads ( 1% of classified reads and WIMP assignment q- score 20)
11 M. catarrhalis 8089 S S. aureus 6597 S. pneumoniae 390 S S. aureus S S16-undepleted S. aureus 6203 Process extraction negative 3 S H. influenzae S S H. influenzae 2827 S H. influenzae S. pneumoniae S S H. influenzae Process extraction negative S H. influenzae S H. influenzae S M. catarrhalis 3831 S H. influenzae S. pyogenes S. aureus S S. pneumoniae 464 S P. aeruginosa S. aureus Process extraction negative H. influenzae S. aureus E. coli S. pyogenes S1-undepleted E. coli
12 S P. aeruginosa S H. influenzae 1644 S E. coli S S Process extraction E. coli 10 negative 6 S E. coli S H. influenzae S P. aeruginosa S S. aureus P. aeruginosa S M. catarrhalis H. influenzae S S. aureus 6138 Process extraction negative E. coli H. influenzae M. catarrhalis P. aeruginosa S. aureus S S. aureus H. influenzae E. coli
13 Supplementary Table 5: Confirmatory qpcr analysis of additional pathogen detection. Discordant results compared to culture Sample qpcr gene target qpcr result (Cq) S Escherichia coli 1 S41 cyaa - S Haemophilus influenzae 1 S12 omp P6 - S S Klebsiella pneumoniae 1 S5 mdh - S Moraxella catarrhalis 1 S14 copb 22.0 S Pseudomonas aeruginosa* S9 oprl - Pseudomonas aeruginosa 1 S7 oprl 32.9 Staphylococcus aureus 1 S29 eap 32.7 S8 - S Streptococcus pneumoniae 1 S11 ply 32.2 S15 - S21 - S28 - Streptococcus pyogenes 1 S27 sdab Detected by metagenomics and not culture *Detected by culture but not metagenomics
14 Supplementary Table 6: Species-specific gene analysis for H. influenzae and S. pneumoniae Pathobionts detected by metagenomics pipeline Haemophilus influenzae Streptococcus pneumoniae Sample Pathobiont specific gene Number of reads aligned to pathobiont specific gene S7 5 S10 85 S12 0 S18 85 S20 67 S21 siat 49 S23 21 S24 61 S25 90 S27 63 S31 5 S36 60 S39 43 S41 2 S8 0 S10 5 S11 19 S15 ply 0 S21 0 S28 0
15 Supplementary Table 7: Microbiology antibiogram and ARMA output for all optimised method samples. Sample Microbiology Antibiogram culture result S1 E. coli Amoxicillin R, Gentamicin S, Co-amoxiclav R, Co-trimoxazole R, Tazocin I, Ciprofloxacin S, Meropenem S, Aztreonam S, Ceftazidime S, Ceftriaxone S, Cefuroxime S, Amikacin S, Ertapenem S, Tigecycline S, Tobramycin S, Cefepime S S2 K. pneumoniae Amoxicillin R, Gentamicin S, Co-amoxiclav R, Co-trimoxazole S, Tazocin I, Ciprofloxacin S, Meropenem S, Aztreonam S, Ceftazidime S, Ceftriaxone S, Cefuroxime S, Amikacin S, Ertapenem S, Tigecycline S, Tobramycin S, Cefepime S S3 P. aeruginosa Gentamicin S, Tazocin S, Ciprofloxacin S, Ceftazidime S, Meropenem S S4 S. marcescens Gentamicin S, Co-trimoxazole S, Ciprofloxacin S, Meropenem S, Aztreonam S, Ceftazidime S, Amikacin S, Tigecycline S, Tobramycin S, Levofloxacin S, Colistin R, Cefepime S, Minocycline S, Ticarcillin S S5 K. oxytoca Gentamicin S, Co-trimoxazole S, Tazocin I, Ciprofloxacin S, Meropenem S, Aztreonam S, Ceftazidime S, Amikacin S, Tigecycline S, Tobramycin S, Levofloxacin S, Colistin S, Cefepime S, Imipenem S, Minocycline S, Ticarcillin R S6 S. aureus Flucloxacillin S, Erythromycin/clarithromycin S, Clindamicin S, Fuscidic acid S, Tetracycline/doxycycline S, Mupirocin S S7 H. influenzae Amoxicillin R, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole S S8 M. catarrhalis Amoxicillin R, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole S S9 P. aeruginosa Gentamicin S, Tazocin S, Ciprofloxacin S, Meropenem S, Ceftazidime S E. coli Co-amoxiclav R, Co-trimoxazole R, Tazocin S, Ciprofloxacin S, Meropenem S, Aztreonam S, Ceftazidime S, Ceftriaxone S, Cefuroxime S, Amikacin S, Ertapenem S, Tigecycline S, Tobramycin R, Cefepime S ARMA Output sul1 mpha dfra17 aada5 ACT-5 oqxb oqxa InuA OXA-50 catb7 APH(3 )- llb AAC(6 )- lc oqxb OXY-4-1 oqxb APH(3')- Ia vgac tet38 teto IsaC ermc sul1 aada2 AAC(3 )- lla mpha dfra12 AAC(3 )- llc S10 NSG ND
16 teto S11 NRF ND S12 NRF ND ErmB S13 S. marcescens Gentamicin S, Co-amoxiclav R, Co-trimoxazole S, Ciprofloxacin S, Meropenem S, Aztreonam S, Ceftazidime S, Ceftriaxone S, Cefuroxime R, Amikacin S, Ertapenem S, Tigecycline I, Tobramycin S, Cefepime S S14 S. aureus Flucloxacillin S, Erythromycin/clarithromycin S, Clindamicin S, Fuscidic acid S, Tetracycline/doxycycline S, Mupirocin S S15 S. aureus Flucloxacillin S, Erythromycin/clarithromycin S, Clindamicin S, Fuscidic acid S, Tetracycline/doxycycline S, Mupirocin S S16 S. aureus/mrsa Penicillin R, Flucloxacillin R, Oxacillin R, Erythromycin S, Clindamycin S, Trimethoprim R, Gentamicin R, Ciprofloxacin R, Fusidic acid R, Mupirocin S, Rifampicin S, Vancomycin S, Teicoplanin S, Tigecycline S, Linezolid S AAC(6 )- lc oqxb tet38 tetq tet38 tetw tet38 meca ErmB S17 NRF ND tetq ErmF CfxA3 S18 H. influenzae Amoxicillin R, Tetracycline/doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Co-trimoxazole S TEM-70 TEM-33 TEM-105 TEM-104 TEM-208 S19 NRF ND tetw ErmX
17 S20 H. influenzae Amoxicillin S, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole S teto S21 NRF ND tet32 S22 NRF ND tetq S23 H. influenzae Amoxicillin S, Tetracycline/doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Co-trimoxazole S S24 H. influenzae Amoxicillin S, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole S S25 H. influenzae Amoxicillin S, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole S S26 M. catarrhalis Amoxicillin R, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole S S27 H. influenzae Amoxicillin S, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole S teto tetd tet32 tetw teta teto ermt tet38 S. aureus Flucloxacillin S, Erythromycin R, Clindamycin R, Fuscidic acid S, Tetracycline S, Mupirocin S S28 NRF ND lsac S29 P. aeruginosa Gentamicin S, Tazocin S, Ciprofloxacin S, Meropenem S, Ceftazidime S S30 P. aeruginosa Gentamicin S, Tazocin S, Ciprofloxacin S, Meropenem S, Ceftazidime S OXA-50 catb7 tet38 OXA-50 catb7 S31 NRF ND S32 NSG ND
18 AAC(3)- lla AAC(3)- llc vgac TEM-1 S33 NRF ND S34 NSG ND lsaa ErmB S35 E. coli Amoxicillin R, Gentamicin S, Co-amoxiclav R, Co-trimoxazole S, Tazocin R, Ciprofloxacin S, Meropenem S, Aztreonam S, Ceftazidime I, Ceftriaxone S, Cefuroxime S, Amikacin S, Ertapenem S, Tobramycin S S36 H. influenzae Amoxicillin R, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole R S37 P. aeruginosa Gentamicin S, Tazocin R, Ciprofloxacin S, Meropenem S, Ceftazidime R S38 P. aeruginosa Gentamicin S, Tazocin S, Ciprofloxacin S, Meropenem S, Ceftazidime S TEM-11 TEM-2 TEM-187 ACT-5 TEM-67 ErmB Mel catb7 OXA-50 tet38 S. aureus Flucloxacillin S, Erythromycin S, Clindamycin S, Fuscidic acid S, Tetracycline S, Mupirocin S S39 H. influenzae Amoxicillin S, Doxycycline S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole R S40 S. aureus (MRSA) Penicillin R, Flucloxacillin R, Oxacillin R, Erythromycin S, Doxycycline S, Clindamycin S, Trimethoprim S, Gentamicin S, Ciprofloxacin R, Fuscidic acid S, Rifampicin S, Vancomycin S, Teicoplanin S, Tigecycline S, Linezolid S, Daptomycin S, Chloramphenicol S S41 S. aureus Flucloxacillin S, Erythromycin S, Clindamycin S, Fuscidic acid S, Tetracycline S, Mupirocin S meca tet38
19 H. influenzae Tetracycline S, Amoxicillin S, Ceftriaxone S, Co-amoxiclav S, Ciprofloxacin S, Cotrimoxazole R R = resistant S = sensitive
20 Supplementary Table 8: All microorganisms identified in all samples tested using the optimised method (above our thresholds). Sample Organism cultured by microbiology Organisms identified from metagenomic pipeline ( 1% of classified reads and WIMP assignment q-score 20) S1 Escherichia coli Escherichia coli S2 Klebsiella pneumoniae Klebsiella pneumoniae S3 Pseudomonas aeruginosa Pseudomonas aeruginosa S4 Serratia marcescens Serratia marcescens Pantoea stewartii Citrobacter freundii Salmonella enterica Serratia plymuthica S5 Klebsiella oxytoca Klebsiella oxytoca Citrobacter freundii Klebsiella sp. M5al Klebsiella pneumoniae Klebsiella michiganensis S6 Staphylococcus aureus Staphylococcus aureus S7 Haemophilus influenzae Pseudomonas aeruginosa Veillonella parvula Rothia mucilaginosa Streptococcus sanguinis Haemophilus influenzae Haemophilus parainfluenzae Neisseria sicca S8 Moraxella catarrhalis Moraxella catarrhalis Streptococcus mitis Veillonella parvula Streptococcus salivarius Rothia mucilaginosa Streptococcus pseudopneumoniae Streptococcus pneumoniae Neisseria sicca Streptococcus gordonii S9 Pseudomonas aeruginosa & Escherichia coli Escherichia coli Lactobacillus paracasei Lactobacillus casei Candida albicans S10 NSG Haemophilus influenzae Streptococcus pseudopneumoniae Streptococcus sp. oral taxon 431
21 Streptococcus pneumoniae Streptococcus mitis S11 NRF Streptococcus sp. A12 Streptococcus mitis Rothia mucilaginosa Bifidobacterium longum Streptococcus pseudopneumoniae Streptococcus pneumoniae Streptococcus sp. I-P16 Streptococcus sp. I-G2 Haemophilus parainfluenzae S12 NRF Haemophilus parainfluenzae Moraxella catarrhalis Streptococcus gordonii Neisseria sicca Haemophilus influenzae S13 Serratia marcescens Serratia marcescens S14 Staphylococcus aureus Staphylococcus aureus Moraxella catarrhalis Rothia mucilaginosa Streptococcus constellatus Fusobacterium nucleatum Fusobacterium periodonticum Streptococcus anginosus Streptococcus intermedius S15 Staphylococcus aureus Staphylococcus aureus Streptococcus mitis Citrobacter koseri Rothia mucilaginosa Streptococcus salivarius Haemophilus parainfluenzae Streptococcus pseudopneumoniae Streptococcus pneumoniae Prevotella aninogenica S16 MRSA Staphylococcus aureus Lactobacillus rhamnosus S17 NRF Streptococcus salivarius Prevotella aninogenica Streptococcus sp. oral taxon 431 Streptococcus sp. A12 Streptococcus sp. NPS 308 Streptococcus sp.fdaargos_192 S18 Haemophilus influenzae Haemophilus influenzae
22 S19 NRF Streptococcus salivarius Streptococcus mitis Streptococcus sp.fdaargos_192 Streptococcus sanguinis Rothia mucilaginosa Veillonella parvula Streptococcus sp. oral taxon 431 Streptococcus gordonii Streptococcus constellatus S20 Haemophilus influenzae Haemophilus influenzae Rothia mucilaginosa Streptococcus mitis S21 NRF Haemophilus influenzae Streptococcus mitis Haemophilus parainfluenzae Streptococcus sp. oral taxon 431 Streptococcus salivarius Streptococcus pneumoniae S22 NRF Prevotella aninogenica Streptococcus mitis Streptococcus sanguinis Rothia mucilaginosa Veillonella parvula Haemophilus parainfluenzae Streptococcus salivarius Streptococcus sp. A12 Streptococcus cristatus S23 Haemophilus influenzae Haemophilus influenzae Streptococcus salivarius Veillonella parvula Streptococcus pseudopneumoniae Streptococcus mitis Streptococcus sp.fdaargos_192 S24 Haemophilus influenzae Haemophilus influenzae Haemophilus parainfluenzae Veillonella parvula Streptococcus salivarius Streptococcus sp. oral taxon 431 S25 Haemophilus influenzae Haemophilus influenzae S26 Moraxella catarrhalis Streptococcus mutans Moraxella catarrhalis Veillonella parvula
23 S27 Haemophilus influenzae & Staphylococcus aureus Streptococcus salivarius Streptococcus gordonii Rothia dentocariosa Lactobacillus salivarius Haemophilus influenzae Streptococcus pyogenes Staphylococcus aureus Streptococcus salivarius S28 NRF Streptococcus mitis Streptococcus salivarius Veillonella parvula Streptococcus sp. A12 Streptococcus sanguinis Rothia mucilaginosa Streptococcus sp. oral taxon 431 Streptococcus pneumoniae Streptococcus pseudopneumoniae Prevotella aninogenica Streptococcus sp. I-G2 Streptococcus sp. I-P16 Haemophilus parainfluenzae S29 Pseudomonas aeruginosa Pseudomonas aeruginosa Staphylococcus aureus S30 Pseudomonas aeruginosa Pseudomonas aeruginosa S31 NRF Haemophilus influenzae Streptococcus anginosus Prevotella intermedia Tannerella forsythia Bifidobacterium longum Rothia mucilaginosa Streptococcus gordonii Veillonella parvula Campylobacter concisus S32 NSG Escherichia coli S33 NRF Streptococcus salivarius Clavispora lusitaniae Streptococcus sp.fdaargos_192 Rothia mucilaginosa Saccharomyces cerevisiae S34 NSG Candida albicans Enterococcus faecalis S35 Escherichia coli Escherichia coli S36 Haemophilus influenzae Haemophilus influenzae Veillonella parvula
24 Streptococcus salivarius Streptococcus mitis S37 Pseudomonas aeruginosa Pseudomonas aeruginosa S38 Staphylococcus aureus & Pseudomonas aeruginosa Staphylococcus aureus Pseudomonas aeruginosa S39 Haemophilus influenzae Moraxella catarrhalis Haemophilus influenzae S40 MRSA Staphylococcus aureus Streptococcus constellatus Streptococcus anginosus Streptococcus intermedius S41 Haemophilus influenzae & Staphylococcus aureus Staphylococcus aureus Haemophilus influenzae Neisseria sicca Escherichia coli
25 Supplementary Table 9: qpcr gene targets and primer sequences. Organism Gene target Forward primer (5-3 ) Reverse primer (5-3 ) Probe (5-3 ) Reference Human Bacteria RNA polymerase A 16S rrna gene V3-V4 fragment TGAAGCCGTGCGGAA GG TCGTCGGCAGCGTCA GATGTGTATAAGAGAC AGCCTACGGGNGGCW GCAG E. coli cyaa CGATAATCGCCAGATG GC H. influenzae omp P6 AGCGGCTTGTAGTTCC TCTAACA K. pneumoniae mdh CGGGCGTAGCGCGTA A M. catarrhalis copb GGTGAGTGCCGCTTTT ACAAC P. aeruginosa oprl AGCCTTCCTGGTCCCC TTAC S. aureus eap ACTGTAACTTTGGCAC TGG S. maltophilia 23S rrna GCCGAAAGCCCAAGG TTT S. pneumoniae ply GCTTATGGGCGCCAA GTCTA ACAAGAGAGCCAAGTGTCG GTCTCGTGGGCTCGGAGATGTGTA TAAGAGACAGGACTACHVGGGTAT CTAATCC CCTAAGTTGCAGGAGATGG CAACAGAGTATCCGCCAAAAGTT GATACCCGCATTCACATTAAACAG TGTATCGCCTGCCAAGACAA CCTAATGAACCCCAGTGTATAAGT TTG GCAGATACCTCATTACCTGC CGACTTTCGTCCTCGCCTTA CAAAGCTTCAAAAGCAGCCTCTA [6FAM]TACCACGTCAT CTCCTTTGATGGCTCC TAT[BHQ1] Sybr Green Master Mix [6FAM]TAGAGCGCCTT CGGTGTCGGT[BHQ1] [6FAM]CGATGCTGCAG GCAATGGTGCT[BHQ1] [6FAM]CCCGGCATGGA TCGTTCCGA [BHQ1] [6FAM]TGCTTTTGCAG CTGTTAGCCAGCCTAA G[BHQ1] [6FAM]TGAACTGACGG TCGCCAACGGTT[BHQ 1] [6FAM]ATCGCAACGAC TGGCGCTA[BHQ1] Sybr Green Master Mix [6FAM]CTCAAGTTGGA AACCACGAGTAAGAGT GATGAA[BHQ1] Designed in house 56 Designed in house Designed in house 15 15
26 S. pyogenes sdab GGRACACGTACCCAA AATGTAGGA TCTTGAGCTCTTTGTTCGGTRTAG [6FAM]CGTGACCAAAA AGGCGGCATGC[BHQ1 ] 15
Mikrobiresistenssitilastot 2017
 Mikrobiresistenssitilastot 2017 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha Escherichia coli, veriviljelylöydökset, 2017 (vain potilaan
Mikrobiresistenssitilastot 2017 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha Escherichia coli, veriviljelylöydökset, 2017 (vain potilaan
Mikrobiresistenssitilastot Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha
 Mikrobiresistenssitilastot 2013 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha Escherichia coli, veriviljelylöydökset, 2013 (vain potilaan
Mikrobiresistenssitilastot 2013 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha Escherichia coli, veriviljelylöydökset, 2013 (vain potilaan
Mikrobiresistenssitilastot Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha
 Mikrobiresistenssitilastot 2014 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha Escherichia coli, veriviljelylöydökset, 2014 (vain potilaan
Mikrobiresistenssitilastot 2014 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha Escherichia coli, veriviljelylöydökset, 2014 (vain potilaan
Mikrobilääkeresistenssitilanne Suomessa ja maailmalla
 Mikrobilääkeresistenssitilanne Suomessa ja maailmalla Jari Jalava, FT 7.4.2017 1 Mikrobilääkeresistenssin seurantamekanismit FiRe - Finnish Study Group for Antimicrobial Resistance Tartuntatautirekisteri
Mikrobilääkeresistenssitilanne Suomessa ja maailmalla Jari Jalava, FT 7.4.2017 1 Mikrobilääkeresistenssin seurantamekanismit FiRe - Finnish Study Group for Antimicrobial Resistance Tartuntatautirekisteri
Mikrobien monilääkeresistenssi. Jari Jalava, FT
 Mikrobien monilääkeresistenssi Jari Jalava, FT Käsitteitä, lyhenteitä MRSA - metisilliiniresistentti Staphylococcus aureus, resistenssi kaikille -laktaamiantibiooteille mukaan lukien kefalosporiinit (meca/c-geenit)
Mikrobien monilääkeresistenssi Jari Jalava, FT Käsitteitä, lyhenteitä MRSA - metisilliiniresistentti Staphylococcus aureus, resistenssi kaikille -laktaamiantibiooteille mukaan lukien kefalosporiinit (meca/c-geenit)
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla / / Antibioottiresistenssitilanne Pohjoispohjanmaalla 2012 J Kauranen
 Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 212 1 Huom! Herkkyysmääritykset perustuvat vuodesta 211 alkaen pääosin EUCAST-standardin menetelmiin ja tulkintoihin, joten tulokset eivät ole kaikilta
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 212 1 Huom! Herkkyysmääritykset perustuvat vuodesta 211 alkaen pääosin EUCAST-standardin menetelmiin ja tulkintoihin, joten tulokset eivät ole kaikilta
Veriviljelypositiiviset infektiot TAYS:ssa Reetta Huttunen, ayl, infektiolääkäri, TAYS, Infektioyksikkö
 Veriviljelypositiiviset infektiot TAYS:ssa 1.10.2013 Reetta Huttunen, ayl, infektiolääkäri, TAYS, Infektioyksikkö Miksi? SIRO-tulosten analysointi sairaalainfektioiden seurantaa varten Tutkimusintressi
Veriviljelypositiiviset infektiot TAYS:ssa 1.10.2013 Reetta Huttunen, ayl, infektiolääkäri, TAYS, Infektioyksikkö Miksi? SIRO-tulosten analysointi sairaalainfektioiden seurantaa varten Tutkimusintressi
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla / / Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 2017 JKauranen
 Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 17 1 Huom! MRSA,VRE ja ESBL-löydöksissä mukana myös Keski-Pohjanmaan, Länsi-Pohjan ja Kainuun seulontalöydöksiä. Kliiniset näytteet tarkoittavat muita
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 17 1 Huom! MRSA,VRE ja ESBL-löydöksissä mukana myös Keski-Pohjanmaan, Länsi-Pohjan ja Kainuun seulontalöydöksiä. Kliiniset näytteet tarkoittavat muita
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla / / Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 2016 JKauranen
 Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 216 1 Huom! n määrä tarkoittaa niiden kantojen lukumäärää, joille on tehty herkkyysmääritys. Löydösmäärät voivat olla isompia. MRSA,VRE ja ESBL-löydöksissä
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 216 1 Huom! n määrä tarkoittaa niiden kantojen lukumäärää, joille on tehty herkkyysmääritys. Löydösmäärät voivat olla isompia. MRSA,VRE ja ESBL-löydöksissä
Supplementary information: Biocatalysis on the surface of Escherichia coli: melanin pigmentation of the cell. exterior
 Supplementary information: Biocatalysis on the surface of Escherichia coli: melanin pigmentation of the cell exterior Martin Gustavsson, David Hörnström, Susanna Lundh, Jaroslav Belotserkovsky, Gen Larsson
Supplementary information: Biocatalysis on the surface of Escherichia coli: melanin pigmentation of the cell exterior Martin Gustavsson, David Hörnström, Susanna Lundh, Jaroslav Belotserkovsky, Gen Larsson
Karbapenemaasin tuottajien epidemiologiaa. Jari Jalava, FT
 Karbapenemaasin tuottajien epidemiologiaa Jari Jalava, FT 26.3.2014 Terminologiaa CPE = Karbapenemaasia tuottava enterobakteeri ESBL = Laajakirjoista beetalaktamaasia tuottava enterobakteeri ST = sekvenssityyppi
Karbapenemaasin tuottajien epidemiologiaa Jari Jalava, FT 26.3.2014 Terminologiaa CPE = Karbapenemaasia tuottava enterobakteeri ESBL = Laajakirjoista beetalaktamaasia tuottava enterobakteeri ST = sekvenssityyppi
Experimental Identification and Computational Characterization of a Novel. Extracellular Metalloproteinase Produced by Clostridium sordellii
 Electronic Supplementary Material (ESI) for RSC Advances. This journal is The Royal Society of Chemistry 207 Supplementary Information Experimental Identification and Computational Characterization of
Electronic Supplementary Material (ESI) for RSC Advances. This journal is The Royal Society of Chemistry 207 Supplementary Information Experimental Identification and Computational Characterization of
Antibioottiresistenssitilanne Pohjois- Pohjanmaalla 2011
 1 Antibioottiresistenssitilanne Pohjois- Pohjanmaalla 211 2 Huom! Herkkyysmääritykset perustuvat vuodesta 211 alkaen pääosin EUCAST-standardin menetelmiin ja tulkintoihin, joten tulokset eivät ole kaikilta
1 Antibioottiresistenssitilanne Pohjois- Pohjanmaalla 211 2 Huom! Herkkyysmääritykset perustuvat vuodesta 211 alkaen pääosin EUCAST-standardin menetelmiin ja tulkintoihin, joten tulokset eivät ole kaikilta
HUSRES HERKKYYSTILASTOT 2018
 HUSRES HERKKYYSTILASTOT 2018 Suvi Korhonen, FM, erikoistuva sairaalamikrobiologi Johanna Haiko, FT, sairaalamikrobiologi Anu Pätäri-Sampo, LT, v.a. osastonylilääkäri Sisällysluettelo Staphylococcus aureus
HUSRES HERKKYYSTILASTOT 2018 Suvi Korhonen, FM, erikoistuva sairaalamikrobiologi Johanna Haiko, FT, sairaalamikrobiologi Anu Pätäri-Sampo, LT, v.a. osastonylilääkäri Sisällysluettelo Staphylococcus aureus
Kliinisesti merkittävien bakteerien jaottelua
 Johdanto kliinisesti merkittäviin bakteereihin Miksi kliininen bakteriologia on tärkeää? Bakteerien luokittelusta Bakteeri-infektiot Patogeeni Tartunnanlähde Ennaltaehkäisy Bakteriologista diagnostiikkaa
Johdanto kliinisesti merkittäviin bakteereihin Miksi kliininen bakteriologia on tärkeää? Bakteerien luokittelusta Bakteeri-infektiot Patogeeni Tartunnanlähde Ennaltaehkäisy Bakteriologista diagnostiikkaa
HUSRES HERKKYYSTILASTOT 2017
 HUSRES HERKKYYSTILASTOT 2017 JOHANNA HAIKO, FT, SAIRAALAMIKROBIOLOGI SUVI KORHONEN, FM, ERIKOISTUVA SAIRAALAMIKROBIOLOGI ANU PÄTÄRI-SAMPO, LT, V.A. OSASTONYLILÄÄKÄRI SISÄLLYSLUETTELO 1/2 HUSRES 2017 4
HUSRES HERKKYYSTILASTOT 2017 JOHANNA HAIKO, FT, SAIRAALAMIKROBIOLOGI SUVI KORHONEN, FM, ERIKOISTUVA SAIRAALAMIKROBIOLOGI ANU PÄTÄRI-SAMPO, LT, V.A. OSASTONYLILÄÄKÄRI SISÄLLYSLUETTELO 1/2 HUSRES 2017 4
KYSRES Herkkyysmääritysstandardi:
 KYSRES mikrobilääkeresistenssitilanne ssä vuonna kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi: Kuopion aluelaboratorio,
KYSRES mikrobilääkeresistenssitilanne ssä vuonna kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi: Kuopion aluelaboratorio,
Alueellinen sairaalahygieniapäivä Epidemiologinen katsaus
 Alueellinen sairaalahygieniapäivä 22.11.2017 -Epidemiologinen katsaus infektiolääkäri Hanna Viskari 1 2 7-vuotiaan tytön virtsan klebsiella. Anamneesissa ei ole erityistä syytä tällaiseen resistenssiin
Alueellinen sairaalahygieniapäivä 22.11.2017 -Epidemiologinen katsaus infektiolääkäri Hanna Viskari 1 2 7-vuotiaan tytön virtsan klebsiella. Anamneesissa ei ole erityistä syytä tällaiseen resistenssiin
Gram-värjäykset. Olli Meurman
 Gram-värjäykset Olli Meurman 5.2.2010 Gram-värjäys Gram-positiivinen Kiinnitys (kuumennus/alkoholi) Gram-negatiivinen Kristalliviolettivärjäys Kiinnitys jodilla Värinpoisto alkoholilla Safraniinivärjäys
Gram-värjäykset Olli Meurman 5.2.2010 Gram-värjäys Gram-positiivinen Kiinnitys (kuumennus/alkoholi) Gram-negatiivinen Kristalliviolettivärjäys Kiinnitys jodilla Värinpoisto alkoholilla Safraniinivärjäys
Ovatko MDR-mikrobit samanlaisia?
 Ovatko MDR-mikrobit samanlaisia? Risto Vuento 1 Onko sillä merkitystä, että MDR-mikrobit ovat samanlaisia tai erilaisia? Yleisesti kaikkeen hankittuun resistenssiin pitäisi suhtautua vakavasti Varotoimet
Ovatko MDR-mikrobit samanlaisia? Risto Vuento 1 Onko sillä merkitystä, että MDR-mikrobit ovat samanlaisia tai erilaisia? Yleisesti kaikkeen hankittuun resistenssiin pitäisi suhtautua vakavasti Varotoimet
Antibioottiresistenssitilanne Varsinais-Suomessa Kaisu Rantakokko-Jalava
 Antibioottiresistenssitilanne Varsinais-Suomessa 2015 Kaisu Rantakokko-Jalava 29.2.2016 Stafylokokkien resistenssi (% R) vuonna 2015 kliiniset näytteet (1 kanta/potilas) S. aureus S. epidermidis aikuiset
Antibioottiresistenssitilanne Varsinais-Suomessa 2015 Kaisu Rantakokko-Jalava 29.2.2016 Stafylokokkien resistenssi (% R) vuonna 2015 kliiniset näytteet (1 kanta/potilas) S. aureus S. epidermidis aikuiset
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla / / Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 2015 JKauranen
 Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 215 1 Huom! n määrä tarkoittaa niiden kantojen lukumäärää, joille on tehty herkkyysmääritys. Löydösmäärät voivat olla isompia. MRSA,VRE ja ESBL-löydöksissä
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 215 1 Huom! n määrä tarkoittaa niiden kantojen lukumäärää, joille on tehty herkkyysmääritys. Löydösmäärät voivat olla isompia. MRSA,VRE ja ESBL-löydöksissä
Mikrobilääkeresistenssi Pohjois-Savossa 2016
 Mikrobilääkeresistenssi Pohjois-Savossa www.islab.fi Terveydenhuollon-ammattilaiselle Mikrobiologian tilastoja mikrobilääkeresistenssitilanne ssä vuonna kliinisesti tärkeimmät bakteerilajit ja näytelaadut
Mikrobilääkeresistenssi Pohjois-Savossa www.islab.fi Terveydenhuollon-ammattilaiselle Mikrobiologian tilastoja mikrobilääkeresistenssitilanne ssä vuonna kliinisesti tärkeimmät bakteerilajit ja näytelaadut
Mikrobilääkeresistenssi Pohjois-Savon sairaanhoitopiirissä 2017
 Mikrobilääkeresistenssi ssä www.islab.fi Terveydenhuollon-ammattilaiselle Mikrobiologian tilastoja kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi:
Mikrobilääkeresistenssi ssä www.islab.fi Terveydenhuollon-ammattilaiselle Mikrobiologian tilastoja kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi:
Mikrobilääkeresistenssi Pohjois-Savon sairaanhoitopiirissä 2018
 Mikrobilääkeresistenssi ssä www.islab.fi Terveydenhuollon-ammattilaiselle Mikrobiologian tilastoja kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi:
Mikrobilääkeresistenssi ssä www.islab.fi Terveydenhuollon-ammattilaiselle Mikrobiologian tilastoja kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi:
Antibioottiresistenssitilanne Varsinais-Suomessa 2018
 Antibioottiresistenssitilanne Varsinais-Suomessa 2018 Kaisu Rantakokko-Jalava Tyks Kliininen mikrobiologia 21.3.2019 Stafylokokkien resistenssi (% R) vuonna 2018 kliiniset näytteet (1 kanta/potilas) S.
Antibioottiresistenssitilanne Varsinais-Suomessa 2018 Kaisu Rantakokko-Jalava Tyks Kliininen mikrobiologia 21.3.2019 Stafylokokkien resistenssi (% R) vuonna 2018 kliiniset näytteet (1 kanta/potilas) S.
tgg agg Supplementary Figure S1.
 ttaggatattcggtgaggtgatatgtctctgtttggaaatgtctccgccattaactcaag tggaaagtgtatagtaatgaatctttcaagcacacagatcacttcaaaagactgtttcaa catcacctcaggacaaaaagatgtactctcatttggatgctgtgatgccatgggtcacag attgcaattcccaagtgcccgttcttttacaccaaaatcaaagaagaatatctccccttt
ttaggatattcggtgaggtgatatgtctctgtttggaaatgtctccgccattaactcaag tggaaagtgtatagtaatgaatctttcaagcacacagatcacttcaaaagactgtttcaa catcacctcaggacaaaaagatgtactctcatttggatgctgtgatgccatgggtcacag attgcaattcccaagtgcccgttcttttacaccaaaatcaaagaagaatatctccccttt
Labquality-päivät OVATKO MONIRESISTENTIT BAKTEERIT KURISSA? Antti Hakanen Tartuntatautiseurannan ja -torjunnan osasto (TATO)
 Labquality-päivät 5.2.2009 OVATKO MONIRESISTENTIT BAKTEERIT KURISSA? Antti Hakanen Tartuntatautiseurannan ja -torjunnan osasto (TATO) Eivät ainakaan ulkomailla Moniresistenssin murheenkryynit Moniresistentti
Labquality-päivät 5.2.2009 OVATKO MONIRESISTENTIT BAKTEERIT KURISSA? Antti Hakanen Tartuntatautiseurannan ja -torjunnan osasto (TATO) Eivät ainakaan ulkomailla Moniresistenssin murheenkryynit Moniresistentti
MALE ADULT FIBROBLAST LINE (82-6hTERT)
 Double-stranded methylation patterns of a 104-bp L1 promoter in DNAs from male and female fibroblasts, male leukocytes and female lymphoblastoid cells using hairpin-bisulfite PCR. Fifteen L1 sequences
Double-stranded methylation patterns of a 104-bp L1 promoter in DNAs from male and female fibroblasts, male leukocytes and female lymphoblastoid cells using hairpin-bisulfite PCR. Fifteen L1 sequences
Bakteeriviljely 1- ja 2-kierrokset. Luettelo näytteissä olleista mikrobeista ja linkit kuvallisiin palautteisiin 2008 lähtien
 Ulkoinen laadunarviointikierros Bakteeriviljely 1- ja 2-kierrokset. Luettelo näytteissä olleista mikrobeista ja linkit kuvallisiin palautteisiin 2008 lähtien Mikrobi Kierros/ Näytteen laatu ja potilastietoja
Ulkoinen laadunarviointikierros Bakteeriviljely 1- ja 2-kierrokset. Luettelo näytteissä olleista mikrobeista ja linkit kuvallisiin palautteisiin 2008 lähtien Mikrobi Kierros/ Näytteen laatu ja potilastietoja
Bakteeriviljely 1- ja 2-kierrokset. Luettelo näytteissä olleista mikrobeista ja linkit kuvallisiin palautteisiin 2008 lähtien
 Ulkoinen laadunarviointikierros Bakteeriviljely 1- ja 2-kierrokset. Luettelo näytteissä olleista mikrobeista ja linkit kuvallisiin palautteisiin 2008 lähtien Mikrobi Kierros/ Näytteen laatu ja potilastietoja
Ulkoinen laadunarviointikierros Bakteeriviljely 1- ja 2-kierrokset. Luettelo näytteissä olleista mikrobeista ja linkit kuvallisiin palautteisiin 2008 lähtien Mikrobi Kierros/ Näytteen laatu ja potilastietoja
ESBL kantajuus Suomessa kliinisen tutkimuksen satoa
 ESBL kantajuus Suomessa kliinisen tutkimuksen satoa FT, yliopisto-opettaja Lääketieteellinen Mikrobiologia ja Immunologia Turun yliopisto Pitääkö olla huolissaan? Lähde: Review on Antimicrobial Resistance
ESBL kantajuus Suomessa kliinisen tutkimuksen satoa FT, yliopisto-opettaja Lääketieteellinen Mikrobiologia ja Immunologia Turun yliopisto Pitääkö olla huolissaan? Lähde: Review on Antimicrobial Resistance
Antibioottiresistenssitilanne Varsinais-Suomessa 2013
 Antibioottiresistenssitilanne Varsinais-Suomessa 2013 Olli Meurman Kliininen mikrobiologia Vuodesta 2011 alkaen tulkinta EUCAST-standardin mukaan, joten tulokset eivät ole kaikilta osin vertailukelpoisia
Antibioottiresistenssitilanne Varsinais-Suomessa 2013 Olli Meurman Kliininen mikrobiologia Vuodesta 2011 alkaen tulkinta EUCAST-standardin mukaan, joten tulokset eivät ole kaikilta osin vertailukelpoisia
Antibioottiresistenssitilanne Varsinais-Suomessa 2017
 Antibioottiresistenssitilanne Varsinais-Suomessa 2017 Kaisu Rantakokko-Jalava Tyks Kliininen mikrobiologia 2.3.2018 Stafylokokkien resistenssi (% R) vuonna 2017 kliiniset näytteet (1 kanta/potilas) S.
Antibioottiresistenssitilanne Varsinais-Suomessa 2017 Kaisu Rantakokko-Jalava Tyks Kliininen mikrobiologia 2.3.2018 Stafylokokkien resistenssi (% R) vuonna 2017 kliiniset näytteet (1 kanta/potilas) S.
FETAL FIBROBLASTS, PASSAGE 10
 Double-stranded methylation patterns of a 104-bp L1 promoter in DNAs from fetal fibroblast passages 10, 14, 17, and 22 using barcoded hairpinbisulfite PCR. Fifteen L1 sequences were analyzed for passages
Double-stranded methylation patterns of a 104-bp L1 promoter in DNAs from fetal fibroblast passages 10, 14, 17, and 22 using barcoded hairpinbisulfite PCR. Fifteen L1 sequences were analyzed for passages
KYSRES Herkkyysmääritysstandardi: Kuopion aluelaboratorio, Kliininen mikrobiologia
 KYSRES mikrobilääkeresistenssitilanne ssä vuonna kliinisesti merkittävimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi: Streptococcus pneumoniae
KYSRES mikrobilääkeresistenssitilanne ssä vuonna kliinisesti merkittävimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi: Streptococcus pneumoniae
Antibioottiresistenssi eilen, tänään ja huomenna
 Antibioottiresistenssi eilen, tänään ja huomenna Martti Vaara 27.8.212 Therapia Fennica -68 % Miten MRSA yleistyi USA:ssa 4 3 Hospital size 3 2 beds 197 1979 1983 1987 1991 199
Antibioottiresistenssi eilen, tänään ja huomenna Martti Vaara 27.8.212 Therapia Fennica -68 % Miten MRSA yleistyi USA:ssa 4 3 Hospital size 3 2 beds 197 1979 1983 1987 1991 199
Bakteerien mikrobilääkeresistenssi Suomessa
 Marianne Gunell Jari Jalava TYÖPAPERI Suomessa Finres 2012 1 2014 TYÖPAPERI 1/2014 Marianne Gunell ja Jari Jalava Suomessa Finres 2012 FiRe ja Terveyden ja hyvinvoinnin laitos Finres 2012 -työpaperin toimittamisesta
Marianne Gunell Jari Jalava TYÖPAPERI Suomessa Finres 2012 1 2014 TYÖPAPERI 1/2014 Marianne Gunell ja Jari Jalava Suomessa Finres 2012 FiRe ja Terveyden ja hyvinvoinnin laitos Finres 2012 -työpaperin toimittamisesta
Antibioottiresistenssitilanne Varsinais-Suomessa 2009
 Antibioottiresistenssitilanne Varsinais-Suomessa 2009 Olli Meurman Kliininen mikrobiologia Streptokokkien resistenssi (% R) vuonna 2009 koko aineisto (1 kanta/potilas) S. pyogenes S. agalactiae Str. -h
Antibioottiresistenssitilanne Varsinais-Suomessa 2009 Olli Meurman Kliininen mikrobiologia Streptokokkien resistenssi (% R) vuonna 2009 koko aineisto (1 kanta/potilas) S. pyogenes S. agalactiae Str. -h
KYSRES Herkkyysmääritysstandardi: Kuopion aluelaboratorio, Kliininen mikrobiologia
 KYSRES mikrobilääkeresistenssitilanne ssä vuonna kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi: Streptococcus pneumoniae
KYSRES mikrobilääkeresistenssitilanne ssä vuonna kliinisesti tärkeimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi: Streptococcus pneumoniae
Plasmid Name: pmm290. Aliases: none known. Length: bp. Constructed by: Mike Moser/Cristina Swanson. Last updated: 17 August 2009
 Plasmid Name: pmm290 Aliases: none known Length: 11707 bp Constructed by: Mike Moser/Cristina Swanson Last updated: 17 August 2009 Description and application: This is a mammalian expression vector for
Plasmid Name: pmm290 Aliases: none known Length: 11707 bp Constructed by: Mike Moser/Cristina Swanson Last updated: 17 August 2009 Description and application: This is a mammalian expression vector for
KOULUTUS ANNOSTELU JA ANNOSTELULAITTEIDEN KÄYTTÖEDUT PUHTAUS- JA HYGIENIA-ALAN ALUEELLINEN KOULUTUS KSSHP
 KOULUTUS ANNOSTELU JA ANNOSTELULAITTEIDEN KÄYTTÖEDUT PUHTAUS- JA HYGIENIA-ALAN ALUEELLINEN KOULUTUS KSSHP 30.11.2017 HELI LANKINEN 30.11.2017 1 TAUDINAIHEUTTAJAT TARTTUVAT HOITOYMPÄRISTÖN KOSKETUSPINNOILTA
KOULUTUS ANNOSTELU JA ANNOSTELULAITTEIDEN KÄYTTÖEDUT PUHTAUS- JA HYGIENIA-ALAN ALUEELLINEN KOULUTUS KSSHP 30.11.2017 HELI LANKINEN 30.11.2017 1 TAUDINAIHEUTTAJAT TARTTUVAT HOITOYMPÄRISTÖN KOSKETUSPINNOILTA
PALAUTE BAKTERIOLOGIAN LAADUNARVIOINTIKIERROKSILTA. Markku Koskela OYS/Mikrobiologian laboratorio (OML)
 PALAUTE BAKTERIOLOGIAN LAADUNARVIOINTIKIERROKSILTA Markku Koskela OYS/Mikrobiologian laboratorio (OML) Bakteriologian kierroksen koostumus 2003 lähtien Näytteet 1 ja 2: Aerobiviljely-> patogeenien tunnistus
PALAUTE BAKTERIOLOGIAN LAADUNARVIOINTIKIERROKSILTA Markku Koskela OYS/Mikrobiologian laboratorio (OML) Bakteriologian kierroksen koostumus 2003 lähtien Näytteet 1 ja 2: Aerobiviljely-> patogeenien tunnistus
AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY
 T126/M25/2015 Liite 1 / Appendix 1 Sivu / Page 1(5) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY KOKEMÄENJOEN VESISTÖN VESIENSUOJELUYHDISTYS RY, KVVY-RAUMALAB WATER PROTECTION ASSOCIATION
T126/M25/2015 Liite 1 / Appendix 1 Sivu / Page 1(5) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY KOKEMÄENJOEN VESISTÖN VESIENSUOJELUYHDISTYS RY, KVVY-RAUMALAB WATER PROTECTION ASSOCIATION
Mikrobilääkeresistenssitilastot
 Mikrobilääkeresistenssitilastot 12-17 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha R+I % 45 35 25 15 5 Escherichia coli 12-17, EPSHP Veriviljelynäytteet,
Mikrobilääkeresistenssitilastot 12-17 Etelä-Pohjanmaan sairaanhoitopiiri (EPSHP alue) Seinäjoen keskussairaala, Kliininen mikrobiologia Kerttu Saha R+I % 45 35 25 15 5 Escherichia coli 12-17, EPSHP Veriviljelynäytteet,
Uutta pikadiagnostiikkaan
 Uutta pikadiagnostiikkaan Joanna Peltola Sairaalamikrobiologi NordLab Rovaniemi Käsitteitä Perinteiset mikrobiologiset menetelmät Viljely Biokemiallinen tunnistus Kiekkoherkkyysmääritys Pikatestit Immunografiset
Uutta pikadiagnostiikkaan Joanna Peltola Sairaalamikrobiologi NordLab Rovaniemi Käsitteitä Perinteiset mikrobiologiset menetelmät Viljely Biokemiallinen tunnistus Kiekkoherkkyysmääritys Pikatestit Immunografiset
AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA
 T022/A19/2018 Liite 1 / Appendix 1 Sivu / Page 1(5) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA VALIO LTD, R&D, CHEMISTRY AND MICROBIOLOGY Tunnus
T022/A19/2018 Liite 1 / Appendix 1 Sivu / Page 1(5) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA VALIO LTD, R&D, CHEMISTRY AND MICROBIOLOGY Tunnus
KYSRES Kuopion aluelaboratorio, Kliininen mikrobiologia
 KYSRES 11 mikrobilääkkeiden resistenssitilanne Pohjois-Savon sairaanhoitopiirissä vuonna 11 kliinisesti merkittävimmät bakteerilajit ja näytelaadut UUTTA: vuoden 11 alussa Suomessa otettiin käyttöön EUCAST-herkkyysmääritysstandardi
KYSRES 11 mikrobilääkkeiden resistenssitilanne Pohjois-Savon sairaanhoitopiirissä vuonna 11 kliinisesti merkittävimmät bakteerilajit ja näytelaadut UUTTA: vuoden 11 alussa Suomessa otettiin käyttöön EUCAST-herkkyysmääritysstandardi
Taulukko 1. RespiFinder RG Panel -tuotteen toteamisraja puhdistus huomioiden (QIAamp MinElute Virus Spin -sarja) 10(0,40) TCID 50 /0,2 ml
 RespiFinder RG Panel Suorituksen ominaispiirteet RespiFinder RG Panel, versio 1, 4692163 Tarkista ennen kokeen suorittamista uusien elektronisten etikettiversioiden saatavuus osoitteesta www.qiagen.com/p/respifinder-rg-panel-ce.
RespiFinder RG Panel Suorituksen ominaispiirteet RespiFinder RG Panel, versio 1, 4692163 Tarkista ennen kokeen suorittamista uusien elektronisten etikettiversioiden saatavuus osoitteesta www.qiagen.com/p/respifinder-rg-panel-ce.
MIKROBILÄÄKERESISTENSSITILANNE 2014
 MIKROBILÄÄKERESISTENSSITILANNE 2014 Seuraavissa taulukoissa tutkittujen tapausten lukumäärä ja niistä lasketut prosenttiluvut on ilmoitettu potilaittain. Esitettyjä lukuja arvioitaessa on huomioitava,
MIKROBILÄÄKERESISTENSSITILANNE 2014 Seuraavissa taulukoissa tutkittujen tapausten lukumäärä ja niistä lasketut prosenttiluvut on ilmoitettu potilaittain. Esitettyjä lukuja arvioitaessa on huomioitava,
KYSRES Herkkyysmääritysstandardi: Kuopion aluelaboratorio, Kliininen mikrobiologia
 KYSRES mikrobilääkeresistenssitilanne Pohjois-Savon sairaanhoitopiirissä vuonna kliinisesti merkittävimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi:
KYSRES mikrobilääkeresistenssitilanne Pohjois-Savon sairaanhoitopiirissä vuonna kliinisesti merkittävimmät bakteerilajit ja näytelaadut yksi kyseisen lajin bakteerikantalöydös/potilas Herkkyysmääritysstandardi:
Labquality Ulkoinen laadunarviointikierros Bakteeriviljely 1 4/2014
 Labquality Ulkoinen laadunarviointikierros Bakteeriviljely 1 4/2014 Kuvat ja teksti: Markku Koskela Mikrobiologian ylilääkäri Nordlab Oulu Huom! Käyttäkää yläpalkin suurennusmahdollisuutta 100-400% pesäkekasvun
Labquality Ulkoinen laadunarviointikierros Bakteeriviljely 1 4/2014 Kuvat ja teksti: Markku Koskela Mikrobiologian ylilääkäri Nordlab Oulu Huom! Käyttäkää yläpalkin suurennusmahdollisuutta 100-400% pesäkekasvun
Supporting Information for
 Supporting Information for Analysis of Sogatella furcifera proteome that interact with P10 protein of Southern rice black-streaked dwarf virus Win Than*, Faliang Qin*, Wenwen Liu, Xifeng Wang ** State
Supporting Information for Analysis of Sogatella furcifera proteome that interact with P10 protein of Southern rice black-streaked dwarf virus Win Than*, Faliang Qin*, Wenwen Liu, Xifeng Wang ** State
Methods S1. Sequences relevant to the constructed strains, Related to Figures 1-6.
 Methods S1. Sequences relevant to the constructed strains, Related to Figures 1-6. A. Promoter Sequences Gal4 binding sites are highlighted in the color referenced in Figure 1A when possible. Site 1: red,
Methods S1. Sequences relevant to the constructed strains, Related to Figures 1-6. A. Promoter Sequences Gal4 binding sites are highlighted in the color referenced in Figure 1A when possible. Site 1: red,
Bakteerien mikrobilääkeherkkyys Suomessa
 TYÖPAPERI luonnos/2013 Marianne Gunell Suomessa Finres 2012 FiRe ja Terveyden ja hyvinvoinnin laitos Finres 2012 työpaperin toimittamisesta vastaa Marianne Gunell Työpaperi-sarjassa julkaistavan raportin
TYÖPAPERI luonnos/2013 Marianne Gunell Suomessa Finres 2012 FiRe ja Terveyden ja hyvinvoinnin laitos Finres 2012 työpaperin toimittamisesta vastaa Marianne Gunell Työpaperi-sarjassa julkaistavan raportin
THL:n laboratoriopohjainen seuranta ja kantakokoelmaan lähetettävät bakteerikannat,
 THL:n laboratoriopohjainen seuranta ja kantakokoelmaan lähetettävät bakteerikannat, 1.1.2015 Bakteeri Lähetysperuste Tyypitys THL:ssa ja vastauskäytäntö Minne lähetetään Metisilliiniresistentti Staphylococcus
THL:n laboratoriopohjainen seuranta ja kantakokoelmaan lähetettävät bakteerikannat, 1.1.2015 Bakteeri Lähetysperuste Tyypitys THL:ssa ja vastauskäytäntö Minne lähetetään Metisilliiniresistentti Staphylococcus
Miten tulkitsen mikrobiologisia laboratoriovastauksia?
 Miten tulkitsen mikrobiologisia laboratoriovastauksia? 08.11.2017 Sisätautimeeting Kerttu Saha, sairaalamikrobiologi Seinäjoen keskussairaala, Kliininen mikrobiologia Mikrobiologiset tutkimukset - Etsitään
Miten tulkitsen mikrobiologisia laboratoriovastauksia? 08.11.2017 Sisätautimeeting Kerttu Saha, sairaalamikrobiologi Seinäjoen keskussairaala, Kliininen mikrobiologia Mikrobiologiset tutkimukset - Etsitään
PRIMARY HPV TESTING IN ORGANIZED CERVICAL CANCER SCREENING
 PRIMARY HPV TESTING IN ORGANIZED CERVICAL CANCER SCREENING Veijalainen O, Kares S, Kujala P, Vuento R, TirKkonen M, Kholová I, Osuala V, Mäenpää J University and University Hospital of Tampere, Finland,
PRIMARY HPV TESTING IN ORGANIZED CERVICAL CANCER SCREENING Veijalainen O, Kares S, Kujala P, Vuento R, TirKkonen M, Kholová I, Osuala V, Mäenpää J University and University Hospital of Tampere, Finland,
AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY
 T042/A24/2015 Liite 1 / Appendix 1 Sivu / Page 1(7) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY KOKEMÄENJOEN VESISTÖN VESIENSUOJELUYHDISTYS RY, KVVY-PORILAB WATER PROTECTION ASSOCIATION
T042/A24/2015 Liite 1 / Appendix 1 Sivu / Page 1(7) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY KOKEMÄENJOEN VESISTÖN VESIENSUOJELUYHDISTYS RY, KVVY-PORILAB WATER PROTECTION ASSOCIATION
VIIKKI BIOCENTER University of Helsinki
 VIIKKI BIOCENTER University of Helsinki Biologian DNA koodi ja sen selvittäminen Petri Auvinen DNA Sequencing and Genomics Laboratory Institute of Biotechnology Kuinka solut kehittyivät? Kolmenlaisia soluja
VIIKKI BIOCENTER University of Helsinki Biologian DNA koodi ja sen selvittäminen Petri Auvinen DNA Sequencing and Genomics Laboratory Institute of Biotechnology Kuinka solut kehittyivät? Kolmenlaisia soluja
VALMISTEYHTEENVETO. Yksi injektiopullo sisältää meropeneemitrihydraattia vastaten 500 mg meropeneemia.
 VALMISTEYHTEENVETO 1. LÄÄKEVALMISTEEN NIMI Meropenem Fresenius Kabi 500 mg injektio-/infuusiokuiva-aine, liuosta varten Meropenem Fresenius Kabi 1 g injektio-/infuusiokuiva-aine, liuosta varten 2. VAIKUTTAVAT
VALMISTEYHTEENVETO 1. LÄÄKEVALMISTEEN NIMI Meropenem Fresenius Kabi 500 mg injektio-/infuusiokuiva-aine, liuosta varten Meropenem Fresenius Kabi 1 g injektio-/infuusiokuiva-aine, liuosta varten 2. VAIKUTTAVAT
SEMINAARI SFS:SSÄ UUDET YHTEISET STANDARDIT YMPÄRISTÖANALYTIIKKAAN? 13.5.2014 PENTTI MANNINEN
 SEMINAARI SFS:SSÄ UUDET YHTEISET STANDARDIT YMPÄRISTÖANALYTIIKKAAN? 13.5.2014 PENTTI MANNINEN PROJECT HORIZONTAL (1) Euroopan komission rahoittama hanke, jonka puitteissa oli tarkoitus luoda yhtenäisiä
SEMINAARI SFS:SSÄ UUDET YHTEISET STANDARDIT YMPÄRISTÖANALYTIIKKAAN? 13.5.2014 PENTTI MANNINEN PROJECT HORIZONTAL (1) Euroopan komission rahoittama hanke, jonka puitteissa oli tarkoitus luoda yhtenäisiä
AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA
 T022/M18/2017 Liite 1 / Appendix 1 Sivu / Page 1(7) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA VALIO LTD, R&D, CHEMISTRY AND MICROBIOLOGY Tunnus
T022/M18/2017 Liite 1 / Appendix 1 Sivu / Page 1(7) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA VALIO LTD, R&D, CHEMISTRY AND MICROBIOLOGY Tunnus
VALMISTEYHTEENVETO. 1. LÄÄKEVALMISTEEN NIMI Doxitin 100 mg tabletti
 VALMISTEYHTEENVETO 1. LÄÄKEVALMISTEEN NIMI Doxitin 100 mg tabletti 2. VAIKUTTAVAT AINEET JA NIIDEN MÄÄRÄT Yksi tabletti sisältää doksisykliiniä 100 mg. Apuaineet: Täydellinen apuaineluettelo, ks. kohta
VALMISTEYHTEENVETO 1. LÄÄKEVALMISTEEN NIMI Doxitin 100 mg tabletti 2. VAIKUTTAVAT AINEET JA NIIDEN MÄÄRÄT Yksi tabletti sisältää doksisykliiniä 100 mg. Apuaineet: Täydellinen apuaineluettelo, ks. kohta
VALMISTEYHTEENVETO. Injektiopullo sisältää keftriaksonidinatriumhemiheptahydraattia vastaten 1 g tai 2 g keftriaksonia.
 VALMISTEYHTEENVETO 1. LÄÄKEVALMISTEEN NIMI Ceftriaxon Villerton 1 g injektiokuiva-aine, liuosta varten Ceftriaxon Villerton 2 g infuusiokuiva-aine, liuosta varten 2. VAIKUTTAVAT AINEET JA NIIDEN MÄÄRÄT
VALMISTEYHTEENVETO 1. LÄÄKEVALMISTEEN NIMI Ceftriaxon Villerton 1 g injektiokuiva-aine, liuosta varten Ceftriaxon Villerton 2 g infuusiokuiva-aine, liuosta varten 2. VAIKUTTAVAT AINEET JA NIIDEN MÄÄRÄT
Labquality Kudos- ja märkänäytteiden bakteeriviljely 2006-2010
 Labquality Kudos- ja märkänäytteiden bakteeriviljely 2006-2010 Markku Koskela OYS/Mikrobiologian laboratorio 5-vuotisseurantajakson kertymä 20 bakteeriviljelykierrosta 80 potilasnäytettä 40 Pu-BaktVi2;
Labquality Kudos- ja märkänäytteiden bakteeriviljely 2006-2010 Markku Koskela OYS/Mikrobiologian laboratorio 5-vuotisseurantajakson kertymä 20 bakteeriviljelykierrosta 80 potilasnäytettä 40 Pu-BaktVi2;
Functional Genomics & Proteomics
 Functional Genomics & Proteomics Genome Sequences TCACAATTTAGACATCTAGTCTTCCACTTAAGCATATTTAGATTGTTTCCAGTTTTCAGCTTTTATGACTAAATCTTCTAAAATTGTTTTTCCCTAAATGTATATTTTAATTTGTCTCAGGAGTAGAATTTCTGAGTCATAAAGCGGT CATATGTATAAATTTTAGGTGCCTCATAGCTCTTCAAATAGTCATCCCATTTTATACATCCAGGCAATATATGAGAGTTCTTGGTGCTCCACATCTTAGCTAGGATTTGATGTCAACCAGTCTCTTTAATTTAGATATTCTAGTACAT
Functional Genomics & Proteomics Genome Sequences TCACAATTTAGACATCTAGTCTTCCACTTAAGCATATTTAGATTGTTTCCAGTTTTCAGCTTTTATGACTAAATCTTCTAAAATTGTTTTTCCCTAAATGTATATTTTAATTTGTCTCAGGAGTAGAATTTCTGAGTCATAAAGCGGT CATATGTATAAATTTTAGGTGCCTCATAGCTCTTCAAATAGTCATCCCATTTTATACATCCAGGCAATATATGAGAGTTCTTGGTGCTCCACATCTTAGCTAGGATTTGATGTCAACCAGTCTCTTTAATTTAGATATTCTAGTACAT
Supplementary Material : Baker et al. 1
 Supplementary Material : Baker et al. 1 Supplementary Material Title: Species identity and human consumption of beaked whales in the Gilbert Islands, Republic of Kiribati Authors: C. Scott Baker, Al Hutt,
Supplementary Material : Baker et al. 1 Supplementary Material Title: Species identity and human consumption of beaked whales in the Gilbert Islands, Republic of Kiribati Authors: C. Scott Baker, Al Hutt,
Asiakaspalautteen merkitys laboratoriovirheiden paljastamisessa. Taustaa
 Asiakaspalautteen merkitys laboratoriovirheiden paljastamisessa Paula Oja, TtT Laboratorio, Oulun yliopistollinen sairaala Potilasturvallisuustutkimuksen päivät 26. 27.1.2011 1 Taustaa Laboratorion tulee
Asiakaspalautteen merkitys laboratoriovirheiden paljastamisessa Paula Oja, TtT Laboratorio, Oulun yliopistollinen sairaala Potilasturvallisuustutkimuksen päivät 26. 27.1.2011 1 Taustaa Laboratorion tulee
Efficiency change over time
 Efficiency change over time Heikki Tikanmäki Optimointiopin seminaari 14.11.2007 Contents Introduction (11.1) Window analysis (11.2) Example, application, analysis Malmquist index (11.3) Dealing with panel
Efficiency change over time Heikki Tikanmäki Optimointiopin seminaari 14.11.2007 Contents Introduction (11.1) Window analysis (11.2) Example, application, analysis Malmquist index (11.3) Dealing with panel
Supporting information
 Supporting information Figure S1. Carotenoid biosynthesis pathway in papaya fruit which is adopted from Blas et al. (2010) (reference 4) and Nisar et al. (2015) (reference 5). Carotenoids are synthesized
Supporting information Figure S1. Carotenoid biosynthesis pathway in papaya fruit which is adopted from Blas et al. (2010) (reference 4) and Nisar et al. (2015) (reference 5). Carotenoids are synthesized
Moderni diagnostiikka, nopeampi vastaus Kokemuksia Puijon laboratoriosta. Sairaalamikrobiologi Jaana Pentikäinen
 Moderni diagnostiikka, nopeampi vastaus Kokemuksia Puijon laboratoriosta Sairaalamikrobiologi Jaana Pentikäinen ISLAB Liikelaitoskuntayhtymä Neljä aluelaboratoriota, yhteensä 7 sairaalaa Kuopio Iisalmi
Moderni diagnostiikka, nopeampi vastaus Kokemuksia Puijon laboratoriosta Sairaalamikrobiologi Jaana Pentikäinen ISLAB Liikelaitoskuntayhtymä Neljä aluelaboratoriota, yhteensä 7 sairaalaa Kuopio Iisalmi
Pihkauutteen mikrobiologiaa
 Pihkauutteen mikrobiologiaa 1. Taustaa Lapin ammattiopiston toimeksiannosta tutkittiin pihka / kasvisöljyseoksen antimikrobista tehoa. 2. Tutkimusmenetelmä Antimikrobinen teho arvioitiin sovelletulla agardiffuusiomenetelmällä
Pihkauutteen mikrobiologiaa 1. Taustaa Lapin ammattiopiston toimeksiannosta tutkittiin pihka / kasvisöljyseoksen antimikrobista tehoa. 2. Tutkimusmenetelmä Antimikrobinen teho arvioitiin sovelletulla agardiffuusiomenetelmällä
Menestyminen ulkoisessa laadunarvioinnissa Miten meillä ja muualla? Eveliina Tarkka HUSLAB Bakteriologia Labqualitypäivät
 Menestyminen ulkoisessa laadunarvioinnissa Miten meillä ja muualla? Eveliina Tarkka HUSLAB Bakteriologia Labqualitypäivät 5.2.2009 Suomi muu maailma Vertailu UK Neqasin laadunarviointitulosten perusteella
Menestyminen ulkoisessa laadunarvioinnissa Miten meillä ja muualla? Eveliina Tarkka HUSLAB Bakteriologia Labqualitypäivät 5.2.2009 Suomi muu maailma Vertailu UK Neqasin laadunarviointitulosten perusteella
Supplementary Information
 Supplementary Information Supplementary Table 1. Strain table Strain name Genotype Reference CEN.PK111-27B MATa leu2 trp1 Euroscarf TC-49 TC-50 pdc5::aro4* aro10::aro7* This study TC-50 MATa leu2 trp1
Supplementary Information Supplementary Table 1. Strain table Strain name Genotype Reference CEN.PK111-27B MATa leu2 trp1 Euroscarf TC-49 TC-50 pdc5::aro4* aro10::aro7* This study TC-50 MATa leu2 trp1
Mikrobiologisia näkökohtia luomumaidosta ja luomujuustosta
 Tapani Alatossava Elintarviketeknologian laitos Helsingin yliopisto Luomuelintarvikepäivät 13.-14.2.2006 Helsinki Taustaa: Mikrobiologisia näkökohtia Ns. luomujuustohanke eli Luomujuustojen tuotantoketjun
Tapani Alatossava Elintarviketeknologian laitos Helsingin yliopisto Luomuelintarvikepäivät 13.-14.2.2006 Helsinki Taustaa: Mikrobiologisia näkökohtia Ns. luomujuustohanke eli Luomujuustojen tuotantoketjun
Pihkauutteen mikrobiologiaa. Perusselvitys pihkajalosteen antimikrobisista ominaisuuksista
 Pihkauutteen mikrobiologiaa Perusselvitys pihkajalosteen antimikrobisista ominaisuuksista Rainer Peltola Täsmätietoa Lapin luonnontuotteista maakunnalle 2016 Pihkauutteen mikrobiologiaa Perusselvitys
Pihkauutteen mikrobiologiaa Perusselvitys pihkajalosteen antimikrobisista ominaisuuksista Rainer Peltola Täsmätietoa Lapin luonnontuotteista maakunnalle 2016 Pihkauutteen mikrobiologiaa Perusselvitys
Tuberkuloosin diagnostiikka
 Tuberkuloosin diagnostiikka 30. Valtakunnalliset Tartuntatautipäivät, Helsinki 13.11.2017 Jussi Eskola, dosentti (ei sidonnaisuuksia) Tuberkuloosidiagnostiikan kehittyminen 1910-luku: tuberkuliinikoe 1920-
Tuberkuloosin diagnostiikka 30. Valtakunnalliset Tartuntatautipäivät, Helsinki 13.11.2017 Jussi Eskola, dosentti (ei sidonnaisuuksia) Tuberkuloosidiagnostiikan kehittyminen 1910-luku: tuberkuliinikoe 1920-
AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA
 T022/A16/2014 Liite 1 / Appendix 1 Sivu / Page 1(7) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA VALIO LTD, R&D, CHEMISTRY AND MICROBIOLOGY Tunnus
T022/A16/2014 Liite 1 / Appendix 1 Sivu / Page 1(7) AKKREDITOITU TESTAUSLABORATORIO ACCREDITED TESTING LABORATORY VALIO OY, T&K, KEMIA JA MIKROBIOLOGIA VALIO LTD, R&D, CHEMISTRY AND MICROBIOLOGY Tunnus
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 2013. / 23.4.2014 / Antibioottiresistenssitilanne Pohjoispohjanmaalla 2013 J Kauranen
 Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 213 1 Huom! Herkkyysmääritykset perustuvat vuodesta 211 alkaen pääosin EUCAST-standardin menetelmiin ja tulkintoihin, joten tulokset eivät ole kaikilta
Mikrobilääkeresistenssitilanne Pohjois-Pohjanmaalla 213 1 Huom! Herkkyysmääritykset perustuvat vuodesta 211 alkaen pääosin EUCAST-standardin menetelmiin ja tulkintoihin, joten tulokset eivät ole kaikilta
Table S2. S. thermophilus CRISPR spacer sequence analysis.
 Table S2. S. thermophilus CRISPR analysis. 001_1_01 TGTTTGACAGCAAATCAAGATTCGAATTGT 30 AF115103 Streptococcus thermophilus bacteriophage Sfi21, compl 40 93 28 2 0 3 30 38304 38331 1 001_1_02 AATGACGAGGAGCTATTGGCACAACTTACA
Table S2. S. thermophilus CRISPR analysis. 001_1_01 TGTTTGACAGCAAATCAAGATTCGAATTGT 30 AF115103 Streptococcus thermophilus bacteriophage Sfi21, compl 40 93 28 2 0 3 30 38304 38331 1 001_1_02 AATGACGAGGAGCTATTGGCACAACTTACA
Utaretulehdusten PCR diagnostiikka - miten tuloksia tulkitaan?
 Utaretulehdusten PCR diagnostiikka - miten tuloksia tulkitaan? Heidi Hiitiö 1*, Rauna Riva 2, Tiina Autio 2, Tarja Pohjanvirta 2, Jani Holopainen 3, Satu Pyörälä 1, Sinikka Pelkonen 2 1 Department of production
Utaretulehdusten PCR diagnostiikka - miten tuloksia tulkitaan? Heidi Hiitiö 1*, Rauna Riva 2, Tiina Autio 2, Tarja Pohjanvirta 2, Jani Holopainen 3, Satu Pyörälä 1, Sinikka Pelkonen 2 1 Department of production
VERISUONIKATETRI-INFEKTIOT
 VERISUONIKATETRI-INFEKTIOT Kirsi Terho Hygieniahoitaja, TtM VSSHP/TYKS 25.3.2014 VERISUONIKATETRIEN INFEKTIOIDEN ESIINTYVYYS Verisuonikatetri-infektiot l. verisuonikatetrihoitoon liittyvät infektiot Riski
VERISUONIKATETRI-INFEKTIOT Kirsi Terho Hygieniahoitaja, TtM VSSHP/TYKS 25.3.2014 VERISUONIKATETRIEN INFEKTIOIDEN ESIINTYVYYS Verisuonikatetri-infektiot l. verisuonikatetrihoitoon liittyvät infektiot Riski
Capacity Utilization
 Capacity Utilization Tim Schöneberg 28th November Agenda Introduction Fixed and variable input ressources Technical capacity utilization Price based capacity utilization measure Long run and short run
Capacity Utilization Tim Schöneberg 28th November Agenda Introduction Fixed and variable input ressources Technical capacity utilization Price based capacity utilization measure Long run and short run
FiRe STANDARDI versio 6 Liite 5 Bakteeriryhmäkohtaiset kommentit Sivu 1(9)
 Bakteeriryhmäkohtaiset kommentit Sivu 1(9) Bakteeriryhmäkohtaiset kommentit Sisällys 1. Enterobakteerit...1 1.0. Beetalaktamaasivälitteinen resistenssi...1 1.1. E. coli...3 1.2. Klebsiella...3 1.3. Proteus
Bakteeriryhmäkohtaiset kommentit Sivu 1(9) Bakteeriryhmäkohtaiset kommentit Sisällys 1. Enterobakteerit...1 1.0. Beetalaktamaasivälitteinen resistenssi...1 1.1. E. coli...3 1.2. Klebsiella...3 1.3. Proteus
Metsälamminkankaan tuulivoimapuiston osayleiskaava
 VAALAN KUNTA TUULISAIMAA OY Metsälamminkankaan tuulivoimapuiston osayleiskaava Liite 3. Varjostusmallinnus FCG SUUNNITTELU JA TEKNIIKKA OY 12.5.2015 P25370 SHADOW - Main Result Assumptions for shadow calculations
VAALAN KUNTA TUULISAIMAA OY Metsälamminkankaan tuulivoimapuiston osayleiskaava Liite 3. Varjostusmallinnus FCG SUUNNITTELU JA TEKNIIKKA OY 12.5.2015 P25370 SHADOW - Main Result Assumptions for shadow calculations
Moniresistentit mikrobit MRSA, ESBL, CPE ja VRE. Alueellinen koulutus Mikrobiologi Terhi Tuhkalainen
 Moniresistentit mikrobit MRSA, ESBL, CPE ja VRE Alueellinen koulutus 5.10.2018 Mikrobiologi Terhi Tuhkalainen Esityksen sisältö Mikrobien antibioottiresistenssi Moniresistentit bakteerit (MDR) MRSA = Methicillin
Moniresistentit mikrobit MRSA, ESBL, CPE ja VRE Alueellinen koulutus 5.10.2018 Mikrobiologi Terhi Tuhkalainen Esityksen sisältö Mikrobien antibioottiresistenssi Moniresistentit bakteerit (MDR) MRSA = Methicillin
Tartuntojen torjunta ja ehkäisy sekä työturvallisuus Välineiden huolto Hygieniahoitaja Nina Pulli 18.5.2016
 Välineiden huolto Hygieniahoitaja Nina Pulli 18.5.2016 Kotkan kaupungin hygieniahoitaja Nina Pulli Karhulan sairaala (Terveysaseman alakerta) Toivelinnankatu 2 48600 Kotka Puh. +358 400 924 788 nina.pulli@kotka.fi
Välineiden huolto Hygieniahoitaja Nina Pulli 18.5.2016 Kotkan kaupungin hygieniahoitaja Nina Pulli Karhulan sairaala (Terveysaseman alakerta) Toivelinnankatu 2 48600 Kotka Puh. +358 400 924 788 nina.pulli@kotka.fi
ReFuel 70 % Emission Reduction Using Renewable High Cetane Number Paraffinic Diesel Fuel. Kalle Lehto, Aalto-yliopisto 5.5.
 ReFuel 70 % Emission Reduction Using Renewable High Cetane Number Paraffinic Diesel Fuel Kalle Lehto, Aalto-yliopisto 5.5.2011 Otaniemi ReFuel a three year research project (2009-2011) goal utilize the
ReFuel 70 % Emission Reduction Using Renewable High Cetane Number Paraffinic Diesel Fuel Kalle Lehto, Aalto-yliopisto 5.5.2011 Otaniemi ReFuel a three year research project (2009-2011) goal utilize the
Capacity utilization
 Mat-2.4142 Seminar on optimization Capacity utilization 12.12.2007 Contents Summary of chapter 14 Related DEA-solver models Illustrative examples Measure of technical capacity utilization Price-based measure
Mat-2.4142 Seminar on optimization Capacity utilization 12.12.2007 Contents Summary of chapter 14 Related DEA-solver models Illustrative examples Measure of technical capacity utilization Price-based measure
Stormwater filtration unit
 Stormwater filtration unit Background, concept and applied design work Olli Hakala 2018 WSP Finland Aalto university Kyttä ym. 2014. Veden äärellä kysely, ENTJUSTESS-hanke. Aalto yliopisto. STORMWATER
Stormwater filtration unit Background, concept and applied design work Olli Hakala 2018 WSP Finland Aalto university Kyttä ym. 2014. Veden äärellä kysely, ENTJUSTESS-hanke. Aalto yliopisto. STORMWATER
Viral DNA as a model for coil to globule transition
 Viral DNA as a model for coil to globule transition Marina Rossi Lab. of complex fluids and molecular biophysics LITA (Segrate) UNIVERSITA DEGLI STUDI DI MILANO - PhD Workshop October 14 th, 2013 Temperature
Viral DNA as a model for coil to globule transition Marina Rossi Lab. of complex fluids and molecular biophysics LITA (Segrate) UNIVERSITA DEGLI STUDI DI MILANO - PhD Workshop October 14 th, 2013 Temperature
Moniresistentit mikrobit MRSA, ESBL, CPE ja VRE. Alueellinen koulutus Mikrobiologi Terhi Tuhkalainen
 Moniresistentit mikrobit MRSA, ESBL, CPE ja VRE Alueellinen koulutus 5.10.2018 Mikrobiologi Terhi Tuhkalainen Esityksen sisältö Mikrobien antibioottiresistenssi Moniresistentit bakteerit (MDR) MRSA = Methicillin
Moniresistentit mikrobit MRSA, ESBL, CPE ja VRE Alueellinen koulutus 5.10.2018 Mikrobiologi Terhi Tuhkalainen Esityksen sisältö Mikrobien antibioottiresistenssi Moniresistentit bakteerit (MDR) MRSA = Methicillin
Vaikeat hengitystieinfektiot mikrobiologinen diagnostiikka
 Vaikeat hengitystieinfektiot mikrobiologinen diagnostiikka Tehohoidon vaikeat hengitystieinfektiot symposium 4.5.212, Helsinki Dos. Maija Lappalainen HUSLAB Infektioiden yleisyys teho-osastoilla 45-6%
Vaikeat hengitystieinfektiot mikrobiologinen diagnostiikka Tehohoidon vaikeat hengitystieinfektiot symposium 4.5.212, Helsinki Dos. Maija Lappalainen HUSLAB Infektioiden yleisyys teho-osastoilla 45-6%
( ( OX2 Perkkiö. Rakennuskanta. Varjostus. 9 x N131 x HH145
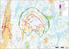 OX2 9 x N131 x HH145 Rakennuskanta Asuinrakennus Lomarakennus Liike- tai julkinen rakennus Teollinen rakennus Kirkko tai kirkollinen rak. Muu rakennus Allas Varjostus 1 h/a 8 h/a 20 h/a 0 0,5 1 1,5 2 km
OX2 9 x N131 x HH145 Rakennuskanta Asuinrakennus Lomarakennus Liike- tai julkinen rakennus Teollinen rakennus Kirkko tai kirkollinen rak. Muu rakennus Allas Varjostus 1 h/a 8 h/a 20 h/a 0 0,5 1 1,5 2 km
Tynnyrivaara, OX2 Tuulivoimahanke. ( Layout 9 x N131 x HH145. Rakennukset Asuinrakennus Lomarakennus 9 x N131 x HH145 Varjostus 1 h/a 8 h/a 20 h/a
 , Tuulivoimahanke Layout 9 x N131 x HH145 Rakennukset Asuinrakennus Lomarakennus 9 x N131 x HH145 Varjostus 1 h/a 8 h/a 20 h/a 0 0,5 1 1,5 km 2 SHADOW - Main Result Assumptions for shadow calculations
, Tuulivoimahanke Layout 9 x N131 x HH145 Rakennukset Asuinrakennus Lomarakennus 9 x N131 x HH145 Varjostus 1 h/a 8 h/a 20 h/a 0 0,5 1 1,5 km 2 SHADOW - Main Result Assumptions for shadow calculations
WindPRO version joulu 2012 Printed/Page :42 / 1. SHADOW - Main Result
 SHADOW - Main Result Assumptions for shadow calculations Maximum distance for influence Calculate only when more than 20 % of sun is covered by the blade Please look in WTG table 13.6.2013 19:42 / 1 Minimum
SHADOW - Main Result Assumptions for shadow calculations Maximum distance for influence Calculate only when more than 20 % of sun is covered by the blade Please look in WTG table 13.6.2013 19:42 / 1 Minimum
RANTALA SARI: Sairaanhoitajan eettisten ohjeiden tunnettavuus ja niiden käyttö hoitotyön tukena sisätautien vuodeosastolla
 TURUN YLIOPISTO Hoitotieteen laitos RANTALA SARI: Sairaanhoitajan eettisten ohjeiden tunnettavuus ja niiden käyttö hoitotyön tukena sisätautien vuodeosastolla Pro gradu -tutkielma, 34 sivua, 10 liitesivua
TURUN YLIOPISTO Hoitotieteen laitos RANTALA SARI: Sairaanhoitajan eettisten ohjeiden tunnettavuus ja niiden käyttö hoitotyön tukena sisätautien vuodeosastolla Pro gradu -tutkielma, 34 sivua, 10 liitesivua
Olli Meurman Labquality-päivät
 Olli Meurman Labquality-päivät 8.2.2007 Värimaljoja virtsaviljelyyn Chromagar Orientation (BD) CPS ID3 (biomérieux) Uriselect 4 (Bio-Rad Laboratories) Chromogenic UTI Medium (Oxoid) Harlequin CLED (Lab
Olli Meurman Labquality-päivät 8.2.2007 Värimaljoja virtsaviljelyyn Chromagar Orientation (BD) CPS ID3 (biomérieux) Uriselect 4 (Bio-Rad Laboratories) Chromogenic UTI Medium (Oxoid) Harlequin CLED (Lab
Korkeakoulujen tietohallinto ja tutkimus: kumpi ohjaa kumpaa?
 Korkeakoulujen tietohallinto ja tutkimus: kumpi ohjaa kumpaa? Kerro meille datastasi työpaja 10.4.2013 Antti Auer Tietohallintopäällikkö Jyväskylän yliopisto Strateginen kehittäminen Johtamista, tutkimushallintoa
Korkeakoulujen tietohallinto ja tutkimus: kumpi ohjaa kumpaa? Kerro meille datastasi työpaja 10.4.2013 Antti Auer Tietohallintopäällikkö Jyväskylän yliopisto Strateginen kehittäminen Johtamista, tutkimushallintoa
